【6.1.1】PDB相关数据库
我们提出了一系列数据库( http://swift.cmbi.ru.nl/gv/facilities/ ),其中包含从蛋白质数据库(PDB)条目中计算得出的信息,并且可能会加强大分子结构研究。这些派生数据库与PDB并行运行,即,每个PDB条目都有一个条目。一些完善的数据库,例如HSSP,PDBREPORT和PDB REDO已被更新和/或改进。例如,创建DSSP数据库的软件已被重写,以更好地处理 π-helices。已经添加了大量数据库以帮助计算结构生物学。一些示例包括与晶体进行接触的残基列表,使用一系列接触定义的接触残基列表或残基可及性列表。对于许多研究而言,PDB文件并不是基础数据的最佳表示形式。因此,我们制作了一系列数据库,这些数据库以更易于使用或更一致的表示形式保存PDB文件。 BDB数据库保存X射线PDB文件,这些文件始终代表B因子。我们还添加了一些可视化工具来帮助我们的数据库用户
一、数据库介绍

DSSP是在PDB条目中分配二级结构元素的事实上的标准。 为了更好地识别π螺旋(8-10),对DSSP(8)软件进行了重写。 π螺旋的确定仍然遵循Kabsch和Sander(8)的最初描述,但是现在π螺旋的分配优先于α螺旋的分配,这应避免低估π螺旋的数量(9 ,10)。
现在使用原始Sander和Schneider(11)算法的改进版本创建了HSSP(11-15)多序列比对(MSA)。 这些文件以原始HSSP格式和Pfam Stockholm格式(16)可用。 HMMER(17)和Jalview(18)等应用程序使用Stockholm格式。 BioJava(19),BioPerl(20)和Biopython(21)之类的项目为这些Stockholm格式的文件提供了解析器。
现在使用稍微修改的算法创建PDBFINDER和PDBFINDER2数据库(22),以更好地处理PDB文件中的异常。 还考虑了对DSSP和HSSP软件的更改。 此外,现在针对每个PDB ID从单独的文件编译了两个单一的纯文本文件。
PDB_REDO决策算法的新进展已在其他地方介绍(23)。 PDB_REDO中的许多最新改进都集中在启用用户友好的数据挖掘和可视化上。 所有重要结构变化的列表,例如改变的旋转异构体和翻转的肽平面(24),都以易于挖掘的格式提供,以快速确定PDB_REDO是否已更改特定PDB条目中的目标残基。 模型验证数据,例如WHAT_CHECK Z得分,晶体学R因子,适合晶体学数据的每个残基量度(实际空间R因子(25)和实际空间相关系数(26)),以及 现在还提供了对配体质量和结构相互作用的全面描述(27)。 补充表S1中给出了来自PDB_REDO条目的所有数据的描述。
PDB_SELECT(28)现在还提供了按质量排序的序列冗余列表。 这些列表不包括出于生物信息学目的被视为不需要的条目,例如 包含太多严重错误,太多不完整或非规范氨基酸或同源性模型的条目。
WHY_NOT索引算法已被改编为处理许多新颖的数据库
1.1 新的主要资料库
高分子不是静态的。晶体结构中原子的位移可以在各种细节水平上建模。 B因子通常用于对单个原子的位移进行建模,而平移,自由度和螺杆旋转(TLS)参数则对原子组的位移进行建模。不幸的是,PDB文件的ATOM记录中B因子值的含义并不总是明确的。例如,对于成千上万的PDB结构模型,已经报告了“残差”(residual)而不是“完整”的B因子,已经对其B因子和TLS参数进行了改进。残余的B因子不包括TLS运动的贡献(29)。具有一致B因子(BDB)的PDB文件数据库(30)使PDB文件中的B因子表示同质化,以帮助依赖B因子的生物信息学和蛋白质工程应用(例如(31–36))。对于每个晶体学PDB条目,都有一个BDB条目。如果已报告了完整的B因子,则BDB中的文件与PDB中的文件完全相同,但是如果PDB文件中的元数据表明有必要,则它们包含根据PDB文件数据计算出的完整B因子。
1.2 WHAT GOOD IS BEAUTY, IF IT IS NOT TO BE SEEN?
PDB文件的主要用户是药物设计,分子生物学或生物燃料工程等领域的生物科学家。 这些研究人员通常并不了解使用PDB文件会带来的所有问题。 例如,请参见我们使用BDB解决的B因子问题。
X射线晶体学解决的所有结构普遍存在的问题是隐性描述对称性相关离子,水和配体(例如,PISA解决了不存在对称性相关大分子的问题(37)。 图1说明了此问题。
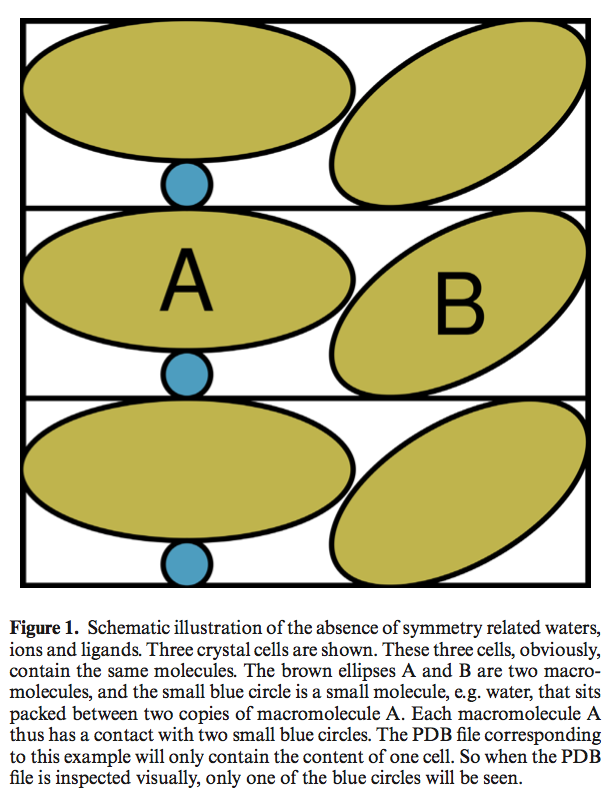
缺少与对称性有关的水,离子和配体的示意图。 显示了三个细胞。 显然,这三个细胞包含相同的分子。 棕色椭圆A和B是两个大分子,蓝色小圆圈是一个小分子,例如 水,堆积在两个大分子A副本之间。因此,每个大分子A与两个小蓝圈接触。 与此示例相对应的PDB文件将仅包含一个单元格的内容。 因此,当目视检查PDB文件时,只会看到一个蓝色圆圈。
图1所示的问题已在两个数据库中解决:
- 一个数据库保存PDB文件,其中包括与对称性有关的水域。
- 第二个数据库是从PDB文件中编译的,其中包含一些与对称相关的残基。
暴露在溶剂中的氨基酸侧链往往是可移动的,因此,在以X射线晶体学中的原子坐标建模为基础的电子密度图中无法观察到。 缺少侧链的可能性不大可能会引起注意,但可能会导致蛋白质结构软件出现问题或以其他方式干扰结构分析。 因此,我们建立了一个数据库,使用rotamer库在其中计算缺失的侧链,该数据库也是WHAT IF同源性建模模块的基础(38)
1.3 专门针对生物信息学的数据库
任何有抱负的蛋白质结构生物信息学家将需要编写或获取PDB文件解析器,然后才能开始从事预期的研究项目。编写一个解析器来解决PDB文件中所有问题的足够大的部分可能是一个主要的实际问题。在许多情况下,我们建立了大量的数据库来解决此问题。这些数据库包括:
- 每个残基的分子和溶剂可及的表面积,
- 处于四个状态(螺旋,链,匝,环 helix, strand, turn, loop)的二级结构,
- 晶体接触数,
- 扭转角和主链角。
- 还创建了盐桥和金属配位残基的列表。
最近,几个小组在从头算蛋白质结构预测领域取得了突破(39-41)。这些方法背后的思想是,在多序列比对中,残基位置i和j的变异性模式之间的相关性指示了这些残基i和j之间的接触。我们认为,这些研究可以从对什么是氨基酸间接触的更好定义中受益。为了支持该领域的研究,已经建立了一大批数据库,其中列出了接触氨基酸。在每个数据库中,接触以不同的方式定义(直接原子接触; Cα–Cα距离;仅侧链接触;等等)
1.4 新型可视化工具
CMBI数据库提供了大量与结构相关的信息。我们旨在在生物信息学家友好的文件中提供此信息。但是,并非所有用户都可以同样舒适地编写脚本来显示3D中每个残基的晶体接触数,从HSSP对齐创建熵变(EV)图或在PDB_REDO优化的结构模型中可视化结构变化。为了方便起见,并且只是为了加速蛋白质结构分析,我们创建了一组可视化工具。
现在可以在程序COOT(42)和CCP4mg(43)中直接获得优化的PDB_REDO结构模型及其相应的电子密度图,也可以在YASARA(44)中直接获得结构模型。 COOT还会显示所有重要结构更改的列表。 PyMOL( http://www.pymol.org/ )的插件可显示结构模型及其电子密度,可从PDB_REDO网站获得。所需的地图以CCP4(45)格式即时生成,并且还受到许多其他程序(例如Jmol(46))的支持。
BDB网站上组合的PDB和BDB B因子图使用户可以快速查看对PDB B因子所做的更正。
在我们最近开发的网络工具pdb-vis( http://www.cmbi.ru.nl/pdb-vis/ )的二级结构上,对称接触和几种可访问性表示形式与蛋白质序列一起以2D形式显示。 PDBsum(47)提供了许多互补的图片。 pdb-vis还提供了几种类型的3D结构场景,例如与晶体形成接触的残留物场景或金属和键合配体的特写镜头。 由于结构,可视化样式和视点存储在场景文件中,因此场景提供了突出显示特定结构特征或局部区域的便捷方法。 始终可以使用免费提供的分子图形程序YASARA_View(44)检查所有场景。
早就知道,MSA的EV分析可以阐明残基的功能作用(48,49)。 EV值可以根据HSSP对齐计算得出,并且可以在3D环境中进行解释。 我们开发了可视化工具VASE(路线,结构和熵的可视化)(50),可在单个浏览器窗口中连接三个组件的结构,路线和熵/可变性。 HSSP MSA中选定的残基位置在3D结构中进行了颜色编码,反之亦然(图2)。 Web界面还显示了表格中选定残基或EV图中所有残基的EV值(48)(图2插图)。

二、材料和方法
DSSP文件是使用新编写的DSSP 2.2.1生成的。 HSSP文件是使用HSSP 2.0生成的。 PDBFINDER文件当前具有9.0版。 当前,PDBREPORT是使用WHAT_CHECK 8.4(51)生成的,但是我们计划很快发布11.0版。 PDBREPORT数据库将相应更新。 PDB_REDO的最新版本是5.35。 PDB_REDO正在积极开发中,PDB_REDO数据库正在不断更新。 在撰写本文时,所有文件都是使用PDB_REDO版本5.00或更高版本创建的,这意味着所有结构模型都需要进行侧链重建和在需要时翻转肽平面方向(52)。 当前使用版本0.6.5创建BDB文件。 其他大多数数据库都是使用WHAT IF软件生成的(53)。
参考资料
- A series of PDB-related databanks for everyday needs . Wouter G. Touw, Coos Baakman, Jon Black, Tim A. H. te Beek, E. Krieger, Robbie P. Joosten and Gert Vriend.Nucl. Acids Res. (2015) 43, D364-D368 . https://academic.oup.com/nar/article/43/D1/D364/2435537
