【9.4.1】蛋白-配体相互作用指纹图谱(PLIF)
一、概述
蛋白-配体相互作用指纹图谱 (Protein Ligand Interaction Fingerprints ,PLIF)是一种通过指纹图谱的方法来描述配体和蛋白之间的相互关系的方法。氢键、离子相互作用、表面接触根据原始的残基来进行分类,然后被构建成代表蛋白-配体复合物的数据库的指纹图谱。
PLIF的输入数据来源广泛,主要包括X-ray crystal structures和对接结果。由于模拟蛋白质与配体的结合存在很多困难,使用指纹集体代表大型数据库中的蛋白质-配体相互作用是处理嘈杂且容易出错的数据库的一种有效方法。
通过PLIF产生的指纹能够被MOE中其他的指纹工具使用。标准的指纹工具,如聚类和区分子集可以应用于由PLIF生成的指纹。有一个专门的可视化界面,考虑每个指纹位的特定结构含义。
为了对相互作用指纹进行分类,蛋白质的每个氨基酸残基被分成几类。 以下是一些常见的相互租用:
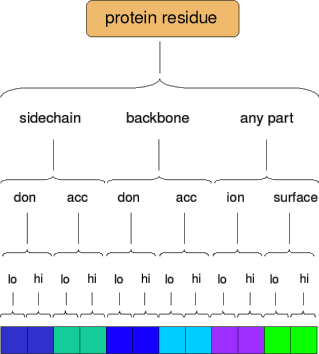
- lo代表low,弱的相互作用
- hi代表high,强的相互作用,用来设定阈值
一个残基可能参与两类相互作用:潜在(基于能量)的接触(Potential Contacts)和表面(patch)接触(Surface Contacts)。 对于潜在的接触,值是残基和配体中任何一对原子之间最强的相互作用的值,而对于表面接触,该值是残基和另一分子之间每种类型的总接触面积。
如果特定类别的相互作用没有被找到,或者没有通过阈值,则不会为该类别设置bits。 如果最强的交互通过较低的交互阈值,则设置低位fingerprint bit。 如果最强的交互通过较高的交互阈值,则低位和高bit都被设置。 因此,每个类别的位模式(bit patterns)可以分别取00,10或11的值。
一般来说,潜在的接触(Potential Contacts)用于表征一个小分子在明确定义的结合位点的结合,而表面接触(Surface Contacts)用于表征蛋白质-蛋白质界面。 每类相互作用的阈值分别基于能量值或表面积。
Potential Contacts
现在一个残基有可能参与potential interactions类型有9种:
- sidechain hydrogen bonds (donor or acceptor), 侧链氢键
- backbone hydrogen bonds (donor or acceptor), 主链氢键
- solvent hydrogen bonds (donor or acceptor), 溶剂氢键
- ionic interactions, 离子相互作用
- metal binding interactions 金属结合作用
- π interactions. π相互作用
Surface Contacts
表面接触相互作用(Surface Contacts)通过计算每个蛋白质链的溶剂暴露表面积,并对其进行补丁分析计算( Patch Analysis)来确定。对于每个残基,计算与另一个表面的总接触面积以及每个接触表面的Protein Patch类型。相互作用根据patches分成五类:疏水性的(hydrophbic,两个表面都是疏水性patches),带电荷的(charged,任一个表面是带电patch),部分疏水性的(partial hydrophobic,一个表面是疏水的,另一个没有patch类型),其他(other,两个表面都没有patch类型)和总数(total,所有接触区域的总和)。对于除total以外的所有类别,将额外阈值应用于 代表特定patch contact的total contact的百分比。例如,疏水性接触(hydrophobic contact)被定义为至少8 Å2且,还有残基总接触(total contact)面积的至少50%。此有助于避免重复计算包含多个patches的相互作用。
二、生成指纹图谱
在使用PLIF之前,有必要准备一个或多个蛋白质作为受体物种,以及一些具有结合构象的配体或蛋白质。 有三种主要方法可以解决这个问题:
- 将受体加载到MOE主窗口中,并提供包含分子类型字段的数据库,其中每个条目包含单个配体或蛋白质构型,其坐标对应于其在活性位点内的取向。
- 提供一个数据库,其中包含每个条目中包含配体和受体的分子类型字段。 在这种情况下,受体和配体必须按照系统管理器中指出的各自的残基类别进行识别。
- 提供一个具有两个独立分子类型的数据库,一个用于受体,一个用于配体,它们一起对应于结合的复合体。
当源数据来自刚性-受体对接结果时,第一种方法是最常用的,其中单个蛋白质结构适用于已计算结合构象的许多配体的许多姿态。 当源数据来自一系列晶体结构时,第二种方法是最常用的,其中每种晶体结构的受体都相似,但不一定相同。 第三种方法适用于精制受体结构不相同的柔性受体(诱导拟合)对接结果或蛋白质 - 蛋白质复合物数据库。 请注意,对于后两种情况,假定受体结构是对齐的并且具有共同的残基编号方案。
Open the MOE database, and select DBV | Compute | PLIF | Generate. The following panel will appear:
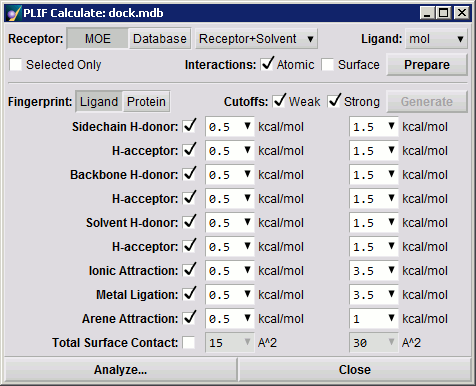
指纹选择提供了两种指纹模式之间的选择:配体或蛋白质。 上面显示的图像对应于默认的配体模式。 如上所述,指纹选择切换潜在和表面交互之间指纹位的显示,并为某些选项设置适当的默认值。 指纹生成计算将使用计算启动时在面板中可见的一组截断值进行。 下图显示了在Protein模式下显示的默认选项。
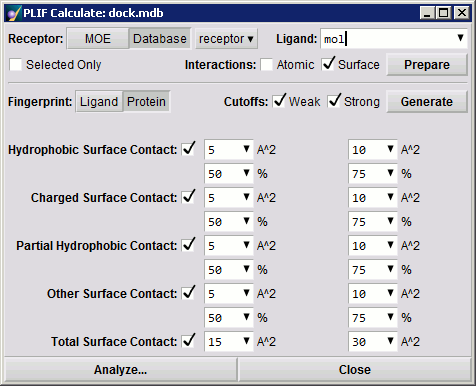
三、参数说明
Receptor/Ligand
对于基础数据库中的每个条目,计算原始相互作用的准备步骤需要定义配体和受体。 对于具有许多位置与单个受体相关的数据库,例如 对接运行时,通常只在数据库中存储配体。 在这种情况下,受体可以加载到MOE中,并且它必须适用于准备过程中正在处理的所有条目。
对于每个入口受体不同的复合物数据库,有必要将每个结合配体的受体储存在同一分子场内。 对于受体,选择MOE以使用当前加载的蛋白质作为要计算相互作用的所有数据库条目的受体。 如果蛋白质和配体原子都存储在数据库中,请选择数据库。
1.当MOE被选中的时候, 用于受体的原子集的选择:
- Receptor Atoms
- Receptor+Solvent
- Selected Atoms
- Selected Residues
- Selected Chains

默认的选项是 Receptor+Solvent.对于这种设置,重要的是确保只装载一种蛋白质,并且底层数据库中的所有配体都具有相对于该蛋白质的活性位点的3D坐标。
对于配体,必须选择数据库中的一个分子字段。 这个字段不会被生成交互数据或指纹所修改。
2.如果receptor type改为Database,那么必须选择分子类型的字段(直接右键)。 在从刚体对接结果创建数据库的情况下,使用专用字段PLIF_pronum将受体结构保存在数据库中,该专用字段是与用于每个条目的受体相对应的整数索引。 如果存在,该字段也可以被选作受体。

当受体是数据库中的分子类型字段时,还必须指定配体字段,并将其指示为文本框。 根据配体领域的选择,有三种可能的情况。
- 配体场与受体相同。 在这种情况下,受体和配体链将由它们的残基类别决定。
- 配体字段不存在。 在这种情况下,受体和配体如上所述测定,但从受体场中提取的配体将被写入新配体场。
- 配体场是与受体不同的分子类型场。 在这种情况下,两个区域中的链将按照受体和配体的规定使用。
Selected Only
如果选中此选项,则只有在数据库查看器中选择的条目才会用于生成交互或指纹。
Interactions
交互复选框控制是否计算不同类型的原始交互:
- Atomic: 计算原子能量值。计算基于能量的指纹(energy-based fingerprints)
- Surface: 计算表面接触面积(surface-surface contact areas)。 这些相互作用需要计算表面修补指纹(surface-patch fingerprints)。
默认情况下,原子相互作用适用于配体模式,表面相互作用适用于蛋白模式。 然而,通过计算两种类型的相互作用,可以在随后的步骤中产生两种类型的指纹。 特别是,如果在配体模式下使用总表面接触(total surface contact),则必须启用表面相互作用。 在受体蛋白质全都不同的情况下(即从数据库中的现场检索),计算所有条目的surface patches可以显着增加计算成本。
Prepare
该按钮启动所选原始交互的计算,这是生成指纹最耗费资源的部分。 原始交互被写入底层数据库,在那里可以检索它们以构建指纹。 如果蛋白质字段的定义表示MOE主窗口中的分子对象,则必须在按下此按钮之前打开它。 默认情况下,将考虑数据库中的所有条目,并仅重新计算那些没有交互数据的条目。
Cutoffs
默认情况下,每个交互类别可以占用多达两个交互位(interaction bits):低和高。 可以通过手动将最小阈值设置为不可能高的值来禁用这些位类型的生成,但为了方便起见,还可以通过取消选择Weak或Strong来禁用两位方案。
Generate
该按钮使用先前计算的原始交互数据来生成指纹位(fingerprint bits)。 默认情况下,重建指纹方案,并在构建时考虑所有数据库条目的贡献。 指纹计算完成并写入数据库后,设置面板将自动关闭,并由结果面板替换。
Analyze
如果已经计算出某些或全部蛋白质配体相互作用指纹,则可以使用此按钮打开结果面板。
Protein-Ligand Fingerprints
以下选项可用于配体模式下的指纹生成。 如果选择了这些指纹位中的任何一个,则必须计算原子相互作用。
- Sidechain H-donor |H-acceptor :每个残基最多可以分配2个作为氢键供体的蛋白质侧链原子反应的指纹位,超过2个作为氢键受体的蛋白质侧链原子反应的指纹位。(Up to two fingerprint bits per residue can be assigned for interactions between protein sidechain atoms acting as H-bond donors, and two more fingerprint bits for protein sidechain atoms acting as H-bond acceptors. )为了方便起见,可以通过取消选中复选框来禁用这些类型的交互的生成。 最低阈值默认设置为低位为0.5千卡/摩尔,高位为1.5千卡/摩尔,并且可以根据需要进行修改。
- Backbone H-donor |H-acceptor : 每个残基最多可以分配2个作为氢键供体的蛋白质骨架原子反应的指纹位,超过2个作为氢键受体的蛋白质骨架原子反应的指纹位。
- Solvent H-donor |H-acceptor : 每个残基最多可以分配2个作为氢键供体的蛋白质溶剂原子反应的指纹位,超过2个作为氢键受体的蛋白质溶剂原子反应的指纹位。
- Ionic Attraction: 每个残基最多可以指定两个指纹位,用于残基中某些带电原子与配体上的一些带相反电荷的原子之间的离子相互作用。
- Metal Ligation : 每个残基最多可以指定两个指纹位用于金属相互作用。 最低阈值默认设置为低位为0.5千卡/摩尔,高位为3.5千卡/摩尔。
- Arene Attraction: 每个残基最多可以指定两个指纹位用于相互作用。 最低阈值默认设置为低位为0.5千卡/摩尔,高位为1千卡/摩尔。
Protein-Protein Fingerprints
以下选项可用于Protein模式下的指纹生成。 如果选择了这些指纹位中的任何一个,则会计算表面交互作用。
- Hydrophobic Surface Contact:每个残基最多可以指定两个指纹位,用于每个表面上疏水斑块(hydrophobic patches)之间的接触。 第一列包含弱相互作用的截断值,其包括:两个贴片(patches)之间的最小接触面积,与另一分子接触的残基总表面的百分比的patch contact最小百分数。第二列包含强相关所需的minimum contact area和total contact百分比。
- Charged Surface Contact : 每个残基最多可指定两个指纹位,用于在任一表面上都包含带电补丁的接触点。 如上所述,强和弱截断。
- Partial Hydrophobic Contact :每个残基最多可以指定两个指纹位,其中一个表面是疏水(patch)贴片的一部分,另一个表面不是任何贴片(patch)的一部分。 如上所述,强和弱截断。
- Other Surface Contact : 每个残留物最多可以指定两个指纹位,用于其中任何表面都不是任何patch的一部分的表面接触。 如上所述,强和弱截断。
- Total Surface Contact :Up to two fingerprint bits per residue can be assigned for the total area per residue in contact with the other surface, independent of any patches on either surface。 对于这种相互作用,只需要强和弱相互作用的最小表面积,因为分数值必然是100%。 total surface contact在配体和蛋白质模式下均出现在面板中,但是在任一情况下指的是相同的指纹位。
四、Using Fingerprints
在生成蛋白质配体相互作用指纹并将其写入数据库之后,FP:PLIF将填充数据,并且指纹可用于通用MOE指纹功能,例如聚类和多种子集计算。 本页面的其余部分介绍了专门为使用PLIF而设计的可视化和衍生工具。
查看PLIF结果 , 打开MOE数据库, 选择 DBV | Compute | PLIF | Analyze
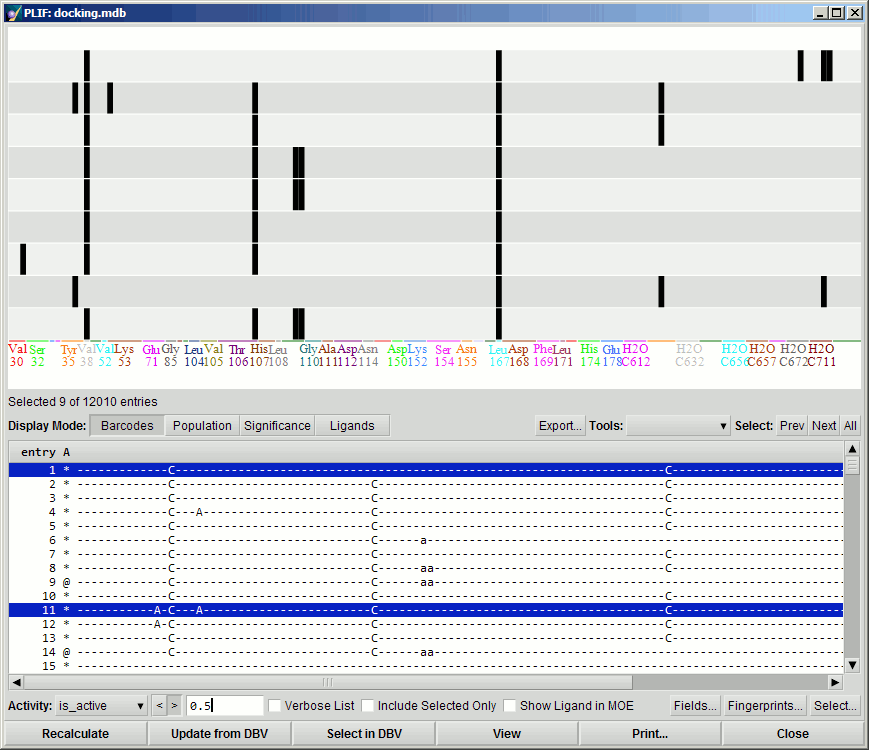
结果面板分为两个主要部分:
- Graphic display (top),它反映了当前选定条目的特定视图。
- Entry list (center), 它显示列表框中的所有指纹和其他字段,允许选择它们。 各种显示选项会影响条目列表中显示的字段,但至少会影响显示可见指纹位的组成。
每个指纹位用一个字符表示以表示其含义:
- bit not set
D sidechain hydrogen bond donor
A sidechain hydrogen bond acceptor
d backbone hydrogen bond donor
a backbone hydrogen bond acceptor
O solvent hydrogen bond
I ionic attraction
M metal ligation
R arene attraction
H hydrophobic surface contact
Q charged surface contact
P partial hydrophobic contact
X other surface contact
C total surface contact
它还配有许多选项和按钮,控制演示和与底层数据的交互。
Display Mode
显示在面板顶部的图形显示包含基于当前在条目列表中选择的条目以及所选显示模式的表示。 可用的显示模式是Barcodes, Population, Significance 和 Ligands ,其中每个都在随后进行描述。 Ligands显示模式仅适用于查看在配体模式下生成的PLIF。
Barcode Display
“条形码”显示将选定的条目和选定的指纹表示为矩阵(每个条目一行,每个指纹位一列),其中一组位被绘制为黑色矩形:
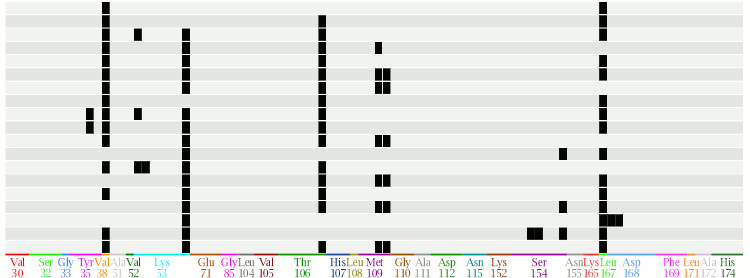
X轴显示对应于每组指纹bit的残基编号,并且用任意颜色序列编码。 可用时提供关于组成残基的氨基酸代码的信息。 如果所涉及的所有蛋白质在相互作用方面具有相同的残基组成,则在残基ID号(例如His,Asp,Met等)上绘制3个字母的氨基酸编码。 如果有残基组合物的混合物,则使用单字母代码(例如H,D,M等)。 如果特定的交互指纹涉及两种不同类型的残基,则两个字母将被连接(例如HD)。 如果涉及三种或更多类型的残基,说明将替换为*。 如果残基编码与天然存在的氨基酸不相符,则描述为?。
Population Display
总体直方图显示每个选定指纹bit的选定条目之间的出现频率。 X轴的构造方式与条形码显示方式相同,而Y轴显示的黑色条形图显示了相应的bit:
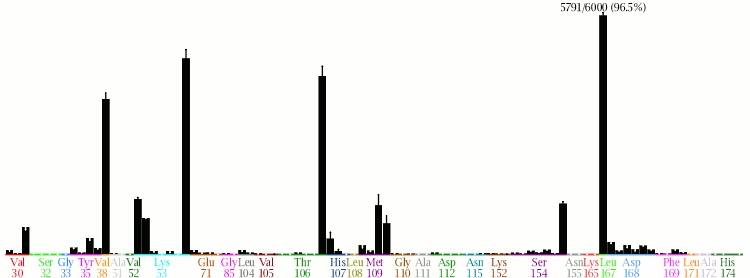
在最大的条的顶部显示:位数,涉及的条目数以及具有该位特征的条目的百分比。
当选择population 显示时,会有一个称为Normalize的附加选项。 如果选中此项,则将基准峰值缩放为占用图形中的最大大小。 如果没有被选中,那么基峰将按其出现的条目的比例按比例缩放(例如,如果选择了100个条目,并且在50个条目中出现最多人口位,那么最大的条将约为 图片大小的一半)
如果有activity信息可用,则每个频率栏都会进一步用活性物质与非活性物质过量的定性指示进行注释。 包含位(a)的活性成分的比例与总体活性成分的比例(b)进行比较。 如果a> b,则通过在条上方绘制黑线来指示其更大的程度。 如果a <b,则通过在该条顶部下方绘制一条反白(白线)来指示其更少的程度:

高度和过度的组合通常表明相应的交互对于活动很重要。 如果相互作用频繁发生,但在活性化合物中不常见,而不是非活性化合物,则不太可能将其作为活性结合配体的显着特征; 类似地,如果活性化合物中的相互作用比非活性化合物中的相互作用更频繁,但其总体发生率很少,则不太可能成为决定性因素。
Significance Display
重要性图与人口直方图相关,除了构建条的高度以指示每个指纹位是活动的重要先决条件的程度。 显示的不同之处在于,条可以具有正值或负值,并且可以使用灰度色调
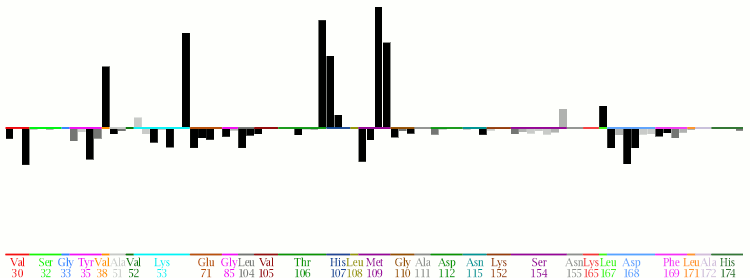
计算略。。
Ligand Display
配体显示图显示了所选条目的配体,以2D图示形式,具有代表结合构象的并行比对,并且进一步用其参与的相互作用的指示进行注释
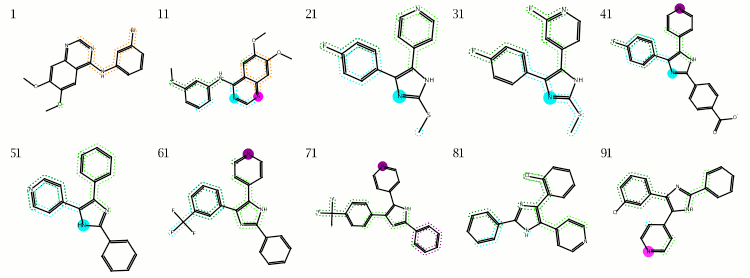
配体的三维坐标首先使用二维描绘方法[Clark 2006]转化为二维坐标,具有平坦化约束,这迫使布局算法损害纯粹的美学,以便找到类似于3D形式的平面表示[Clark2007年]。 一旦所有的姿势变平,就会叠加到原来的3D位置上,然后共同旋转到页面上。 为了组成这个图形,它们被并排显示。
只有当所选条目的配体坐标相对于相同蛋白质,或者相对于叠加在公共空间上的多种蛋白质时,配体的相对布局才有意义。
Export
允许使用许多不同的格式将图形显示的当前状态导出为图片
Tools
点击后,提供一系列专为PLIF使用而设计的外部工具。
Select
选择按钮“上一个”(Prev),“下一个”(Next)和“全部”(All)用于方便地选择主要条目列表中的条目组。 当“全部”(All)按钮始终选择所有条目时,“上一个”和“下一个”的行为取决于条目的排序顺序。 默认情况下,条目按照数据库中出现的顺序进行排序,因此这两个按钮都将选择一个条目,该条目位于当前选择之前或之后。 如果提供了排序标准,这些按钮将选择具有相同主索引值的前一个或下一个不同的组。 例如,如果数据库按名称为id的字段进行排序,该字段的值为1,2和3,则按Next按钮将循环显示具有此值的条目。
Activity
对于一些可视化和计算目的,有必要将条目定义为具有二进制活动分类,其评估为1(有效)或0(无效)。 如果这些信息不可用,则不会使用活动。 活动字段由三部分组成:字段,比较和值。 字段列表包含底层数据库中的数字字段。 比较可以大于或小于,并且该值可以是任何数字。
为了方便起见,结果面板检查是否有一个名为is_active的数字字段,如果是,选择此字段并定义条目在数值大于0.5时处于活动状态,如果所有条目都包含 活动数值为1,非活动数值为0。 如果活动以其他形式存储,如pKi或IC50,则可根据需要定制阈值。 如果没有任何字段包含活动,请选择标记为<无>的字段。
请注意,在条目列表中,分类为活动的条目在列标题A下标有"*“符号
Verbose List
该选项控制条目列表中显示的详细程度。 如果打开(默认),位模式将被分成残基组,每个残基组在列标题中都用残差编号标记:
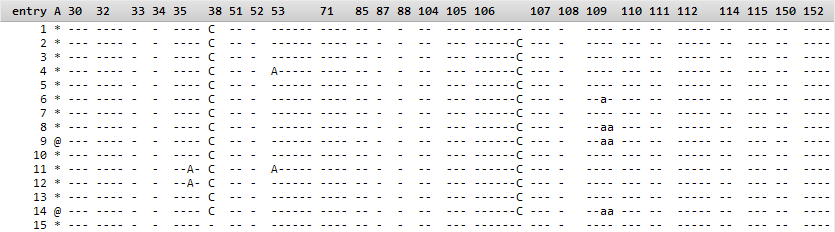
如果关闭,则每个条目的指纹都以简洁的字符填充形式显示,没有标签来指示哪些残基与每个位相关联:
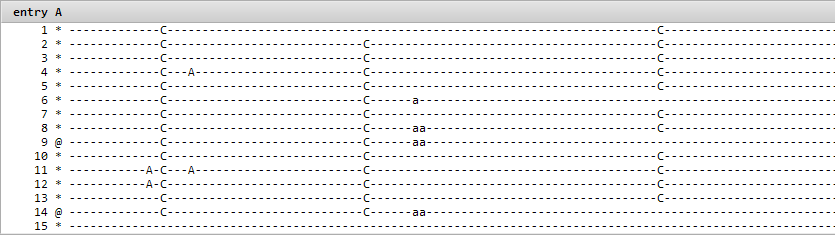
Include Selected Only
打开时,条目列表将被折叠以仅显示选定的行。 切换关闭时,选择将保留,同时展开条目列表以显示底层数据库中的所有条目。 按下来自DBV的更新将更新展开或折叠视图中的数据和选择。 当没有选定的条目时,所有条目都被认为是被选中的。
提示要进一步优化条目列表,请选择合拢显示内的条目,然后切换两次该选项。
Show Ligand in MOE
如果启用或每次更改,则在主MOE窗口中创建与条目列表中所选条目相对应的配体,并通过链条颜色着色。 当条目选择改变时,或者该选项关闭,或面板关闭时,这些分子将被移除。 此选项仅适用于查看在配体模式下生成的PLIF。
Fields…
显示“选择”对话框。
Recalculate
显示指纹设置对话框,该对话框可用于修改数据库中的指纹。
Update from DBV
重新从源数据库读取底层数据,并替换当前内容。 如果数据库的内容已被外部手段改变,则这是必要的,并且期望结果面板反映这些改变。 这也将从DBV复制选择状态,以便在PLIF显示中选择相同的条目。 如果选中仅包含选定内容,则只显示选定的条目。
Select in DBV
将条目列表中的选择与相应的数据库查看器同步。 如果尚未打开数据库查看器,它将被打开。
View
对于条目列表中的选定条目,配体和相应的蛋白质将在MOE主窗口中创建,删除任何先前存在的内容。 然后使用配体相互作用面板查看整体。 如果有多个选定的条目,则默认使用并发对齐方法。 注意:如果指纹是在配体模式下生成的,则会出现此选项。
Epitopes…
列出可用于浏览姿势群集的表位分析面板。 注意:如果指纹是在蛋白质模式下生成的,则会出现此选项。
Print…
调出标准的MOE打印预览对话框,在内容部分有以下选项:
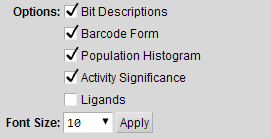
如果Bit Descriptions打开,则页面布局以指纹方案的摘要开始,所述指纹方案提供指纹索引,残差ID和每种类型的交互类型。 字体大小可根据需要进行调整。
如果选中Barcode Form, Population Histogram, Activity Significance or Ligands,则这些图形显示类型中的每一种都将包含在页面布局中。 它们的绘制方式与面板相同。
Field Selection
默认情况下,主条目列表显示每个条目的索引,活动和指纹位,并按数据库顺序对其进行排序。 可以在此显示中显示其他字段,也可以按数字字段排序:
显示的字段下方列出的字段可以打开或关闭,并且会在主条目列表中相应更新。
Fingerprint Selection
指纹位组可以根据需要选择或取消选择,这可以清除主要条目列表和图形显示中的空间:
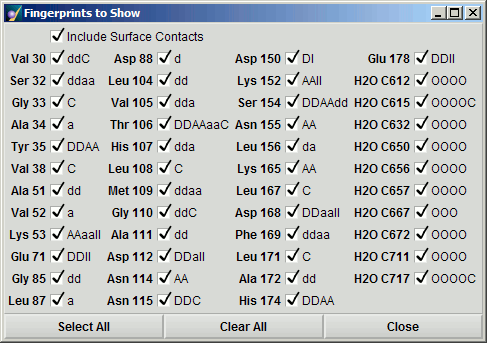
每个复选框都对应一个特定的残留ID。 如果未选中,则不会显示相应的指纹位,并且可用更多空间可视化剩余的位。
Entry Selection by Fingerprint
使用选择面板,可以根据选定指纹位的存在选择主列表中的条目:

必须在此对话框的主列表中选择指纹说明符的某些组合。 对于指纹方案中的每个残基,有一个标记为“任何相互作用”的条目,如果设置了与该残基相关的任何指纹位,则转换为正匹配。 对于每种不同的位类型,都提供了一个附加条目。
根据所选指纹和Logical Operation的值,每个条目匹配或不匹配。 在OR情况下,如果任何选定的指纹适用,则条目匹配。 在AND的情况下,只有在所有选择的指纹适用的情况下,条目才匹配。
主要结果面板中对选择的更改随后将通过Current Selection 状态进行修改。 如果设置为“清除”,则无论条目是否匹配,选择都会被完全替换。 如果Add To,则选择任何匹配的条目,并且之前选择的任何内容仍然如此。 如果Subtract,那么匹配的任何条目将从选择中删除,如果它以前被选择的话。 如果Subtract,那么一个条目必须匹配并且已经被选择以保持如此。
Fingerprint Tools
本节介绍的工具不是PLIF的组成部分,但可以通过PLIF结果面板的工具菜单方便地访问。 每种工具都利用现有的指纹数据,并可能派生出特定的属性,或者修改数据库的内容
每个工具都带来一个独立操作的面板。 由外部工具对数据进行的任何更改都不会自动反映到相应的PLIF结果面板中,因此可能需要使用Update from DBV按钮才能刷新数据视图。 类似地,需要提供一组条目的工具不从PLIF结果面板获得该选择,而是可以使用数据库查看器中的选择状态,其可以使用Select in DBV按钮来同步。
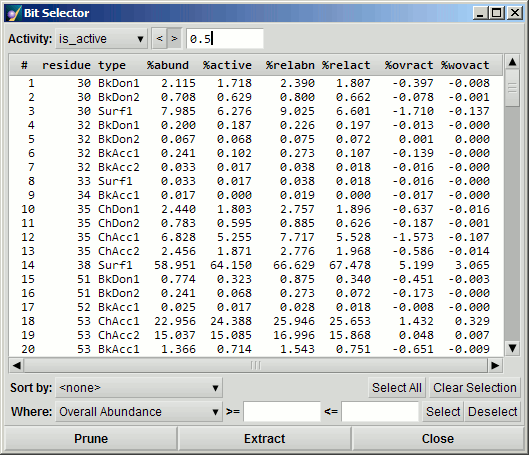
Bit Selector
Pose Selector
交互指纹的一个可能用途是通过研究一些配体的结合模式,并在上下文中确定它们的哪些假定姿势最可能是正确的,从而消除计算对接算法产生的许多姿势 的所有配体。 姿态选择工具使用配体的活动数据来发现指示活性的共同结合基序。 最常见于活性配体的相互作用模式应该包含一个结构基序,如果发现它,将导致消除许多多余姿势的方法。
这种集体方法与传统的对接评分函数非常正交,通常认为每个配体都是独立的,通常会产生许多姿势,得分相似,但具有完全不同的结合方向。 然而,该工具所使用的方法基于以下假设:(1)仅存在一种结合模式,并且(2)对于大多数配体,存在至少一个表明存在重要相互作用的姿势。
该算法通过假设一系列的形式假设来操作:
ACTIVE = {contains bits A} AND {does not contain bits B}
其中A和B是任意位组。 如果配体的任何成分被假设分类为活性,则认为配体被预测为活性的。 每个假设的效力取决于:
SCORE = {% true positives} + {% true negatives} - {% false positives} - {% false negatives}
假设最初是随机生成的,其中集合A被加权以选择在活性化合物中经常观察到的位。 在预定的时间之后,最好的随机确定的假设经历迭代改进以改善功效评分。
如果最终假设的得分接近零,则意味着没有发现区分活性物质和非活性物质的模式。 如果最终得分接近100%,那么所选主题具有近乎完美的区分活性物和非活性物的能力。
对于属于活动配体的每组姿势,如果只有一个姿势满足该假设,则该姿势被选择为最可能的活动姿势。 如果没有一个或多个姿势满足该假设,那么与集合A的Tanimoto相似度最高的那个被保留,或者在平局的情况下保留第一个条目。
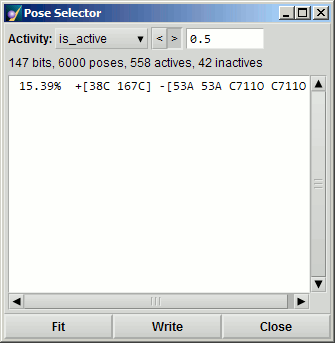
当选择Fit按钮时,选择活动姿势的算法开始。 完成后,Write按钮变为可用,结果可写入底层数据库。 如有必要,将创建一个数据库字段:PLIF_apose,对于被分类为非活动的任何条目,其值将为0。 对于来自活性配体的每组姿势,一个条目将具有1的值,而其他条目将具有0的值。因此该数据库将被分成三个部分:活动姿势,无活性姿势和无活性配体。
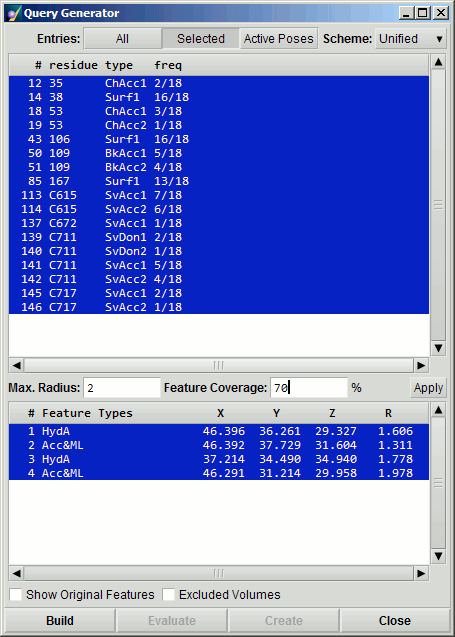
Query Generator (Available only for small-molecule ligands.)
由于配体相互作用指纹直接来源于实际结构信息,因此可以随时将指纹数据回溯至实际的3D相互作用。 查询生成器工具的工作原理是,适度选择具有均匀相互作用指纹集合的姿势最有可能具有同类的药效特征点集合。 药效学特征点可以根据它们与哪些残基相互作用进行分组和过滤,并且具有足够紧密分组的那些可以转化为药效团查询特征。
由此产生的查询特征集合的严密性高度依赖于所选姿势的一致性。 尽管并非所有以这种方式生成的查询都适合筛选无结合模式的构象数据库,但当以绝对模式应用于已经对接到蛋白质中时,或作为进一步对接计算的限制时,所得查询是非常有效的方法 利用PLIF收集的交互信息。
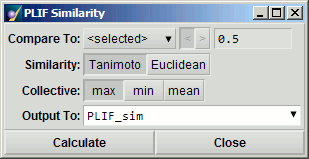
Similarity
使用交互指纹比特来比较数据库的选定子集与其余所有内容之间的相似性可以是以各种方式估计属性或分离数据的有用方式。 例如,如果已经从已知活性配体的晶体结构获得了一个或多个条目,则剩余数据的活性可以通过比较它们的指纹与其余条目之间的Tanimoto系数来估计。
参考资料:
- MOE: Protein Ligand Interaction Fingerprints
- file:///Applications/moe2018/html/apps/plif.htm
