【4.5.2】抗体编号系统(CCG/Kabat/Chothia/IMGT 与ANARCI)
更系统的说明见该博文
- 对抗体氨基酸序列进行编号用以鉴定等同位置。
- 这些注释对于抗体序列比较,蛋白质结构建模和工程是有价值的。
- 存在多种不同的编号方案,它们在用于注释残留位置的命名法,它们对位置等同性的定义以及它们在不同科学学科中的普及性方面存在差异。
抗体和T细胞受体(TCR)的可变结构域含有这些蛋白质的主要结合区域。 这些可变序列与编号方案的比对允许等同的残基位置被注释并且可以比较不同的分子。编号是免疫信息学分析和治疗分子合理工程的基础(Shirai,2014)。
已经提出了几种编号方案,每种方案都受到不同免疫学科的科学家的青睐:
- Kabat方案(Kabat等,1991)是基于相同结构域类型的序列之间的高序列变异区域的位置而开发的。 它对抗体重(VH)和轻(Vλ和Vκ)可变结构域的编号不同。
- Chothia的方案 ( Al-Lazikani,1997) 与Kabat的方案相同,但校正了在第一个VH互补决定区 (CDR)周围插入注释的位置,使其对应于结构环。同样,增强型Chothia计划(Abhinandan和Martin,2008)对插入位置进行了进一步的结构修正。
- 与这些类似Kabat的方案相反,IMGT(Lefranc,2003)和AHo(Honegger和Plückthun,2001)都定义了抗体和T细胞受体(TCR)(Vα和Vβ)可变结构域的独特方案。 因此,可以容易地在域类型之间比较等效残基位置。 IMGT和AHo在他们注释的位置数量(分别为128和149)以及他们认为indel发生的位置上有所不同。
存在可以应用每个编号方案的单独的在线界面:Kabat,Chothia和Enhanced Chothia通过Abnum(Abhinandan和Martin,2008); IMGT通过DomainGapAlign(Ehrenmann,2010); 和AHo通过PyIgClassify(Adolf-Bryfogle等,2015)。 包括下面会提到的
一、概述
-
Kabat Numbering和Eu Numbering是两回事。
-
Eu是指上个世纪60年代末(1968)Gerald M.Edelman(因抗体序列结构方面的研究得过诺贝尔奖)等人分离纯化得到一个人IgG1免疫球蛋白,命名为Eu,并测定了其氨基酸序列并遍了号,就被沿用为Eu Numbering,现在已经很少使用了。
-
Kabat Numbering应该是在Eu Numbering的基础上结合更多的Ig序列进行了改善后的编号系统,也是迄今仍广为使用的抗体序列编号系统。
-
现在有几套抗体编号系统在并行使用,经典的Kabat,Chothia编号,IMGT编号;还有一些使用面较小的编号系统。
-
各编号系统的对比:http://www.imgt.org/IMGTScientificChart/#B
-
下图对比了抗体C domain的IMGT、kabat及Eu编号:http://www.imgt.org/IMGTScientificChart/Numbering/Hu_IGHGnber.html
二、编号工具
2.1 MOE中操作
SEQ – 粘贴序列 – annotate - antibody – CCG/Kabat/Chothia/IMGT
2.2 ANARCI
网页版:
http://opig.stats.ox.ac.uk/webapps/sabdab-sabpred/ANARCI.php
本地化以后的命令:
本地化安装:
需要先安装好 hmmer
git clone https://github.com/oxpig/ANARCI.git
cd ANARCI
python setup.py install
使用:
results = anarci.anarci([('seq_name', seq)], scheme="kabat", output=False)
print results
# Unpack the results. We get three lists
numbering, alignment_details, hit_tables = results
2.3 网页工具
三、ANARCI
ANARCI是一种对抗体和T细胞受体氨基酸可变区序列进行分类和编号的工具。 它可以用五种最流行的编号方案来注释序列:Kabat,Chothia,Enhanced Chothia,IMGT和AHo
ANARCI将单个或多个氨基酸蛋白质序列作为输入。 该程序使用HMMER3将每个序列与一组隐马尔可夫模型(HMM)对齐(Eddy,2009)。 每个HMM描述特定物种(人,小鼠,大鼠,兔,猪或恒河猴)的结构域类型(VH,Vλ或Vκ,Vα或Vβ)的推定种系序列。 然后使用最重要的对齐来应用五种编号方案中的一种。
3.1 创建HMM
每个物种的每种域类型的HMM都是按以下方式构建的:
1.从IMGT / Gene数据库(Giudicelli,2005)下载每种可用物种和结构域类型的v基因区段的预比对(缺口)种系序列。 还下载了j基因区段的序列。 使用Muscle(Edgar,2004)将这些序列与单个参考序列比对,gap重重的罚分(-10)。
2.采用相关v和j基因区段的所有可能的成对组合以形成一组推定的种系结构域序列。 对于VH结构域,不包括d基因区段。 对齐中的每个位置代表IMGT编号方案中的128个位置之一。
3.从对齐中,使用hmmbuild工具构建HMM。 这里,指定'-hand’选项以保留对齐的结构。
总共建立了24个HMM,描述了来自六个不同物种的可变域类型。 使用hmmpress将这些HMM组合成单个HMM数据库
3.2 编号输入序列
使用hmmscan将输入序列与每个HMM对齐。 如果对齐的bit-score小于100,则不会进一步考虑。 该阈值证明可有效防止其他IG-like蛋白的错误识别。 否则,最重要的对齐将其域类型分类,并且对齐被转换为选择的编号方案。
ANARCI可以将Kabat,Chothia,Extended Chothia,IMGT或AHo方案应用于VH,Vλ和Vκ结构域序列。 IMGT和AHo方案也可以应用于Vα和Vβ结构域序列。 在可能的情况下,HMM对齐中的位置用编号方案中的等效位置注释。 在对齐和编号方案之间没有直接等同性的区域中,序列根据相应出版物中描述的规范进行编号。 例如,取决于CDRH1的长度,VH序列的HMM比对位置40等同于Kabat位置31-35X。
对于每个编号的域,编写标题以描述最重要的比对,包括物种,链类型和比对范围。 编号后面是列分隔格式。 或者,用户可以将ANARCI作为Python模块导入,并在自己的脚本中使用API
3.3 注
- 能批量处理数据,速度还很快
- 支持网页化
四、CDR定义
这篇文章很赞: Martin A C R . Protein Sequence and Structure Analysis of Antibody Variable Domains[M]// Antibody Engineering. Springer Berlin Heidelberg, 2010。 写得很清楚,有空再整理出来。
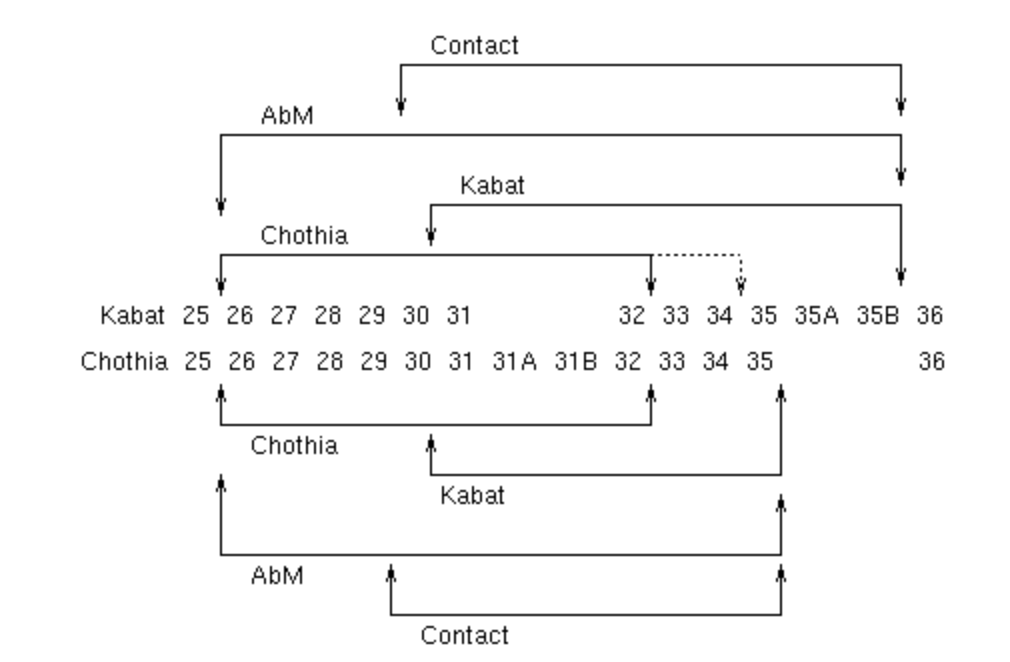
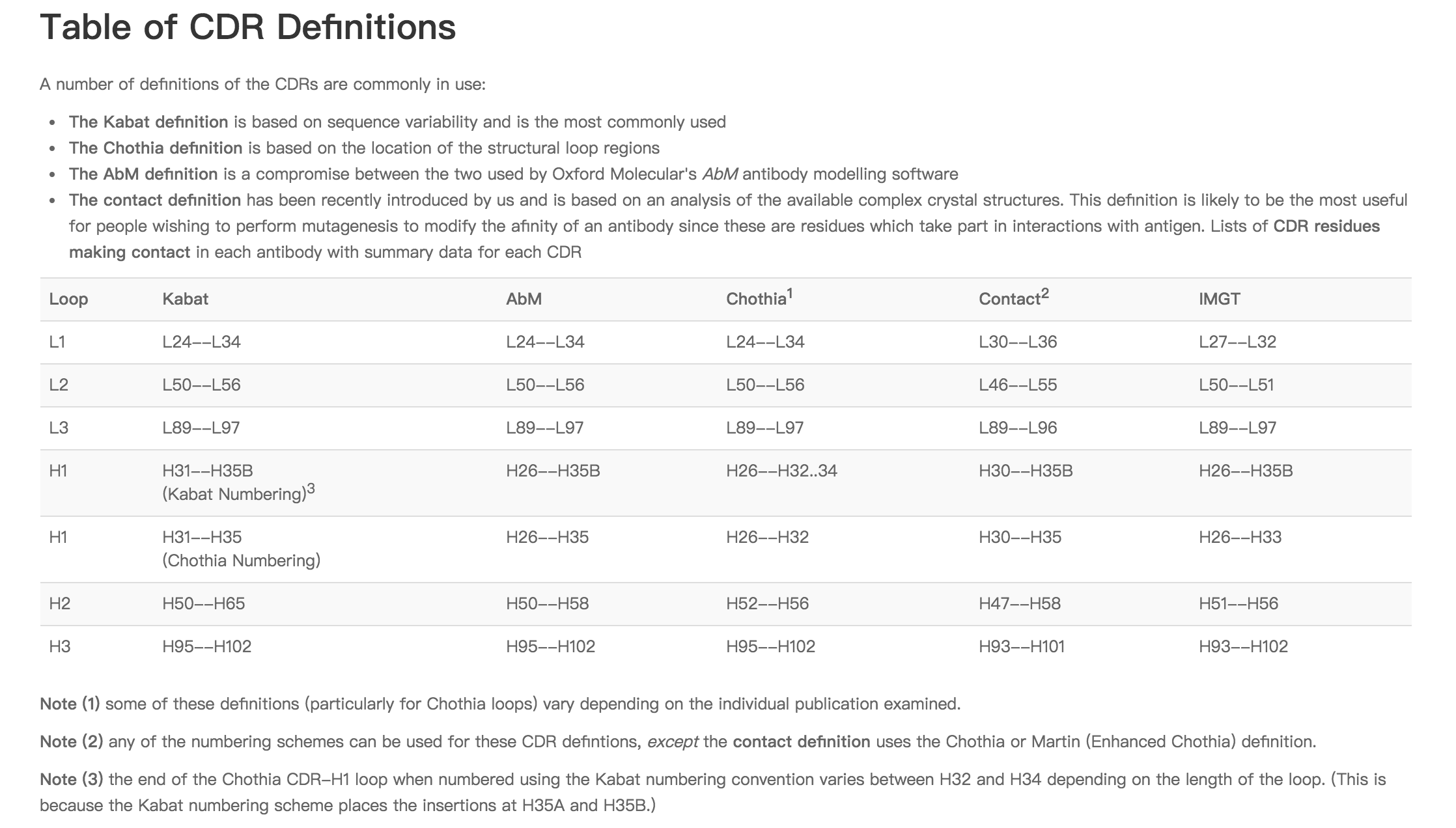
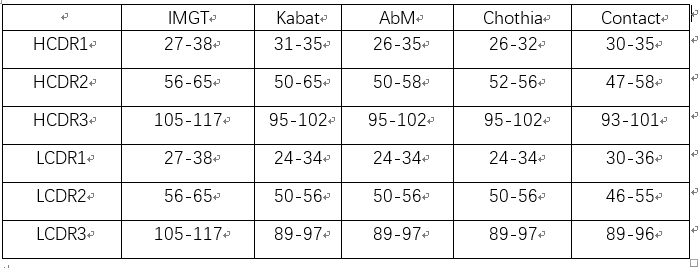
Chothia, based on the location of the structural loop regions. (Note that Chothia definition has varied between different papers – this is the one judged to be most relevant. AbM Compromise between the Kabat and Chothia. Whitelegg N & Rees AR, Protein Eng. 13(2000):819-824 Methods Mol Biol. 248(2004)51-91). Contact: Based on an analysis of which residues contact antigen in crystal structures. (MacCallum RM, Martin ACR & Thornton JM, J. Mol. Biol. 262(1996)732-745).
参考资料
- http://www.dxy.cn/bbs/thread/35439553#35439553
- Dunbar J and Deane CM. ANARCI: Antigen receptor numbering and receptor classification. Bioinformatics (2016)
- http://bioinf.org.uk/abs/info.html
- Martin A C R . Protein Sequence and Structure Analysis of Antibody Variable Domains[M]// Antibody Engineering. Springer Berlin Heidelberg, 2010 。 https://citeseerx.ist.psu.edu/viewdoc/download?doi=10.1.1.461.3807&rep=rep1&type=pdf
- https://plueckthun.bioc.uzh.ch/plueckthun/antibody/Numbering/NumFrame.html
