9.2.多元因子分析(MFA)及其在R中实现
多元因子分析(multiple factor analysis,MFA)是主成分分析(PCA)的扩展,旨在描述多组变量集的相互关系(Escofier and Pagès, 1990)。通过平衡每组变量的影响,综合各组变量集的贡献确定对象之间的距离,分析由几组变量共同描述的对象特征,以及观测变量之间的综合关系。
一、MFA简介
MFA是一种探索性的对称排序方法,不是因果关系的假设检验,变量集之间的关系通过某种类型的相关性衡量。
MFA允许输入多组不同类型的变量集:
- 各组变量集所含变量数量允许不同,变量的性质(分类或定量)在各变量集之间也允许各异,
- 但各变量集内部所有变量的性质必须相同,且所有变量集的对象必须相同。
1.1 MFA的计算步骤如下:
首先标准化各组变量集的所有变量。
对标准化后的各变量集执行特征分解,全为定量变量的变量集执行主成分分析(PCA),全为分类变量的变量集执行多重对应分析(MCA)。
将各组变量集中的变量除以各自PCA(或MCA)第一轴的特征根获得归一化的变量集,并将所有归一化的变量集都汇总到一个大数据表中,对该表执行全局PCA分析。
将每组变量投影到全局PCA排序图上,通过对象和变量的排序图评估数据集的共同结构和差异。

(Abdi et al, 2013)
1.2 应用领域
MFA已被广泛用于多种领域,例如:
-
调查分析,对象为被调查的个体,变量为调查问题,问题可按主题划分为变量集;
-
感官分析,对象是食品,第一组变量包括味觉变量(甜,苦等),第二组包括化学变量(pH值、葡萄糖速率等);
-
生态学分析,对象为观测地域,第一组变量描述了环境特性,第二组描述物种数量;
-
多组学分析,对象是样本,不同的变量集可以为转录组、蛋白组、代谢组等测量数据;
-
时间序列,可以将各变量集定义为每个变量在不同时间的测量值;
二、例子
本篇简介R包FactoMineR的MFA方法,以及结合factoextra包实现可视化。分析中将展示如何揭示重要的变量,这些变量对解释数据集的方差起最大的作用。
2.1 数据集
FactoMineR包的内置数据集wine,记录了21种葡萄酒的信息。
library(FactoMineR)
#数据集,详情 ?wine
data(wine)
head(wine[1:6])
该数据集中,每一行代表一种葡萄酒,共21行。
每一列代表了葡萄酒的属性信息,共31列。其中前2列是分类变量,label(Saumur、Bourgueil或Chinon)代表了葡萄酒的产地,soil(Reference、Env1、Env2或Env4)代表了植物的土壤环境;第3-31列是定量变量,代表了葡萄酒各类品质特征的打分。
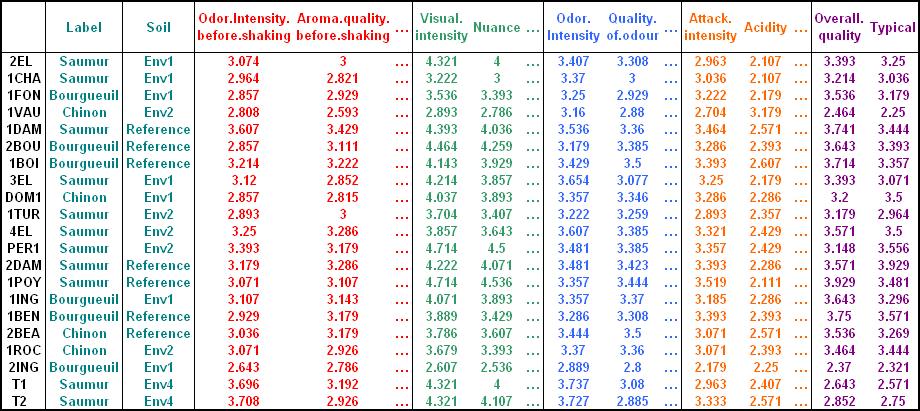
整体而言,对于数据集中所有代表葡萄酒属性的变量,可将它们组合为6组变量集,包含1组分类变量和5组定量变量:
-
第1-2列,共2列,包括葡萄酒产地等属性,该变量集代表葡萄酒“来源属性”(origin);
-
第3-7列,共5列,包括摇动前的气味强度、香气质量、果味等属性,该变量集代表葡萄酒“摇动前的气味属性”(odor before shaking);
-
第8-10列,共3列,包括视觉上的区分度等属性,该变量集代表葡萄酒“视觉属性”(visual);
-
第11-20列,共10列,包括摇动后的气味强度、香气质量、果味等属性,该变量集代表葡萄酒“摇动后的气味属性”(odor after shaking);
-
第21-29列,共9列,包括酸度等属性,该变量集代表葡萄酒“味觉属性”(taste);
-
第30-31列,共2列,包括质量等属性,该变量集代表葡萄酒“质量属性”(quality)。
接下来期望根据这些组合属性,对21种葡萄酒的品质进行综合评价,以及寻找它们之间的共同特征或差异,即可通过MFA来完成。
2.2 R包FactoMineR的MFA
FactoMineR中,MFA通过MFA()函数执行。
#执行 MFA,详情 ?MFA
#其中,摇动前后的气味、视觉和味觉作为 active groups,来源和质量作为 supplementary groups
res.mfa <- MFA(wine, group = c(2, 5, 3, 10, 9, 2), type = c('n', 's', 's', 's', 's', 's'),
name.group = c('origin', 'odor_before_shaking', 'visual', 'odor_after_shaking', 'taste', 'quality'),
num.group.sup = c(1, 6), graph = TRUE)
summary(res.mfa)
其中,group指定列的集合作为变量集(数据集的列需要提前按组排列好)。
type指定变量标准化方法,与各变量集一一对应。MFA的目标是整合描述相同观察对象的不同变量集,由于各变量的量纲不一致,为了平衡每组变量的影响(或者说为了使各变量集具有可比性),需对各变量集执行标准化处理。对于定量变量集(将用于PCA),通常通过将每个变量除以其标准差进行标准化(即Z-scores);对于分类变量集(将用于MCA),每组通过将其所有元素除以称为其第一奇异值(该值是标准差矩阵等效项)的量进行标准化。type参数中,n即用于这种对分类变量的标准化,s用于定量变量的标准化,详情参阅帮助文档。
name.group为变量集指定名称。
> summary(res.mfa)
Call:
MFA(base = wine, group = c(2, 5, 3, 10, 9, 2), type = c("n",
"s", "s", "s", "s", "s"), name.group = c("origin", "odor_before_shaking",
"visual", "odor_after_shaking", "taste", "quality"), num.group.sup = c(1,
6), graph = TRUE)
Eigenvalues
Dim.1 Dim.2 Dim.3 Dim.4 Dim.5 Dim.6 Dim.7 Dim.8 Dim.9 Dim.10
Variance 3.462 1.367 0.615 0.372 0.270 0.202 0.176 0.126 0.105 0.079
% of var. 49.378 19.494 8.778 5.309 3.857 2.887 2.506 1.796 1.502 1.124
Cumulative % of var. 49.378 68.873 77.651 82.960 86.816 89.703 92.209 94.005 95.506 96.630
Dim.11 Dim.12 Dim.13 Dim.14 Dim.15 Dim.16 Dim.17 Dim.18 Dim.19 Dim.20
Variance 0.074 0.060 0.029 0.022 0.019 0.011 0.009 0.006 0.003 0.002
% of var. 1.054 0.861 0.409 0.313 0.273 0.156 0.131 0.091 0.047 0.035
Cumulative % of var. 97.684 98.545 98.954 99.268 99.541 99.697 99.827 99.918 99.965 100.000
铁汉 18:32:50
Groups
Dim.1 ctr cos2 Dim.2 ctr cos2 Dim.3 ctr cos2
odor_before_shaking | 0.782 22.591 0.380 | 0.620 45.346 0.239 | 0.374 60.695 0.087 |
visual | 0.855 24.688 0.728 | 0.040 2.937 0.002 | 0.014 2.337 0.000 |
odor_after_shaking | 0.925 26.712 0.625 | 0.469 34.309 0.161 | 0.180 29.263 0.024 |
taste | 0.900 26.009 0.722 | 0.238 17.408 0.050 | 0.047 7.705 0.002 |
Supplementary groups
Dim.1 cos2 Dim.2 cos2 Dim.3 cos2
origin | 0.296 0.033 | 0.643 0.156 | 0.196 0.015 |
quality | 0.619 0.380 | 0.254 0.064 | 0.010 0.000 |
Individuals (the 10 first)
Dim.1 ctr cos2 Dim.2 ctr cos2 Dim.3 ctr cos2
2EL | 0.239 0.078 0.016 | -0.797 2.211 0.182 | 0.936 6.775 0.250 |
1CHA | -2.045 5.751 0.419 | -1.383 6.667 0.192 | 1.514 17.725 0.229 |
1FON | -1.220 2.048 0.367 | -0.459 0.734 0.052 | 0.062 0.030 0.001 |
1VAU | -4.381 26.404 0.874 | 0.995 3.446 0.045 | -0.033 0.009 0.000 |
1DAM | 2.696 9.996 0.754 | -0.120 0.050 0.002 | -0.690 3.683 0.049 |
2BOU | 0.869 1.038 0.219 | -0.326 0.371 0.031 | 0.391 1.183 0.044 |
1BOI | 1.553 3.318 0.617 | -0.280 0.272 0.020 | -0.414 1.324 0.044 |
3EL | 0.129 0.023 0.003 | 0.789 2.167 0.115 | 1.858 26.707 0.636 |
DOM1 | -0.066 0.006 0.002 | -0.253 0.222 0.027 | -0.459 1.629 0.090 |
1TUR | -1.202 1.987 0.310 | -0.375 0.489 0.030 | -0.716 3.964 0.110 |
Continuous variables (the 10 first)
Dim.1 ctr cos2 Dim.2 ctr cos2 Dim.3 ctr cos2
Odor.Intensity.before.shaking | 0.591 4.497 0.349 | 0.667 14.530 0.445 | -0.023 0.039 0.001 |
Aroma.quality.before.shaking | 0.835 8.989 0.698 | -0.075 0.186 0.006 | -0.354 9.092 0.125 |
Fruity.before.shaking | 0.716 6.606 0.513 | -0.151 0.741 0.023 | -0.537 20.939 0.289 |
Flower.before.shaking | 0.439 2.480 0.192 | -0.409 5.469 0.168 | 0.637 29.439 0.406 |
Spice.before.shaking | 0.038 0.019 0.001 | 0.865 24.420 0.748 | 0.128 1.187 0.016 |
Visual.intensity | 0.881 7.912 0.776 | 0.238 1.466 0.057 | 0.141 1.139 0.020 |
Nuance | 0.862 7.577 0.744 | 0.234 1.408 0.055 | 0.142 1.155 0.020 |
Surface.feeling | 0.950 9.198 0.903 | 0.049 0.063 0.002 | -0.027 0.043 0.001 |
Odor.Intensity | 0.627 2.416 0.393 | 0.576 5.155 0.331 | 0.214 1.581 0.046 |
Quality.of.odour | 0.791 3.844 0.626 | -0.410 2.612 0.168 | -0.221 1.684 0.049 |
Supplementary continuous variables
Dim.1 cos2 Dim.2 cos2 Dim.3 cos2
Overall.quality | 0.747 0.558 | -0.504 0.254 | 0.130 0.017 |
Typical | 0.766 0.586 | -0.466 0.217 | 0.039 0.001 |
Supplementary categories
Dim.1 cos2 v.test Dim.2 cos2 v.test Dim.3 cos2 v.test
Saumur | 0.533 0.483 1.343 | 0.350 0.209 1.405 | 0.235 0.094 1.404 |
Bourgueuil | -0.392 0.176 -0.596 | -0.504 0.291 -1.219 | -0.216 0.054 -0.780 |
Chinon | -0.877 0.537 -1.022 | -0.207 0.030 -0.384 | -0.322 0.072 -0.889 |
Reference | 1.437 0.823 2.442 | -0.567 0.128 -1.534 | -0.164 0.011 -0.662 |
Env1 | -0.949 0.614 -1.613 | -0.467 0.149 -1.263 | 0.455 0.141 1.834 |
Env2 | -0.794 0.554 -1.067 | 0.191 0.032 0.409 | -0.382 0.129 -1.218 |
Env4 | 0.277 0.008 0.216 | 3.141 0.971 3.899 | -0.062 0.000 -0.116 |
结果概要显示,前两轴承载了68.87%的总方差,具有较高的代表性。
接下来对于对象(葡萄酒)和变量(属性)间的关系,以及变量对排序空间的贡献等,可主要通过排序图评估。
graph = TRUE参数,可在计算过程中自动输出图形。不过我们有更便捷的可视化方法,见下文。
同样地,对于MFA主要信息的提取,除了可直接在结果中提取外,也和排序图的展示一样使用其它工具辅助完成,见下文。
#提取主要信息,例如
> res.mfa
**Results of the Multiple Factor Analysis (MFA)**
The analysis was performed on 21 individuals, described by 31 variables
*Results are available in the following objects :
name description
1 "$eig" "eigenvalues"
2 "$separate.analyses" "separate analyses for each group of variables"
3 "$group" "results for all the groups"
4 "$partial.axes" "results for the partial axes"
5 "$inertia.ratio" "inertia ratio"
6 "$ind" "results for the individuals"
7 "$quanti.var" "results for the quantitative variables"
8 "$quanti.var.sup" "results for the quantitative supplementary variables"
9 "$quali.var.sup" "results for the categorical supplementary variables"
10 "$summary.quanti" "summary for the quantitative variables"
11 "$summary.quali" "summary for the categorical variables"
12 "$global.pca" "results for the global PCA"
> res.mfa$eig #各轴特征值
eigenvalue percentage of variance cumulative percentage of variance
comp 1 3.461950436 49.37838239 49.37838
comp 2 1.366768270 19.49444613 68.87283
comp 3 0.615429078 8.77796864 77.65080
comp 4 0.372199671 5.30874662 82.95954
comp 5 0.270382485 3.85651094 86.81605
comp 6 0.202403306 2.88691245 89.70297
comp 7 0.175713405 2.50622990 92.20920
comp 8 0.125898681 1.79571410 94.00491
comp 9 0.105275528 1.50156260 95.50647
comp 10 0.078791202 1.12381220 96.63029
comp 11 0.073892411 1.05393990 97.68423
comp 12 0.060338440 0.86061733 98.54484
comp 13 0.028705009 0.40942438 98.95427
comp 14 0.021962548 0.31325552 99.26752
comp 15 0.019163991 0.27333923 99.54086
comp 16 0.010940292 0.15604323 99.69691
comp 17 0.009156013 0.13059375 99.82750
comp 18 0.006370966 0.09087016 99.91837
comp 19 0.003303413 0.04711714 99.96549
comp 20 0.002419756 0.03451339 100.00000
2.3 R包factoextra的可视化方法
接下来展示使用factoextra包来帮助解释和可视化MFA结果。
尽管下述方法并非全部来自factoextra包。
2.3.1 特征值/方差
首先查看MFA的特征值概况。
#借助 factoextra 包更好地提取数据和可视化
library(factoextra)
#提取每个维度(轴)的特征值/承载的方差
> eig.val <- get_eigenvalue(res.mfa)
> head(eig.val)
eigenvalue variance.percent cumulative.variance.percent
Dim.1 3.4619504 49.378382 49.37838
Dim.2 1.3667683 19.494446 68.87283
Dim.3 0.6154291 8.777969 77.65080
Dim.4 0.3721997 5.308747 82.95954
Dim.5 0.2703825 3.856511 86.81605
Dim.6 0.2024033 2.886912 89.70297
#可视化各轴特征值/方差
fviz_screeplot(res.mfa)
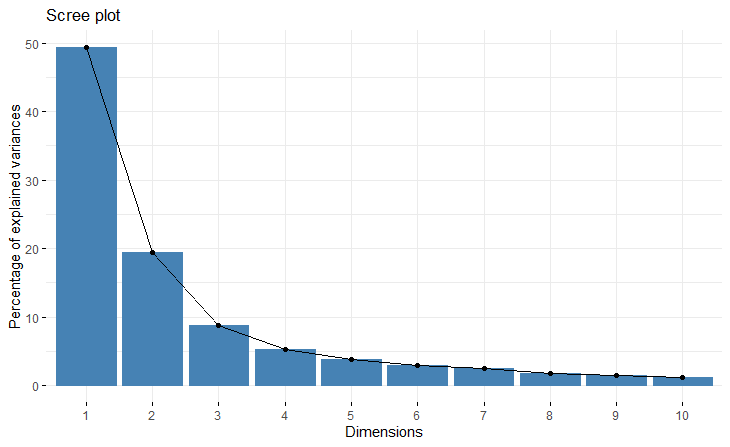
eigenvalue为各MFA轴的特征值,即各轴承载的方差;variance.percent为各轴特征值与所有轴特征值总和的比例,即各轴的方差贡献率;cumulative.variance.percent为累积贡献率。
2.3.2 变量集整体间的关系及其对排序空间的贡献
MFA结果中,可首先观测各变量集整体之间的关系。
> #变量集结果,用于反映变量集整体对 MFA 排序空间的贡献
> group <- get_mfa_var(res.mfa, 'group')
> group
Multiple Factor Analysis results for variable groups
===================================================
Name Description
1 "$coord" "Coordinates"
2 "$cos2" "Cos2, quality of representation"
3 "$contrib" "Contributions"
4 "$correlation" "Correlation between groups and principal dimensions"
查看细节部分,例如
> #各变量集作为整体,计算了它们与各 MFA 轴的相关性
> head(group$correlation)
Dim.1 Dim.2 Dim.3 Dim.4 Dim.5
odor_before_shaking 0.8880205 0.9563908 0.8866671 0.4819997 0.4191427
visual 0.9260559 0.2211940 0.1580575 0.2208213 0.1730153
odor_after_shaking 0.9687094 0.8935706 0.8957362 0.5658340 0.6589160
taste 0.9500510 0.8684367 0.2989476 0.2540307 0.5166468
排序坐标也可用于反映各变量集与各 MFA 轴的相关程度,前两轴为例展示
> head(group$coord)
Dim.1 Dim.2 Dim.3 Dim.4 Dim.5
odor_before_shaking 0.7820738 0.61977283 0.37353451 0.17260092 0.08553276
visual 0.8546846 0.04014481 0.01438360 0.04550736 0.02966750
odor_after_shaking 0.9247734 0.46892047 0.18009116 0.10139051 0.11589439
taste 0.9004187 0.23793016 0.04741982 0.05270088 0.03928784
> fviz_mfa_var(res.mfa, 'group', axes = 1:2)

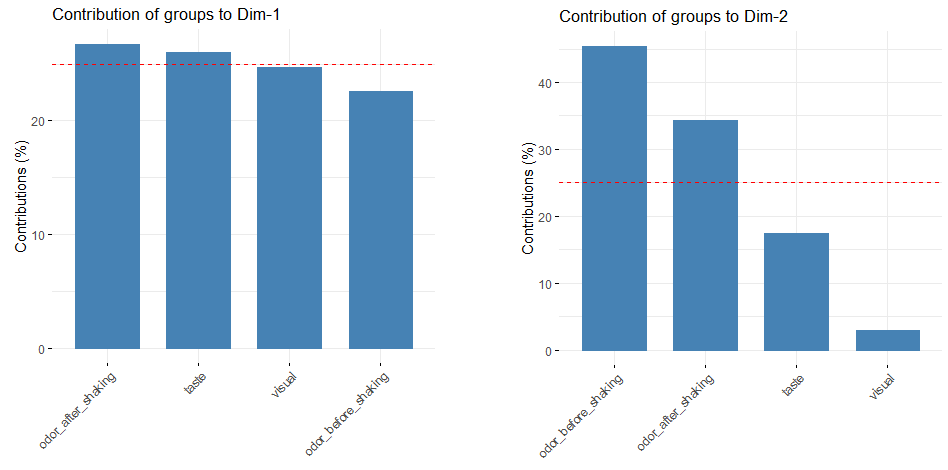
对应MFA命令行参数,摇动前后的气味、视觉和味觉作为active groups,图中红色显示;来源和质量作为supplementary groups,图中绿色显示。
根据变量集在排序图中的位置,可以评估各变量集与MFA轴之间的相关性及相对贡献。第一轴上4个active groups的坐标几乎相同,意味着它们对第一维的贡献相似;对于第二轴,摇动前后的气味具有相对较大的坐标,表明它们对第二维的贡献最大。
直接显示了各变量集对各排序轴的贡献度
> head(group$contrib)
Dim.1 Dim.2 Dim.3 Dim.4 Dim.5
odor_before_shaking 22.59055 45.345861 60.694972 46.37321 31.63399
visual 24.68795 2.937207 2.337166 12.22660 10.97242
odor_after_shaking 26.71250 34.308703 29.262699 27.24089 42.86313
taste 26.00900 17.408230 7.705163 14.15930 14.53047
>
> #可视化各变量集对前两轴的贡献度
> fviz_contrib(res.mfa, 'group', axes = 1)
> fviz_contrib(res.mfa, 'group', axes = 2)
变量集之间的相关性通过 RV 系数衡量
#变量集之间的相关性矩阵
> res.mfa$group$RV
origin odor_before_shaking visual odor_after_shaking taste quality
origin 1.0000000 0.4110211 0.1698138 0.3731336 0.2933589 0.2047620
odor_before_shaking 0.4110211 1.0000000 0.4359249 0.7067631 0.5092398 0.3697345
visual 0.1698138 0.4359249 1.0000000 0.6013347 0.7493245 0.4236970
odor_after_shaking 0.3731336 0.7067631 0.6013347 1.0000000 0.7581927 0.6992595
taste 0.2933589 0.5092398 0.7493245 0.7581927 1.0000000 0.7608006
quality 0.2047620 0.3697345 0.4236970 0.6992595 0.7608006 1.0000000
MFA 0.3771260 0.8050704 0.7998813 0.9100320 0.8761798 0.6598390
MFA
origin 0.3771260
odor_before_shaking 0.8050704
visual 0.7998813
odor_after_shaking 0.9100320
taste 0.8761798
quality 0.6598390
MFA 1.0000000
#相关图
library(corrplot)
corrplot(res.mfa$group$RV, method = 'number', number.cex = 0.8, diag = FALSE, tl.cex = 0.8)
corrplot(res.mfa$group$RV, add = TRUE, type = 'upper', method = 'pie', diag = FALSE, tl.pos = 'n', cl.pos = 'n')
#这种相关性可通过置换检验确定重要性 #例如摇动前的气味(原始数据集的 3-7 行)和摇动后的气味(原始数据集的 11-20 行)之间的相关性的置换检验 #记得执行 Z-scores 标准化变量集
> coeffRV(scale(wine[3:7]), scale(wine[11:20]))$p.value
[1] 2.221008e-07

2.4 各变量间的关系及其对排序空间的贡献
上述查看了变量集整体间的关系,接下来可以查看各变量间的关系。
各具体变量对排序空间的贡献,数据结构同上述“变量集整体”
> quanti.var <- get_mfa_var(res.mfa, 'quanti.var')
> quanti.var
例如同样以排序图可视化各变量与 MFA 轴的关系,前两轴为例展示
> head(quanti.var$coord) #排序坐标
> fviz_mfa_var(res.mfa, 'quanti.var', axes = 1:2, palette = 'jco', col.var.sup = 'violet', repel = TRUE)

图中颜色代表了变量所属的变量集,定量变量展示为向量。相关圈(correlation circle)代表所有定量变量范数标准化向量。
两个变量之间的夹角为锐角,则二者存在正相关;钝角为负相关;趋于正交则相关性很低。变量与哪个轴的夹角越小,表明其越作用于哪个轴;变量在某轴的投影长度越长,则表明其对该轴的贡献越大。
#直接显示了各变量对各排序轴的贡献度
head(quanti.var$contrib)
#展示 top20 变量对前两轴的贡献度
fviz_contrib(res.mfa, choice = 'quanti.var', axes = 1, top = 20, palette = 'jco')
fviz_contrib(res.mfa, choice = 'quanti.var', axes = 2, top = 20, palette = 'jco')
12.png
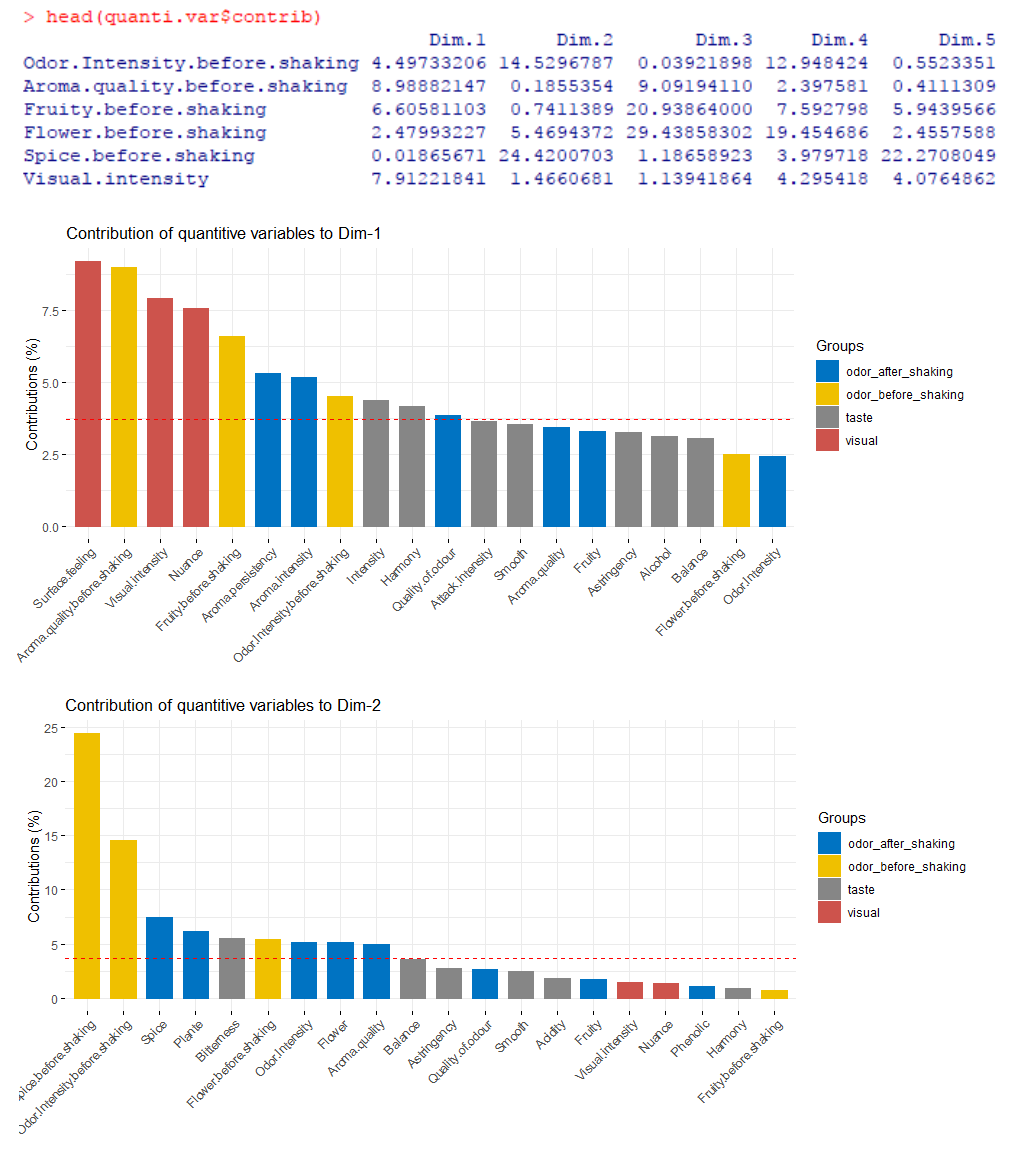
红色虚线表示平均贡献值,便于观测哪些变量大于平均贡献。
#可选使用渐变色,在排序图中按变量贡献对 active groups 变量上色
#这里方便观测数据,将所有变量以点展示
fviz_mfa_var(res.mfa, 'quanti.var', axes = 1:2, col.var = 'contrib', geom = c('point', 'text'),
gradient.cols = c('#00AFBB', '#E7B800', '#FC4E07'), col.var.sup = 'violet', repel = TRUE)
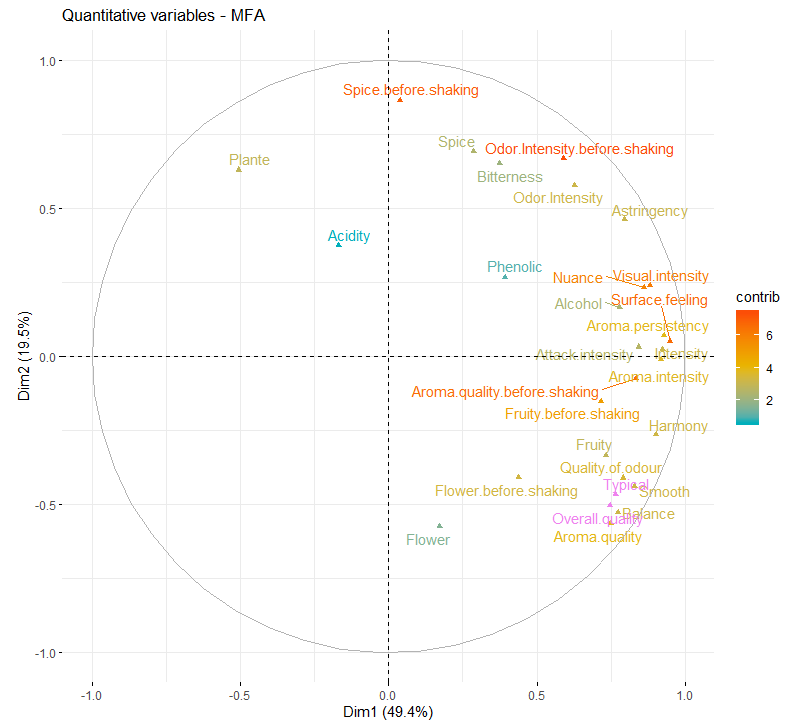
#变量太多了,评估最具典型的一些变量
select_val <- dimdesc(res.mfa, axes = 1:2, proba = 0.001)
summary(select_val)
#保留最具典型的一些变量的排序图
varsig <- res.mfa$quanti.var$cor
varsig <- subset(varsig, rownames(varsig) %in% unique(c(rownames(select_val$Dim.1$quanti), rownames(select_val$Dim.2$quanti))))
plot(varsig[ ,1:2], asp = 1, type = 'n', xlim = c(-1, 1), ylim = c(-1, 1))
abline(h = 0, lty = 3)
abline(v = 0, lty = 3)
symbols(0, 0, circles = 1, inches = FALSE, add = TRUE)
arrows(0, 0, varsig[,1], varsig[,2], length = 0.08, angle = 20)
for (v in 1:nrow(varsig)) {
if (abs(varsig[v,1]) > abs(varsig[v,2])) {
if (varsig[v,1] >= 0) pos <- 4 else pos <- 2
} else {
if (varsig[v,2] >= 0) pos <- 3 else pos <- 1
}
text(varsig[v,1], varsig[v,2], labels=rownames(varsig)[v], pos = pos)
}
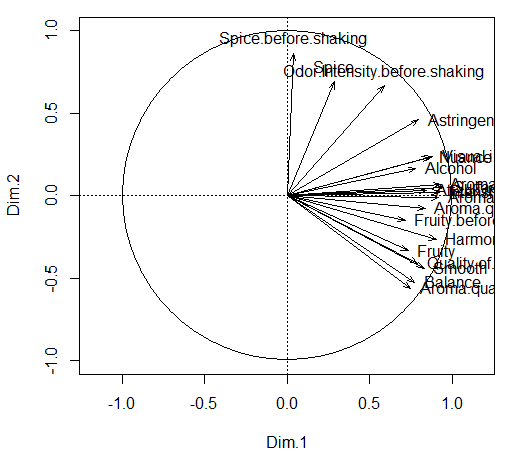
2.5 对象间以及对象和变量间的关系
观测对象(葡萄酒)在排序空间中的位置及相互关系,以及对象和变量的关系。
#查看排序对象,数据结构类上上述
ind <- get_mfa_ind(res.mfa)
ind
#例如查看对象排序坐标
head(ind$coord)
#展示对象在前两轴中的排序图
#如果不想在图中展示分类变量,可使用参数 invisible = 'quali.var'
fviz_mfa_ind(res.mfa, axes = 1:2, palette = c('#00AFBB', '#E7B800', '#FC4E07'),
habillage = 'Label', addEllipses = TRUE, ellipse.type = 'confidence', repel = TRUE)
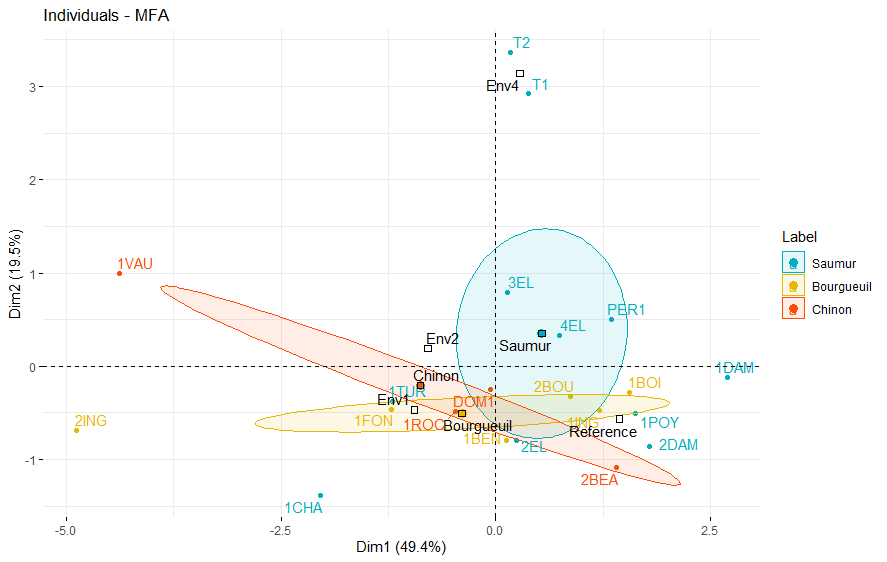
分类变量将默认和对象一起展示在该图中。分类变量显示为黑色,Env1、Env2、Env3代表变量集中的土壤类别,Saumur、Bourgueuil和Chinon代表酒的产地。在该图中,对象(葡萄酒)按其产地着色显示。
若两种葡萄酒的品质较为相似,则它们在排序图中的位置相互接近。
可结合上文中变量集或变量对各排序轴的贡献,评价葡萄酒特征。例如,葡萄酒T1和T2在第二轴上对应较高的数值,而葡萄酒摇动前后的气味两组变量集对第二轴具有较高的贡献,那么可知T1和T2应当属于更为“芳香浓郁”类型。
以及评估分类变量和对象以及定量变量间的关系。例如,分类变量Env4在第二轴上具有较高坐标值,靠近葡萄酒T1和T2,暗示改土壤环境中的葡萄更具香甜品质。
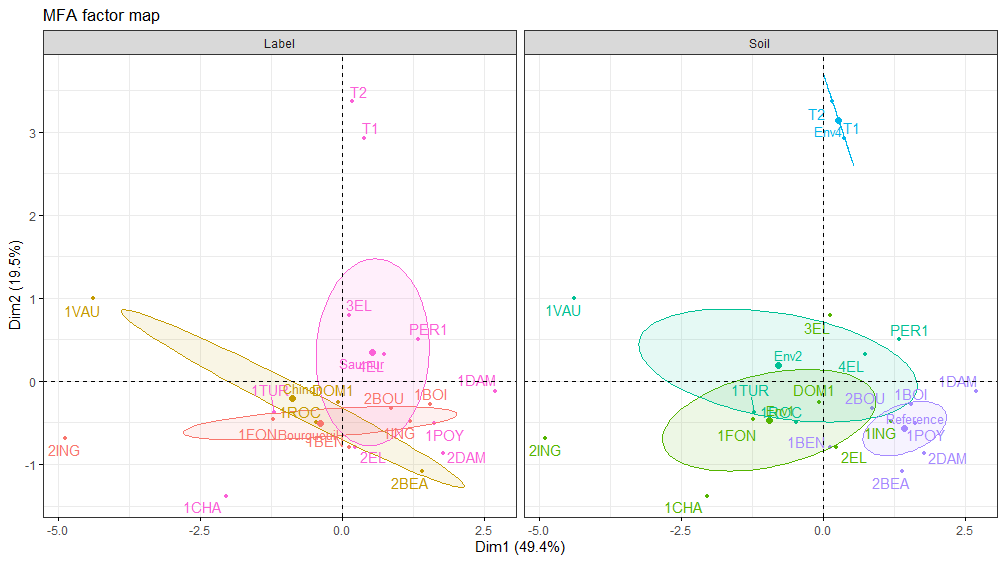
#其它可选可视化方案,例如分别按两组分类变量特征,在图中对代表葡萄酒的点标注颜色
fviz_ellipses(res.mfa, axes = 1:2, c('Label', 'Soil'), repel = TRUE)
在排序图中展示MFA的全局PCA与各组变量集PCA中对象得分的关系。
#MFA 图中对象位置与各组变量集 PCA 中的对象位置,前两轴为例展示
fviz_mfa_ind(res.mfa, axes = 1:2, partial = 'all')
#可选择部分对象展示
fviz_mfa_ind(res.mfa, axes = 1:2, partial = c('1DAM', '1VAU', '2ING'))
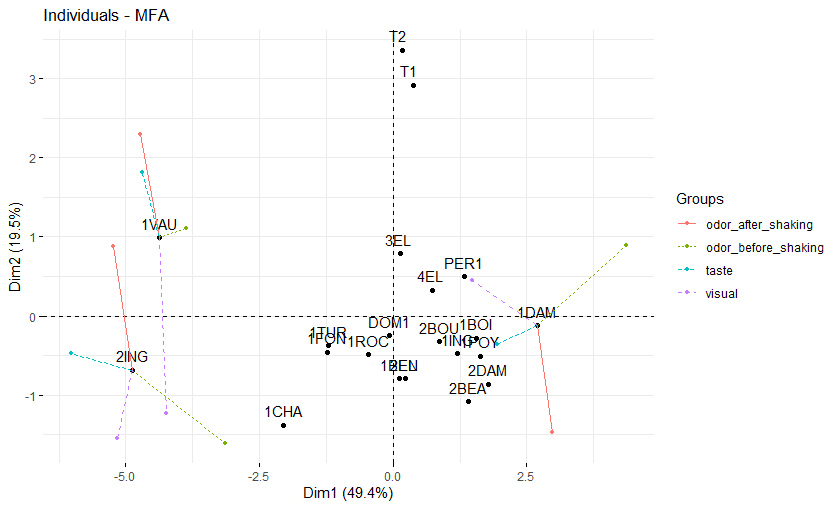
图中黑点代表MFA排序空间内的对象坐标;与对象点相连的不同颜色的线则代表了各变量集各自的PCA中对象的坐标,它们的形心即为MFA中对象坐标。
偏轴图
偏轴(partial axes)代表了所有变量集各自PCA(或MCA)所得特征向量(排序轴)投影到MFA排序空间中的情况,图中圆圈的半径(等于1)代表标准化偏轴最大的长度。
#偏轴图,前两轴为例展示
fviz_mfa_axes(res.mfa, axes = 1:2)
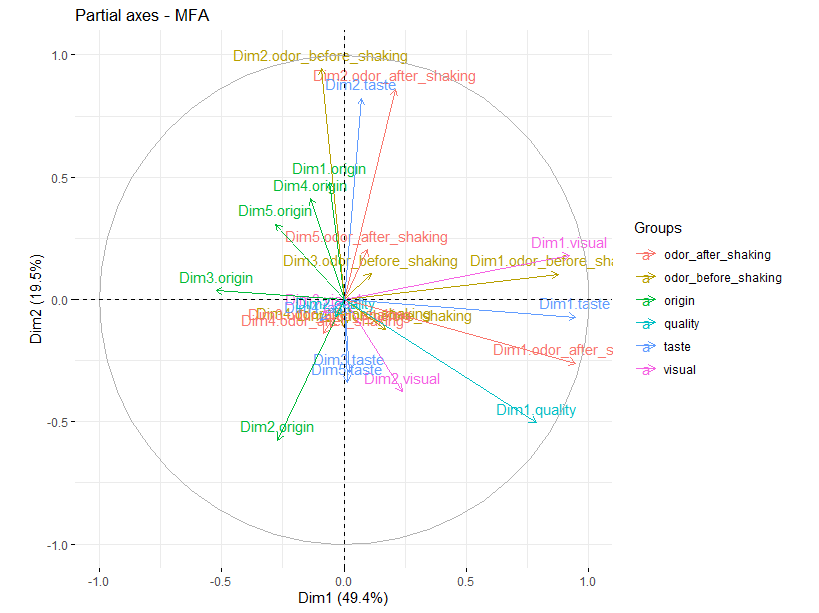
通过该图可评估哪些变量集的特征对MFA排序空间具有更高的贡献,这些变量集的前两轴与MFA的前两轴是否高度相关,MFA的各维度主要受哪种类型的属性集支配等。
三、我的案例
## 安装相关的包
install.packages('readxl')
install.packages('ggplot2')
install.packages('FactoMineR')
install.packages('corrplot')
setwd("D:/project/antibody/result")
library(readxl)
library(ggplot2)
library(FactoMineR)
library(corrplot)
df = read_excel('merged_info.xlsx','analysis');
df = as.data.frame(df);
df = df[,c('Ab', 'SingalP', 'chain','Yield',"CAI(%)")];
res.mfa <- MFA(df, group = c(1,1,1,1,1), type = c('n','n','n','s','s'), name.group = c('Ab', 'SingalP', 'chain','Yield',"CAI(%)"),graph = TRUE);
corrplot(res.mfa$group$RV, method = 'number', number.cex = 0.8, diag = FALSE, tl.cex = 0.8)
corrplot(res.mfa$group$RV, add = TRUE, type = 'upper', method = 'pie', diag = FALSE, tl.pos = 'n', cl.pos = 'n')
四、报错
> test.mfa <- MFA(test, group = c(1,1,1,1,1,1,1,1),type = c('n','n','n','n','n','s','s','s'),num.group.sup = c(1, 3),graph = TRUE)
Error in U[, num, drop = FALSE] : 量度数目不对
居然是有一列的数据完全一样,都是为n
参考资料
- http://wap.sciencenet.cn/blog-3406804-1211632.html
- Abdi, Hervé, Williams L J, Valentin D. Multiple factor analysis: principal component analysis for multitable and multiblock data sets. Wiley Interdisciplinary Reviews: Computational Statistics, 2013, 5(2):149-179.
- DanielBorcard, FranoisGillet, PierreLegendre, et al. 数量生态学:R语言的应用(赖江山 译). 高等教育出版社, 2014.
- Escofier, B. and Pagès, J. Multiple factor analysis. Computational Statistics & Data Analysis, 1990, 18, 121–140.
- MFA - Multiple Factor Analysis in R: Essentials
参考资料
