【7.1.3】蛋白质可以被设成没有功能么
蛋白质具有各种大小,形状和折叠,并具有多种功能。 关于蛋白质如何从肽序列折叠以及如何折叠以产生特定结构的研究已经持续了数十年(1),并激发了从头设计蛋白质的努力,即从第一原理合理设计结构化的微型蛋白质折叠。 最终,目标不仅在于设计特定的折叠,还在于创造出能够执行功能的蛋白质。理想地,功能超出了自然界中发现的所有功能。 当所需功能涉及小分子的结合时(在许多应用中就是这种情况),此要求增加了复杂性和挑战性的附加水平。 在本期杂志的第1227页上,Polizzi和DeGrado(2)开发了一种搜索算法,即结合位点聚合motifs(COMBS,Convergent Motifs for Binding Sites),通过该算法可以真正地重新设计配体选择性结合蛋白,从而提供了一个急需的工具 推进功能蛋白设计
首次报道了真正的人工蛋白质折叠的从头设计,它是TOP7的一种93个氨基酸混合 α/β折叠(3)。 从那时起,蛋白质结构的从头设计通过增强的理解,蛋白质数据库中经过实验验证的结构的扩展,更强大的计算能力以及对合成基因的可承受的获取而获得了可观的进步,从而可以进行更大的实验验证。 人造肽还被设计成可组装成以前未知的结构,包括a-螺旋barrrels,其特征是可通过其核心(4),大型球形笼(5)和多面体形状(6)进入通道。 最近,蛋白质折叠和在线计算机游戏设计的发展甚至使公民科学家能够设计以前未开发的蛋白质折叠(7)。
蛋白质功能可以通过诸如胶原蛋白之类的实际结构来实现,但更常见的是通过与生成传感器,受体或催化剂的复杂小分子配体的结合来实现。必须设计一个具有大小和形状的腔体,以用作目标配体的互补结构,使目标配体对齐以实现良好的相互作用,但又不会干扰正确的折叠。设计结构然后引入功能的常见策略通常是以适当的结构折叠或稳定性为代价的,并且通常是不成功的。更具吸引力的方法是同时设计结构和功能。先前报道的策略倾向于将目标配体相对于氨基酸侧链的相互作用原子进行定位(8、9)。这种方法可以生成大量结构以进行计算评估,并且还可以导致坐标和配体旋转异构体的组合,而这些组合在实验中并未实际观察到。通常,迄今为止的方法最初并没有达到牢固的结合,因此通常需要随后的几轮实验验证和重新设计。
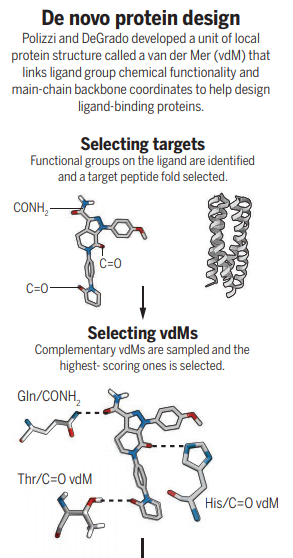
Polizzi和DeGrado设计了一种人工蛋白质折叠,该蛋白质折叠与天然蛋白质没有序列同源性,并且还具有对复杂的小分子有选择性的结合位点。在这种情况下,这种药物是血液稀释性药物阿哌沙班(apixaban)(见图)。 他们开发了一种新的蛋白质结构单元,称为范德(vd, van der Mer),可将配体化学基团功能(例如羰基,羧酸根,羧酰胺和胺)直接映射到肽残基的骨架坐标上。至关重要的是,vds是通过与侧链,主链或两者的紧密接触生成的。配体的化学基团位置与骨架坐标相关,与侧链无关,因此vds直接与蛋白质折叠连接。 vds是从存放在蛋白质数据库中的实验确定的结构中提取的,然后使用整个vds在蛋白质数据库中的发生率对它们进行评分。令人惊讶的是,只有很少数量的vds非常流行(prevalent),因此采用这种方法在计算上很有吸引力
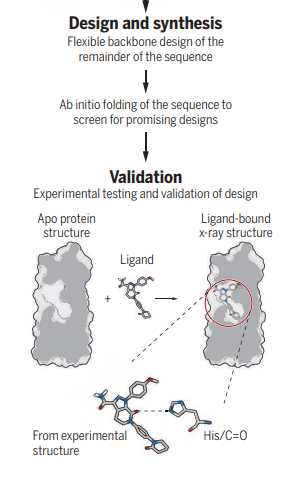
结合位点被工程化为设计的四螺旋束折叠,这些折叠在结构上与Xa因子无关,而Apixaban是抑制剂(10),并且通常不与小分子结合。 作者搜索了可以叠加到蛋白质骨架模板上并呈现出可以与目标配体重叠的化学基团的vds组合(有利于分数更高的组合)。 灵活的主链序列设计用于构建序列的其余部分。 最后,通过从头开始折叠序列来验证低能量,包装良好的设计,以建立在没有结合的靶配体的情况下保留未折叠和预先组织的结合位点的设计。 不需要后续的下游重新设计来增强结构稳定性,功能或配体结合活性。 这一成就代表了从头开始功能性蛋白设计的重要一步。
为了充分发挥从头蛋白质设计的潜力,需要同时进行功能设计。 未折叠的蛋白质,或具有预期或偶然突变的蛋白质,可能没有功能,而生物学上成功的和预期的折叠提供功能。 鉴于生物学功能库是通过选择的进化压力进化而来的,因此设计的具有相同功能的蛋白质不可能提供实质性的优势,因此,超出生物学范围的新功能是目标。 最近的发展,包括Polizzi和DeGrado同时设计蛋白质结构和配体结合位点,将为传感,光捕获和存储,诊断, 治疗和催化等。 蛋白质设计界现在准备设计功能性蛋白质,这些蛋白质可以开始解决当今社会面临的一些最紧迫的挑战,包括能源,保健和可持续性方面的挑战。 如果要记住具有新的创造功能的研究人员要实现蛋白质设计的全部潜能,则需要使非专家用户能够使用新的蛋白质设计算法,类似于蛋白质设计在线计算机游戏(7)。
参考资料
