【6.1.2】用于抗体治疗发现和开发的噬菌体展示库
摘要:噬菌体展示技术(Phage display technology)在发现和优化适用于多种应用的抗体(尤其是基于抗体的药物)的显着进步中发挥了关键作用。这项技术最初是由乔治·史密斯(George Smith)在1980年代中期开发的,并于1990年代初由约翰·麦卡菲蒂(John McCafferty)和格雷戈里·温特(Gregory Winter)应用于抗体工程。在这里,我们比较了过去十年发布的九种噬菌体展示抗体库,它们代表了使用噬菌体展示发现和开发治疗性抗体的最新技术。
- 我们首先讨论文库的质量以及用作构建文库的底物的各种抗体库:即天然(naïve),合成和半合成的。
- 其次,我们根据每次淘选的阳性克隆数,命中率,亲和力和所选抗体的可开发性来回顾文库的性能。
- 最后,我们重点介绍了与噬菌体展示平台和相关展示技术有关的当前机遇和挑战。
一、前言
噬菌体展示方法彻底改变了蛋白质工程领域,对抗体工程产生了重大影响。噬菌体展示方法的显着发展中的第一个突破,已成为1985年由George Smith发表的最成功的工程化基于抗体的药物技术平台之一[1]。史密斯(Smith)表明,外源DNA片段可以插入编码丝状噬菌体外壳蛋白III(pIII)的基因中,表达为肽-噬菌体融合体,并通过与针对该肽的特异性抗体亲和而比野生型噬菌体富集1000倍以上。三年后,Parmley和Smith [2]描述了如何通过使用对肽特异的生物素化抗体来富集一百万个野生型病毒体中的肽-噬菌体融合物,其比例可低至一个肽-噬菌体融合物,这种方法被称为生物淘选(biopanning)。随后,三组[3,4,5]表明,数百万个随机短肽的文库产生了可以紧密结合用作选择子的多种抗体的特异性肽, 因此,无需事先了解其特异性即可为鉴定任何给定抗体的特定肽奠定基础。
在1990年代初,John McCafferty和Gregory Winter [6]将噬菌体展示方法的应用扩展到了抗体工程领域。将编码鼠类抗鸡抗鸡卵白溶菌酶(HEL)抗体D1.3 [7]可变区(V)的基因插入pIII,并在噬菌体表面展示为融合蛋白。然后通过HEL亲和色谱分离D1.3噬菌体融合物,类似于早期工作中所述的特定肽[3,4,5]。进一步的研究表明,只要pIII融合物对大肠杆菌无毒,噬菌体展示方法就无法确定将哪种类型的蛋白质[8]或抗体[9,10]基因插入到噬菌体外壳蛋白中。噬菌体表面并选择特异性抗体或配体(图1)。相对于杂交瘤技术,噬菌体展示的这种破坏性优势在将近二十年前就已发展[11],对治疗性抗体的发展产生了重大影响。虽然杂交瘤技术仅限于获得单克隆啮齿动物抗体,但噬菌体展示可分离人抗体,因此在用于人类治疗时可将与非人抗体相关的免疫反应降至最低[12]。
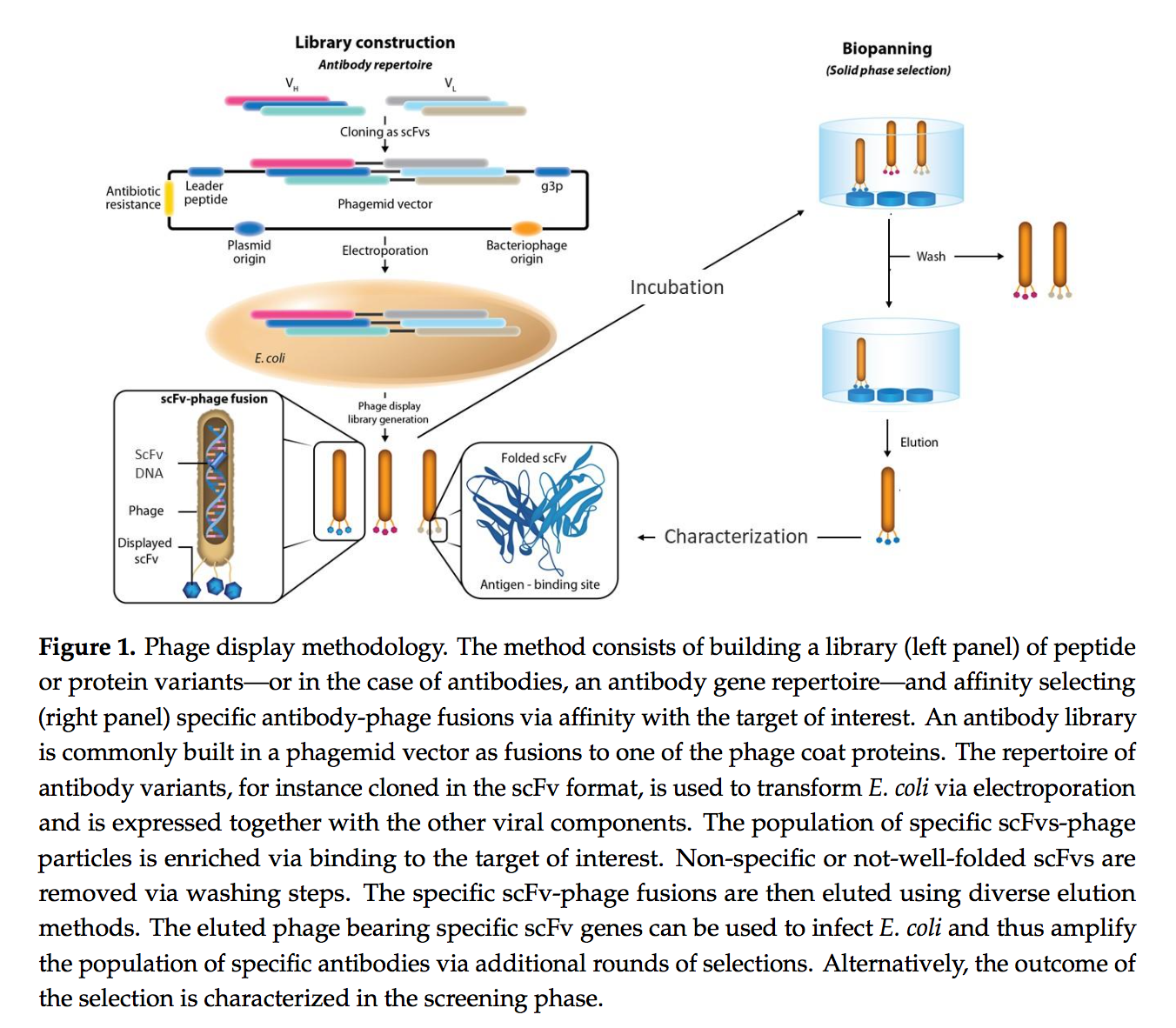
由于噬菌体展示选择过程是在体外进行的,因此这种新技术平台还可以在多种环境中分离治疗性抗体。 例如,通过使用噬菌体展示与嵌合抗体西妥昔单抗竞争获得了necitumumab一种用于治疗癌症的人抗体)[13]。 另一个例子是阿达木单抗(Adalimumab),它是第一个进入市场的全人类抗体,也是全球最畅销的药物。 通过小鼠抗体的指导选择,通过噬菌体展示发现并优化了阿达木单抗[14]。 实际上,直到2017年,通过噬菌体展示发现和/或工程改造的六种完全人类治疗性抗体[15]已获得美国食品药品管理局(FDA)和/或欧洲药品管理局(EMA)的批准,数百种已经通过 进行临床试验[16]。
最初,噬菌体展示抗体发现活动的主要目标是选择特异性和高亲和力的抗体[17]。 然而,在最近的十年中,已经了解到,除了特异性和亲和力以外,其他特性也说明了治疗性抗体在临床试验中的成功。 此类特性统称为可开发性(developability)[18,19],包括(但不限于)与人类靶标和相关毒素物种和动物模型的直系同源物的交叉反应性,溶解度,生产细胞中的表达量以及热和长效 长期稳定性。 由于这些性质是在抗体的氨基酸序列中编码的,因此迫切需要生成噬菌体展示抗体库,该库不仅可以选择高特异性和高亲和力的抗体,而且还可以选择更可开发的抗体。
结果,在约翰·麦卡菲蒂(John McCafferty)和格雷戈里·温特(Gregory Winter)在1990年进行开创性工作后,已经建立了许多噬菌体展示抗体文库[6]。这些文库,它们的构建过程,具有不同靶点的验证以及所选抗体在诊断和/或治疗方面的应用已在文献中进行了广泛的综述[20,21,22,23,24,25]。在这里,鉴于有关噬菌体展示的大量信息以及空间限制,我们回顾了过去十年发布的九种噬菌体展示scFv和Fab库(表1)。这九个文库是最新一代的噬菌体展示抗体文库,代表了人类治疗性抗体发现的最新平台。我们
- 首先讨论抗体库的大小,质量和多样性方面的文库功能。
- 其次,我们审查并比较选择过程的结果,即阳性克隆的数量,命中率和所选抗体的亲和力。
- 第三,我们根据所选抗体的可开发性来讨论文库的性能。
- 最后,我们重点介绍了与噬菌体展示平台和其他展示技术相关的当前机遇和挑战。

二、 噬菌体展示抗体库的大小
抗体文库的大小与选择所需抗体的可能性之间的相关性有些直观,即,抗体库越大,分离出更多多样和亲和力更高的抗体的机会就越高,因此,可能性就越高。选择所需分子的过程, Alan Perelson [34,35]将这个概念形式化为 $ P = e^{-Np}$,其中P是给定抗体无法识别随机形状的表位的概率,N是抗体库的大小,p是概率所述抗体以一定亲和力接触所述表位。如果p = 5 µM(5×10^-6 M),这是一个很弱的解离常数,但可测量且与非特异性结合不同,则N = 10^6抗体文库将产生 $ p = 6.8×10^{-3(≈2.7 −(1,000,000×0.000005))} $。这意味着在一百万个抗体变异体的文库中,几乎所有可能的随机表位都将被亲和力约为5 µM的抗体识别。为了达到相同的(低)p值,但解离常数为5 nM(5×10-9 M),N应该是10^9个抗体变异体。因此,文库越大,抗体以更高的亲和力特异性结合随机表位的机会就越高。
N可以达到的最大值或所有可能的独特抗体变体的范围实际上是无限的。抗原结合位点由六个互补决定区(CDR)组成-VL中的三个:LCDR1,LCDR2和LCDR3,VH中的三个:HCDR1,HCDR2和HCDR3,它们与称为框架区的相对保守的区域交替( V域中的FR)。考虑到CDR的Kabat定义[36]和人类中最常见的CDR长度[37]:LCDR1 = 11,LCDR2 = 6,LCDR3 = 8,HCDR1 = 4,HCDR2 = 19和HCDR3 = 12,CDR处于打开状态平均长度为十个氨基酸。如果六个符合抗原结合位点的CDR的每个位置都被20个天然氨基酸所多样化,则相应的理论库将包含20^(6×10)= 20^60或1.2×10^78个独特的抗体变体。显然,只能在噬菌体展示抗体库中取样这10^78种独特抗体变体中的很小一部分。
另一方面,实际因素将噬菌体展示抗体库的最大大小限制为10^10–10^11抗体变体。这些因素是:
- 大肠杆菌的转化效率,每微克DNA介于10^10和10^11菌落转化单位(cfu,colony transforming units)之间(例如,参见ThermoFisher目录中的电感受态细胞);
- 在进行多次电穿孔以增加结扎后cfu数量的情况下,扩展文库所需的体积。
表1中列出的所有文库平均达到10^11 cfu,大小从1.5×10^10 cfu(人类抗体基因文库,HAL9 / 10)到3.6×10^11 cfu(XscFv2)不等。 因此,这里综述的所有文库都具有(或接近于)最大可能的N。与Perelson原理一致,这些文库在淘选了多种靶标后产生了单个nM或亚nM抗体,如本综述后面所述 。
三、噬菌体展示抗体库的有效大小
理想地,文库的大小应等于其有效大小,这意味着构成该文库的所有10^11抗体变体都应作为功能性抗体分子展示在噬菌体表面上。尽管如此,基因合成的质量,无论是对纯天然文库使用RT-PCR(逆转录聚合酶链反应)[9],还是对合成和半合成文库使用化学方法[29,38],都会影响文库的有效大小。具有导致截短序列的终止密码子的核苷酸序列不产生与病毒粒子融合的功能性抗体片段。一个或两个核苷酸的插入或缺失会改变基因序列的阅读框,从而产生截短的序列,其中可能会破坏折叠的氨基酸序列或与溶剂暴露的疏水性氨基酸的克隆会导致聚集。此外,由于在大肠杆菌中密码子使用欠佳和/或在细胞区室中的转运效率低下,一些符合读框的抗体基因表达低下[39]。这些变体相对于其他变体以低比例显示,或者根本不显示在噬菌体表面上,这进一步侵蚀了文库的有效大小。此外,基于寡核苷酸或简并的NNK密码子的混合物的文库合成方法,可能会由于遗传密码的冗余和/或在目标位置的多样化所编码的不需要的氨基酸而导致氨基酸水平的多样性降低。
表2总结了表1中列出的开放阅读框(ORF)的百分比和抗体表达或展示的百分比。应注意,一些作者没有报告ORF和/或展示的百分比,而其他作者进行了测量这些参数以多种方式。例如,Xoma [26]根据信号序列开始到XFab1的VL和VH两者之间以及信号序列开始到XscFv2 VL结束之间的完整序列数,计算了ORF的百分比。 基于在Fab(scFv)的周质表达的ELISA(酶联免疫吸附测定)中具有OD(光学密度)的背景的三倍以上的克隆数,计算表达或表达百分比。 Kim等[28]报道那些没有终止密码子的序列作为功能性ORF;他们没有报告表达式,也没有显示在选择之前。 pIX V3.0的作者[29]估计了无终止密码子的ORF序列的百分比以及ELISA中Fab展示的功能。对于HuCAL(人类组合抗体库)PLATINUM [30]和Ylanthia [31],MorphoSys通过将实际序列与设计进行比较来分析序列正确性。 Weber等(PHILODiamond,噬菌体展示库克隆[32])使用PCR和斑点印迹分析表达的Fab,研究了表达抗体的克隆的频率。我们(ALTHEA Gold Libraries™[33])将ORF报告为符合读框和不间断的密码子序列,并在ELISA中显示为A蛋白结合。
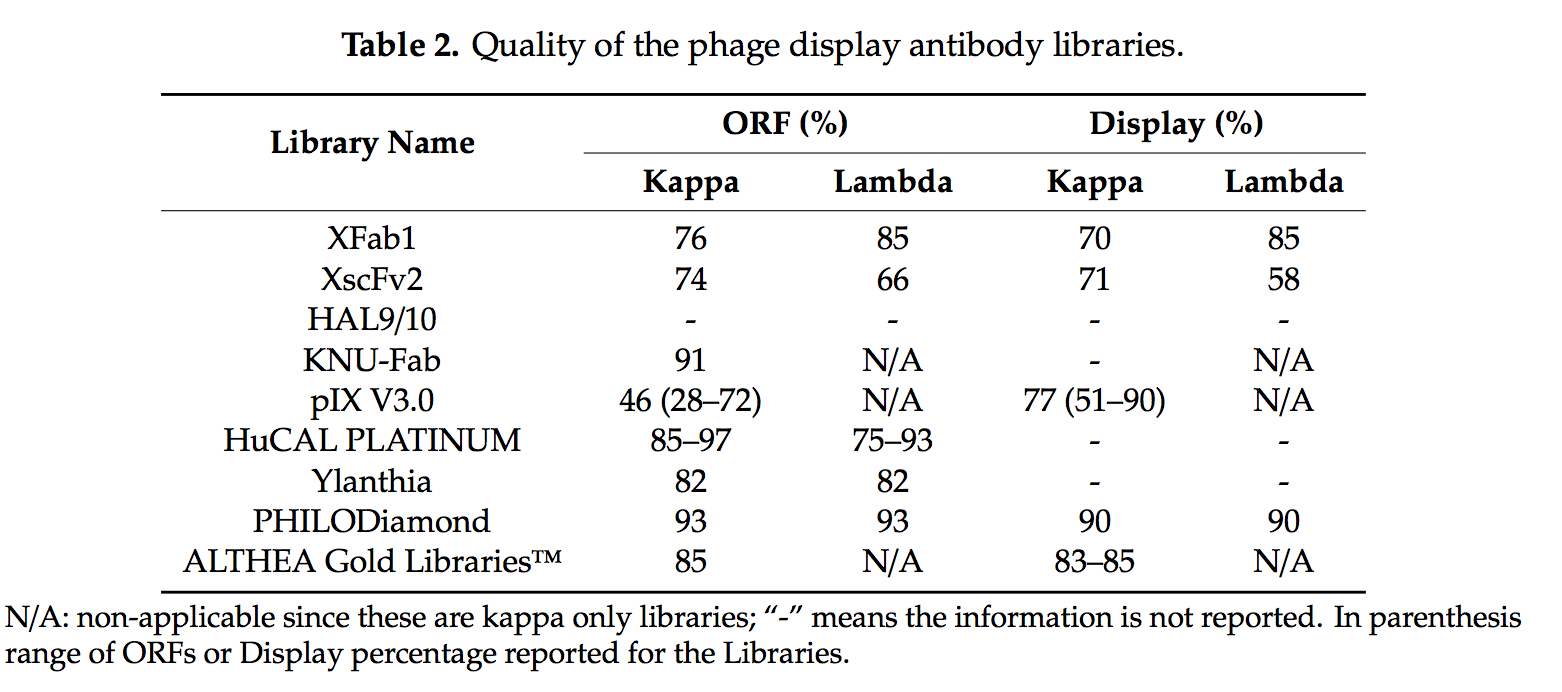
尽管衡量库功能的方法多种多样,但仍可以突出一些有意义的趋势。 除pIX V3.0(可以被认为是一个异常值)外,ORF的百分比范围从Lambda XscFv2中的66%到PHILODiamond中的93%。 显示范围从58%(也在XscFv2 lambda中)到90%(PHILODiamond)。 如预期的那样,显示略低于ORF的百分比。 考虑到显示的百分比为库的有效大小,表1的库的有效大小为60-90%。 因此,有效大小比文库,即10^10抗体变体的绝对大小低一个数量级。
四、抗体库的类型
虽然文库的大小可以用一个数字(N)定义,有效大小可以通过抗体展示的百分比来评估,但文库的多样性在某种程度上难以捉摸。有时,使用抗体库的大小作为代理来表征其多样性。但是,假设抗体库是抗原结合位点形状和氨基酸侧链的随机集合。此外,并非所有抗体库都是相同的。
例如,如果要通过用20个氨基酸使LCDR1的所有11个位置多样化来生成抗体变体文库,则最终库将是20^11或2×10^14抗体变体。基于其大小,这似乎是一个多样化的库,因此可能会导致产生大量功能性抗体。尽管如此,LCDR1中的大多数氨基酸都位于抗原结合位点的外围,并且在定义抗体的结合特性(特别是对于中小型靶标)中起着边缘作用[40]。因此,这样的库很可能用途有限。相反,相同的文库大小(例如11个位置突变为20个氨基酸),但在HCDR3中(可能位于抗原结合位点的中心并在识别各种靶标中起关键作用),可能会产生抗体具有合理的亲和力[41]。因此,多样性不仅是框内和/或无毒抗体变体的数目,而且还是能够识别尽可能多的多样靶标的功能分子的数目。
用作构建噬菌体展示抗体库的底物的第一个功能性抗体库是从免疫动物[42]或人类[43]获得的。这种类型的文库(称为免疫)作为人类治疗性抗体发现的通用平台,用途有限。从免疫动物获得的那些是一次性库,因为这些库倾向于识别用作免疫原的抗原。更重要的是,从免疫文库获得的抗体是非人类蛋白,需要进一步的工程设计(人源化),这增加了抗体药物开发过程的成本。对于具有免疫力的人类抗体文库,由于伦理原因,它们大多已用于分离抗传染病的抗体[44]。然而,值得注意的是,骆驼V区免疫组库的相对较新的研究显示出与人V种系基因库中相同的CDR构象[45]。这种相似性导致ArgenX开发了基于骆驼科动物免疫的发现平台。 ArgenX已经发现了几种有希望的治疗性抗体,这些抗体很难通过其他方式获得[46],其中一些处于临床试验的晚期阶段 ( https://www.argenx.com/en-GB/content/argenx-in-short/2/ ).
第一个人类通用或通用库于1990年代初发布[9]。该文库是用编码循环抗体(circulating antibodies)的人类基因库生成的。用这种抗体基因来源生成的文库因为没有偏向任何特定靶标而被称为天然(naïve)。尽管很成功,但天然的文库包括对大肠杆菌有毒的抗体基因,如上所述,这损害了文库的有效大小。随后合成抗体文库部分减轻了这种局限性[38]。在这种替代方法中,通过对支架的数量,多样化的位置,设计中要包括的氨基酸类型以及多样化每个位置中每种氨基酸的比例进行假设,精心设计了文库。这些假设并不总是成立,尤其是在HCDR3。为避免对CDR的结构做出假设,已使用合成支架与天然CDR的组合[47,48],从而构建了所谓的半合成文库。
表1包括四个天然的库和四个合成库,以及一个半合成库。 Xoma开发了两个天然的库[26];一个为Fab(XFab1),另一个为scFv(XscFv2)。库格勒等人出版了第三个naïve的库,位于不伦瑞克工业大学,生物化学与生物信息学研究所(TU-IB)。它结合了显示为scFv的kappa(HAL10)和lambda(HAL9)轻链。江原国立大学(KNU)开发了第四个也是最近发布的天然库[28]。它仅包含κ型轻链,因此我们将其称为KNU-Fab。表1中列出的合成库包括MorphoSys的两代库:HuCAL PLATINUM [30]和Ylanthia [31]。两者都是初始HuCAL和HuCAL GOLD [38,49]库的优化版本。此外,表1列出了Janssen Biotherapeutics的一种合成文库,即pIX V3.0 [29]。这是展示为pIX而非pIII融合蛋白的第一个Fab抗体库。第四个合成库是PHILODiamond [32]。这是一个仅基于三个支架,一个VH和两个替代VL,一个kappa和另一个lambda构建的简约库。 PHILODiamond是Dario Neri实验室在过去20年间生成的一系列抗体库的最新版本,该库的大小和复杂性不断增加,例如ETH2(EidgenössischeTechnische Hochschule 2)[50],ETH2Gold [51]和PHILO-1和PHILO-2 [52]。半合成库被称为ALTHEA(从希腊语为“治愈”)金库[33]。它是由我们的小组与抗体设计实验室(ADL)和三机构疗法发现研究所(TDI)合作出版的。 ALTHEA Gold Libraries™将一个VH支架与两个Vκ支架结合在一起。 HCDR3的多样性来自200个人类供体的庞大库。在以下各节中,我们将详细描述表1中列出的每个原始库,合成库和半合成库。
4.1 Naïve Libraries
Naïve的文库不对抗体库的多样性做任何假设。基本原理是人类抗体库进化为以合理的特异性和亲和力识别任何靶标。因此,构建纯天然文库的目的是反映人类抗体库的多样性,同时避免由于少数人的免疫学史和/或给定种族的库中存在的罕见多态性抗体基因而造成的偏倚和冗余。 Xoma使用30个种族不同的健康捐献者和各种组织,通过RT-PCR扩增VH和Vκ和Vλ链。组织样本包括20个外周血单核细胞(PBMC)样本,8个骨髓样本,1个脾脏样本和1个淋巴结样本。扩增策略涵盖所有免疫球蛋白(Ig)类:IgM,IgG,IgA,IgE和IgD。文库以Fab或scFv格式展示,以根据展示格式评估抗体选择中的潜在差异。两个文库都产生了相似数量的具有相似亲和力的独特抗体(下文将详细讨论),这表明显示格式不会显着影响选择的结果。
HAL9 / 10还使用了来自不同种族的血液样本,包括高加索人,非洲人,印度人和中国人。这些文库,一个κ(HAL10)和另一个λ(HAL9),包含从98个供体获得的相同的VH谱表,但它们的轻链谱表不同。 HAL9包括来自98个供体的所有lambda亚家族,而HAL10由54个供体生成,并包含除IGKV7假基因外的所有kappa家族。扩增策略包括用于衍生自IgM的V区的反向引物,因此有利于Naïve抗体基因的扩增,即接近种系构型,因此在VH中很少或没有体细胞突变。
与Xoma和HAL9/10库相比,使用更大的供体库生成KNU-Fab库。它包括803个PBMC供体,两个淋巴结供体,两个脾供体和两个骨髓供体,总共809个样本。从健康的韩国人类捐赠者那里获得了33份PBMC样本,从商业样本中获得了770份样本,这些样本可能来自不同种族背景的捐赠者。使用在V区杂交的正向和反向引物扩增抗体基因,因此与Ig类别,同种型,或抗体是否接近种系基因构型或具有大量体细胞突变无关。
对于KNU-Fab和HAL9/10文库,报告了选择文库之前的基因使用情况,而对于Xoma文库,仅描述了基因家族的使用情况。 研究了来自KNU-Fab库的总共7373个独特的VH序列和41,804个独特的Vκ序列,以评估在选择任何靶标之前的多样性。 同样,对于HAL9 / 10,分析了来自HAL9的827个全长scFv序列和来自HAL10的466个序列。 图2显示了KNU-Fab和HAL10报告的十个最普遍的IGHV(左)基因和五个最常用的IGKV(右)基因的频率。 作为参考,我们添加了Glanville [53]对辉瑞纯天然文库的基因使用和存放在数据库中的抗体序列的研究。
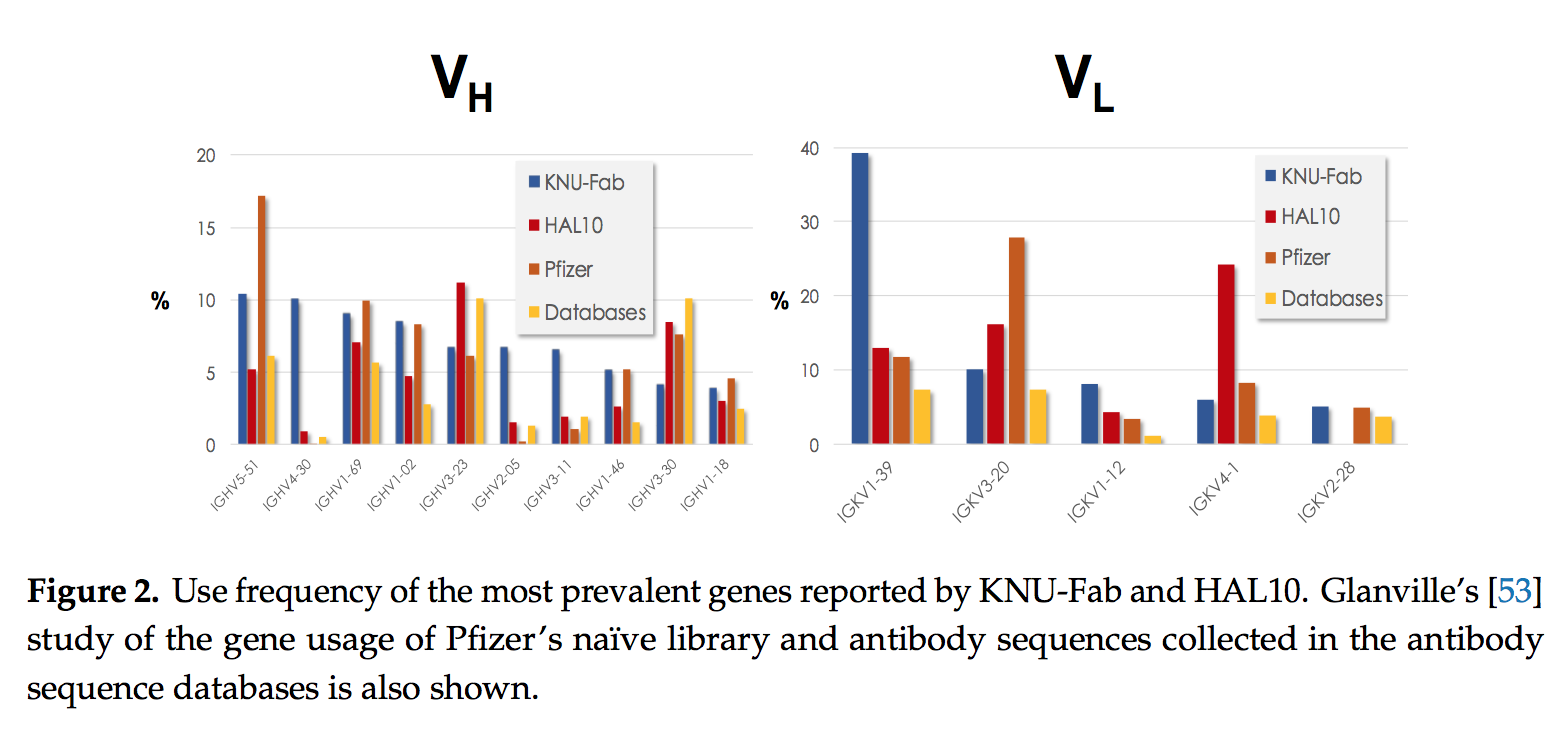
十个最流行的IGHV基因仅占人类基因组中≈50个潜在功能性IGHV基因的20%[54,55]。这十个基因覆盖了70%的KNU-Fab研究序列,接近所有HAL9 / 10研究序列的一半(47%)。除了IGHV4-30,IGHV2-05和IGHV3-11基因(它们在KNU-Fab中过量表达,但在其他样本中的频率较低)外,这些IGHV基因的频率在所有样本中均遵循相似的趋势。有趣的是,已发现来自IGHV4家族的基因在其他文库中被阴性选择[56],因为据报道它们对B细胞有毒性[57]。在VL中,人类基因组中≈35个[58](14%)潜在功能性IGKV基因中的五个IGKV基因解释了68%的KNU-Fab序列和58%的HAL10序列。 IGKV1-39在KNU-Fab中过分表达,占所有序列的近40%,而IGKV4-1基因在HAL10中过分表达,频率接近25%。有趣的是,除HAL10外,所有样品均表达IGVK2-28。综合起来,nature IGV基因在naïve文库中的用法表明,只有少数抗体基因足以覆盖噬菌体展示抗体文库中识别各种抗原所需的多样性。
关于HCDR3多样性,所有四个天然的文库都显示了在人类中观察到的HCDR3长度的典型高斯分布[59]。根据免疫遗传数据库(IMGT®)的定义[60],这两个Xoma库均具有相似的HCDR3长度分布,最常见的长度在5至25之间,平均长度为15个氨基酸,而13个氨基酸基于卡巴特的定义[36]。 HAL9和HAL10的HCDR3范围为5至35个氨基酸,低频的长度较长(25至35个氨基酸),中值为14个氨基酸(也是IMGT的定义),或者使用Kabat的定义为12。 KNU-Fab HCDR3的长度为4至19个氨基酸,最常见的HCDR3环的长度为11或12个氨基酸(Kabat的定义)。
总而言之,为了建立天然(naïve)的文库,使用了不同的人类供体库。在某些情况下,例如,在Xoma和KNU-Fab文库中,不管体型突变的同种型或数目如何,作者都尽可能地撒网以扩增所有人类基因。在其他情况下,例如HAL9 / 10,扩增策略仅限于种系基因配置中的序列。在所有文库中,只有少数抗体基因代表了所研究序列的大部分,与人抗体库中观察到的模式一致[53]。在所有文库中,HCDR3长度分布也相似,并且反映了人类抗体典型的高斯分布。
4.2 Synthetic Libraries
合成库旨在通过使用表达良好且可开发的支架,靶向多样化的位置(不破坏V区折叠)并选择氨基酸的类型和频率来促进抗体文库的功能最大化,从而有利于选择任意给定的不同结合物目标。第一个合成库HuCAL和HuCAL GOLD [38,49]由MorphoSys在1990年代后期开发。与其前身HuCAL GOLD一样,HuCAL PLATINUM(表1)由七个VH和七个VL主支架组成,这些支架(scaffolds)相结合时可产生49个抗体亚文库。这些支架被设计成具有代表人类库中IGV基因家族的共有序列。优化支架的序列以在大肠杆菌中高表达并在噬菌体上展示。使用三核苷酸诱变(TRIM)技术将六个CDR随机分组[61]。该合成技术基于编码20个氨基酸的三聚体,而不是寡核苷酸的混合物。这样,由于TRIM在目标位置实现了氨基酸的精确组合以实现多样化,同时避免了可能破坏抗体折叠的终止密码子和不需要的氨基酸,因此合成基因的质量显着提高。
GOLD和PLATINUM之间的主要区别之一是后者包括新设计的HCDR3序列。新策略基于对不同环长度的HCDR3序列每个位置的氨基酸使用情况的系统分析。然后基于不同的特定长度依赖的氨基酸使用频率,使用不同的氨基酸频率来设计合成的HCDR3片段,而不是针对所有不同长度的HCDR3相对均匀的氨基酸分布。此外,PLATINUM使用的环长度为4至23个氨基酸,覆盖了天然HCDR3长度的95%以上。此外,从Platinum中除去了通过NXT / S模式在HuCAL和GOLD中产生的潜在N-糖基化位点(其中N是天冬酰胺,X是任何氨基酸,S / T是丝氨酸/苏氨酸)。由于这些变化, HuCAL GOLD和PLATINUM的并排比较[30]表明,后者产生的独特抗体和亲和力抗体大约高出4倍。
Ylanthia [31]是MorphoSys迈出的又一步,旨在增强其抗体发现平台的性能。 Ylanthia并非共有的VH和VL主支架,而是在种系基因配置中使用了选定的VH:VL组合。导致最终设计的选择过程包括在人抗体库中最普遍的人种系基因和覆盖人抗体中所见的规范结构库的那些。典范结构是由Cyrus Chothia和Arthur Lesk [62]在1980年代后期发现的。这些作者发现,尽管CDR顺序有所不同,但是六个CDR中的五个(LCDR1,LCDR2,LCDR3,HCDR1和HCDR2)具有有限的主链构象或规范结构。规范结构模型在最近的二十年中已更新[63,64],并有助于开发3D建模策略[65]。从结构功能的角度来看,规范的结构模型表明结构约束在抗原识别中起作用[66]。最近,在300种非冗余抗体结构上应用聚类算法[67]通过识别具有规范结构的CDR长度的28种组合,进一步将规范结构组合进行了分层,而先前的分析[63]仅涵盖了20种。
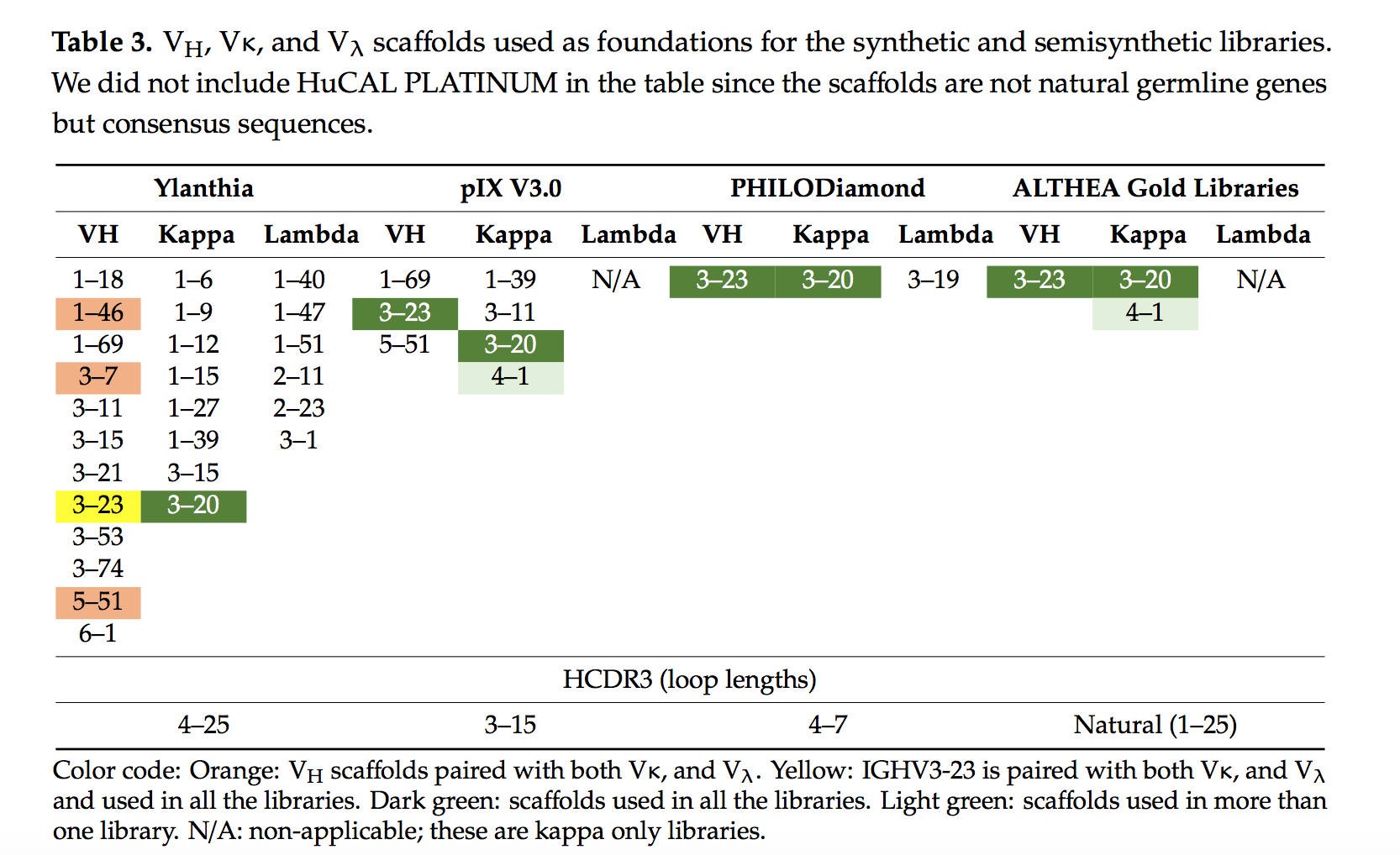
为了构建Ylanthia [31],这是第一个基于抗体基因使用和规范结构的计算机选择筛选器,生成了400个随机的VH:VL组合。 然后在几种可显影性试验中对这组支架进行实验测试。 该测定评估了Fab和IgG1形式的表达水平,热和血清稳定性以及聚集倾向。 另外,测试了最初的VH:VL组合的Fab CysDisplay的相对水平(MorphoSys使用的一个平台,该平台基于通过二硫键连接至噬菌体颗粒的抗体片段的表达)。 经过实验可开发性筛选后,使用36个VH:VL组合(包括12个VH,12个Vκ和8个Vλ支架)构建了文库(表3)。
除了对HuCAL系列产品的这一改进之外,基于对大量重排的人抗体序列和潜在的可开发性成库的系统分析,Ylanthia的CDRs也得到了多样化。 此外,在HCDR3序列的产生中使用了一种称为Slonomics [68]的新合成技术。 Slonomics是由Sloning Biotechnology开发并由MorphoSys收购的全自动蛋白质合成平台。 该平台基于编码20个氨基酸的双链DNA三联体的集合。 该平台可以高度保真地对各种组合基因文库进行高度受控的合成,即将设计氨基酸的预期频率与在文库中观察到的频率紧密匹配。 作为一种TRIM技术,这种新的合成方法避免了在设计位置出现终止密码子和不需要的氨基酸混合。
Janssen Bio的文库[29]是在pIX上展示的第一个组合合成Fab文库,而不是在所有其他文库中都使用过的融合伴侣pIII。用由人种系编码的三个VH和四个VL支架设计文库(表3和图3)。选择这些支架是基于它们在抗体人类库和naïve噬菌体展示库中的大量使用(请参见第4.1节“天然库”)以及结构上的考虑。具体而言,在文库中显示了最倾向于结合蛋白质和肽的规范结构[69,70,71,72,73]。 CDR在与蛋白质和肽靶标接触时经常出现的位置上多样化[37,40]。多样化机制反映了人类种系基因和从天然来源分离的抗体中观察到的氨基酸和频率的变异性[37,74]。建立了两套文库,一套称为pIX V2.0,通过将VL保持在种系基因构型中,具有针对VH的多样性。本文中讨论的另一个称为pIX V3.0,在VH和VL域中均具有多样性。
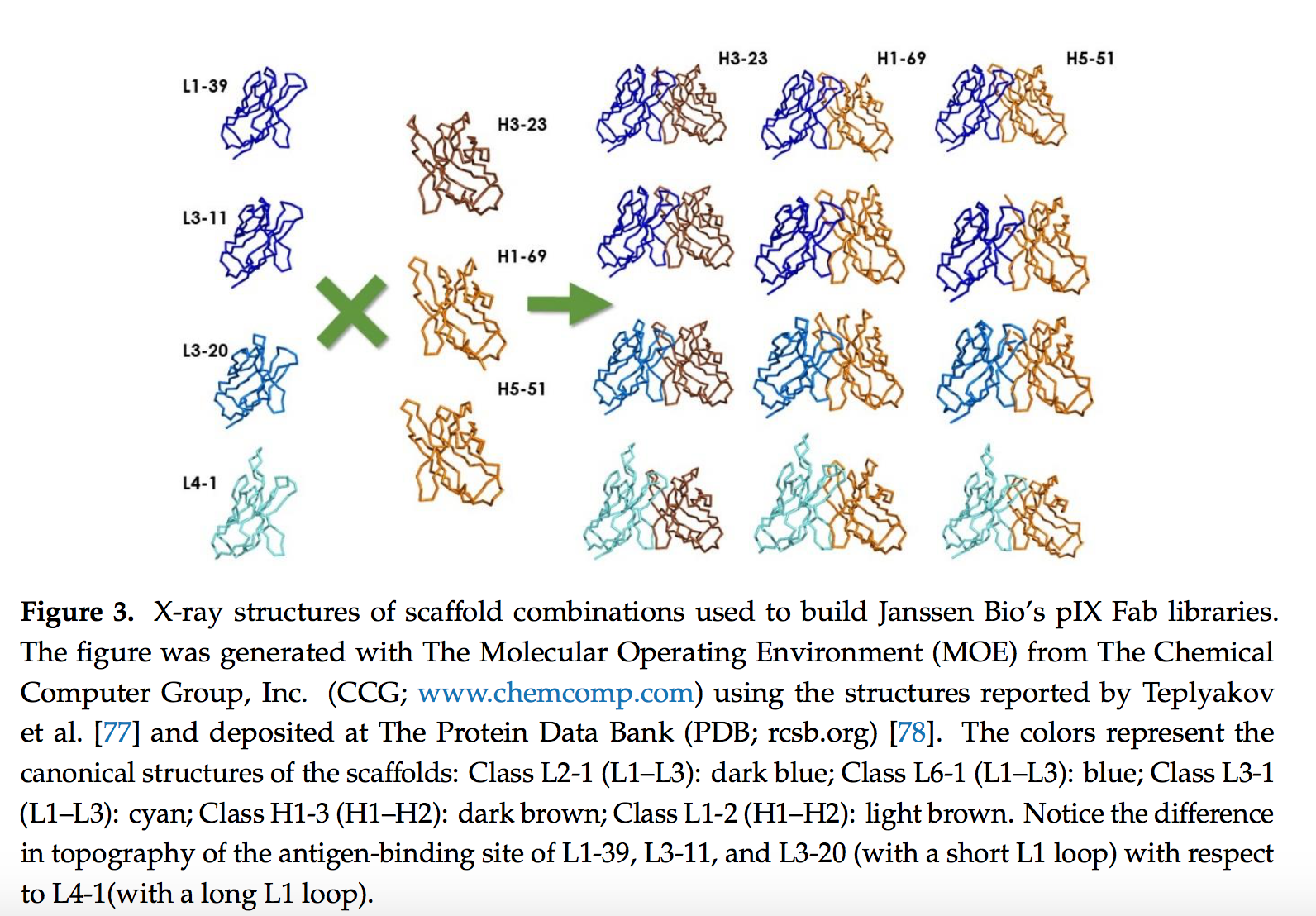
最近,Teplyakov等人[75]确定了pIX V3.0中使用的VH:VL组合的结构,这在其他合成库和ALTHEA Gold Libraries™(图3)中也很常见。在人抗体中观察到的两个VL支架(IGKV1-39和IGKV3-11)在LCDR1处的环最短[76]。另一个(IGKV3-20)在LCDR1中有一个插入,尽管它仍然比较短。第四个支架(IGKV4-01)是人类种系基因库中最长的LCDR1,相对于IGKV1-39和IGKV3-11插入了六个残基。将LCDR1的长度从短环更改为长环,分别改变了结合蛋白或肽靶标的偏好[37,69]。因此,包含这些具有不同LCDR1长度的IGKV基因为pIX V3.0库提供了识别各种类型靶标的潜力。在VH中,虽然在所有三个支架中HCDR1的长度都相同,并且发现IGHV3-23和IGHV5-51中HCDR1的构型非常相似[75],但IGHV1-69却显示出较大的结构变异性。这可能是由于IGHV1-69的HCDR1中有两个甘氨酸残基,从而提供了更多的构象自由度。此外,HCDR2具有两个替代构象,一个在IGHV3-23 * 01和IGHV5-51 * 01中,另一个在IGHV1-69中。综上所述,在pIX V3.0中看到的结构变异性为库提供了独特的拓扑结构和结构多样性,以识别各种目标。
Teplyakov等人还评估了Fab形式的pIX V3.0 VH:VL支架组合的热稳定性(Tm)。 [75]。除IGHV3-23 * 01:IGKV * 01以外,所有VH:VL支架组合的Tm值均高于68°C。考虑到人类IgG1(hIgG1)的最不稳定结构域是温度为68°C的CH2 [79],因此预期从pIX 3.0衍生的Fabs在转化为hIgG1时应该可以developable,这是最常用的治疗形式[ 12]。
PHILODiamond [32]仅用三个支架制成:一个VH(IGHV3-23)和两个VLs(图3)。 VL支架为κ(IGKV3-20)或λ(IGLV3-19 * 01)。 PHILODiamond是ETH2Gold库的新版本[52]。 ETH2Gold库的改进包括新的HCDR3设计,其长度为4至7个残基,每个位置有20个氨基酸。在ETH2Gold库中,HCDR3具有四个和六个随机的连续氨基酸。 PHILODiamond还具有LCDR3完全多样化(20个氨基酸)的五个或六个位置。此外,VH的残基52被设计为天冬酰胺(N),以促进氢键相互作用。
4.3 Semisynthetic Libraries
如前一部分所述,已经进行了多次迭代来提高合成文库的质量,其中HCDR3的设计是一个不变的主题,并且也许是主要的改进机会。 HCDR3是定义抗体的特异性和亲和力的关键要素,但它也是迄今为止抗原结合位点最多样化的区域,因此难以设计。 3D建模方法[80,81]可以预测HCDR3以外的所有CDR的结构,准确度<1.0Å[81]。 但是,目前尚无方法可以可靠地预测HCDR3结构,从而限制了我们正确设计此抗原结合位点区域多样性的能力。
为了避免对HCDR3的结构和多样性做出任何假设,ALTHEA Gold Libraries™[33]内置了HCDR3和从天然来源分离的连接片段(H3J片段)。将天然H3J片段与合成支架相结合,合成支架是基于人类种系基因设计的,该基因在人类抗体以及众多scFv和Fab文库中占主导地位(表3)。一个通用的VH支架与两个VL支架配对。与在pIX V3.0中一样,我们使用了一个VL支架来识别蛋白质(IGKV3-20)和其他结合肽(IGKV4-01)。因此,通过使用适当的VL支架,我们假设可以选择针对蛋白质或肽靶标的抗体[37,69]。当组合使用时,它将潜在地产生结合给定靶标上不同表位的抗体。同样,由IGHV3-23 * 01种系基因编码的通用VH支架的HFR3与金黄色葡萄球菌的蛋白A天然结合[82,83]。 VH结构域中的A蛋白结合位点是通过折叠折叠在一起的一级序列中不连续的氨基酸序列形成的。因此,蛋白A提供了一种在ALTHEA Gold Libraries™的构建过程中选择折叠良好的scFv的方法。
我们使用了三步策略(图4)来生成库。 第一步,设计,克隆完全合成的抗体文库( synthetic primary antibody libraries,PLs),并以scFv的形式展示在噬菌体表面。 其次,我们进行了选择过程,在该过程中,PL经受了热冲击,并进一步与蛋白A一起选择了框架内和热稳定的变体。 我们称此步骤为过滤库(FL)的产品。 第三,通过将FLs与从200个供体的大量库中获得的天然H3J片段结合,生成了功能强大且高度多样化的二抗文库(SLs)。 通过使用此三步构建过程,被评估为从文库中随机选择的A蛋白结合物的ALTHEA Gold Libraries™的功能从PL中的≈65%增加到SL中的≈85%,这意味着提高了20%。 就10^10个变体的库中功能性克隆的数量而言,这意味着要选择2×10^9个其他抗体序列。

五、平移协议和目标 Panning Protocols and Targets
表4显示了靶标的数量,每个文库平均研究的克隆数,命中率以及从前面部分讨论的9个文库中选择的抗体的亲和力。 使用固相或与生物素化靶标进行生物淘选进行选择。 在大多数选择方案中,使用了三轮平移(Panning)。 PHILODiamond进行了两轮选择,KNU-Fab和ALTHEA Gold Libraries™进行了四轮选择。

目标数量从pIX V3.0 [29]的6个到HAL9 / 10 [15]的440个不等。作为靶标的一个例子,Xoma用肽和蛋白质(包括胃泌素14-mer,β-半乳糖苷酶和TIE-1-Fc嵌合体)进行了文库的淘选。对于Ylanthia,主要使用蛋白质,例如重组人(rh)ErbB4,rhTNFα和人IgG1抗体来产生抗独特型抗体。 PHILODiamond已通过纤连蛋白,胶原蛋白I,腱糖蛋白C(BCD)和人基质金属蛋白酶1(MMP1)和3(MMP3)进行了测试。 MorphoSys用衍生自HuCAL GOLD文库的人Fc融合蛋白,CD20和人IgG1抗体测试了HuCAL PLATINUM。 Kim等 (KNU-Fab)使用粘附分子(L1CAM),血管生成素2(Ang2)和激活诱导型TNF受体配体(AITRL)。 ALTHEA Gold Libraries™的作者除了其他四个未公开的治疗靶标之外,还探索了具有学术靶标HEL,治疗靶标人血清白蛋白(HSA)和TNFα的文库的功能。
六、克隆阳性和命中率丰富 Enrichment with Positive Clones and Hit Rate
最后一轮选择后,使用直接ELISA对靶标进行阳性克隆的筛选。在大多数选择中,牛血清白蛋白(BSA)或牛奶被用作特异性对照。分析结合的克隆数量差异很大。 ALTHEA Gold Libraries™和KNU-Fab库筛选了从最后一轮淘选中随机挑选的45–90个菌落。 Xoma分析了数百个菌落,并针对数千个HuCAL PLATINUM,Ylanthia和HAL9 / 10进行了筛选。 XscFv2中阳性克隆的平均百分比也从大约20%到接近60%不等。对于某些目标,例如淘选PHILODiamond时的胶原蛋白I,结果只有少数(1%)阳性克隆。当使用XscFv2文库进行淘选时,其他目标(例如Tie2 / Ang1和Tie2 / Ang2)分别产生多达85%和88%的阳性克隆。在具有挑战性靶标(例如抗独特型选择)的HuCAL PLATINUM中,观察到较低百分比的阳性和独特克隆,其中靶标上的独特表位仅限于抗体的抗原结合位点[30]。
大多数作者将命中率定义为阳性和独特序列的数量。 MorphoSys(HuCAL PLATINUM和Ylanthia)将独特的克隆算作HCDR3序列。尽管存在这些差异,但所有库中的平均命中率都非常相似(约10%),范围从2%到40%。无论文库如何,总体相似的命中率都意味着,随着筛选出的克隆数量的增加,获得的独特克隆的数量也会增加。因此,如HuCAL PLATINUM所示,涉及十个96孔常规ELISA板(≈1000个克隆)的标准筛选有望产生100个或更多个独特的抗体。大量独特的克隆也意味着所选抗体变体的多样性更高,这反过来又转化为数量更多的功能更好,更易于开发的抗体。 HuCAL GOLD和PLATINUM的并排比较[30]显示,在阳性克隆方面,文库的性能没有显着差异。但是,与GOLD的结果相比,在PLATINUM选择中发现了更多的独特抗体库。如上所述,PLATINUM是GOLD的新一代,具有一些改进,例如消除了可开发性负债和更好的HCDR3设计。
七、选定抗体的亲和力
对于许多疗法而言,更高的亲和力抗体是理想的甚至是必需的。较高亲和力的抗体可能还需要较低的剂量,这除其他因素外,直接影响了商品和治疗成本的降低。因此,自从噬菌体展示方法首次应用于治疗性抗体发现以来,亲和力一直是评估文库性能的最相关成功标准之一[17]。表1中的所有文库,除PHILODiamond和KNU-Fab外,均报告了亚nM结合物。从PHILODiamond KD值获得的抗体通常在9到150 nM之间。值得注意的是,亲和力用单体scFv片段测量。因此,可以预期,由于亲和力作用,当以IgG测量时,这些结合物中的一些会达到接近或低于nM的亲和力。另一方面,KNU-Fab将三个Fabs转化为IgG。用竞争性ELISA测定,其中之一对人和小鼠靶标的亲和力分别为7.3nM和14nM。对于小鼠靶标,另一种抗体的KD值为1.7 nM。
八、可开发性
近十年前,可开发性的概念被应用于抗体药物的开发[18]。可开发性包括分子的设计原理和对分子特性的实验评估,这些分子需要进一步开发或制造,配制和稳定化,以实现所需的治疗效果。Developability受多种因素影响,包括分子的固有生物物理和生化特性,以及诸如离子强度,pH和配方添加剂等外部参数。随着越来越多的抗体进入市场,并且许多抗体未能在临床前开发和临床试验中发挥作用,人们已经认识到,尽管具有所需的特异性和亲和力,但选自多种文库的抗体在配制和生产开发过程中往往会失败生物物理特性欠佳而导致的过程[84,85]。这包括可以削弱结合的抗原结合位点残基的糖基化。这对于选择用于噬菌体展示文库的抗体来说尤其重要,因为大肠杆菌不会糖基化蛋白质。因此,可以在噬菌体展示发现活动期间选择在CDR中具有糖基化位点的变体,但是当在哺乳动物细胞中转化并表达以进行进一步表征时,它们会失去结合。未成对的半胱氨酸可能导致混乱的二硫键,从而产生共价聚集体。此外,抗体会经历氨基酸的翻译后修饰(PTM),例如天冬酰胺(N)脱酰胺,蛋氨酸(M)氧化和天冬氨酸(D)异构化[79]。如果所述氨基酸参与抗原相互作用,则这些氨基酸化学修饰可导致抗体制备中的异质性和/或效力不足。在其他情况下,色氨酸(W)暴露于溶剂会诱导聚集,从而导致免疫原性反应或在治疗指征所需的浓度下缺乏溶解性[85]。
仔细研究来自naïve文库的抗体序列,发现某些选定的抗体编码了developability。例如,选择了IGHV7家族的唯一人类功能基因(IGHV7-4-1 * 01),其在HAL10和HAL9中的频率分别为0.4%和0.7%。该人类基因在HFR3(UniProtKB:A0A0J9YVY3)中具有未配对的半胱氨酸,这可能导致混乱的二硫键。来自IGKV1家族的另外两个基因(IGKV1-8 * 1和IGKV1D-8 * 1)在LFR3处具有N-糖基化位点,并在HAL10中以0.8%的频率选择。除了在CH2结构域具有典型N-糖基化的糖基化抗体外,由于其在生产过程中可能导致异质性,因此未进行进一步开发[79]。因此,这些基因损害了文库的有效大小和功能,和/或在选择过程中可能与可发展的序列竞争。显然,可以通过增加筛选过程中研究的抗体数量(见下文)和/或通过扫描序列并除去具有不希望的developability liabilities的序列,和/或通过实验评估可开发性来规避从naïve库中获得可开发抗体。 [19]。然而,更多的筛选和/或其他测试需要更高的成本和更长的开发时间。
新一代的合成库和半合成库已合并了过滤器,以消除设计阶段的developability liabilities。而且,生物物理测定法已被用于评估用作文库基础的支架的可发展性。例如,在Ylanthia的构建中[31],使用了几种可开发性测定法,以从一组400个初始VH:VL对组合中选择最可开发的支架。有趣的是,在使用不同靶点进行选择后,大多数分子紧密保留了亲本VH:VL支架对的生物物理特征,而轻微的生物物理差异则归因于某些HCDR3序列的影响[31]。 ALTHEA Gold Libraries™的构建过程包括可开发过滤器,因为它遵循的过滤过程包括70°C的热冲击。此过程导致使用多种靶标选择的抗体在70°C或更高温度下的热稳定性提高≈20%[33]。
九、当前的机遇与挑战
噬菌体展示方法已被证明是发现和开发人类治疗性抗体的宝贵,强大而有效的平台。然而,直到最近,噬菌体展示的商业用途仅限于少数拥有其技术专利的公司[16]。实际上,据我们所知,表1中列出的库不可用于研究目的。尽管如此,大多数涉及噬菌体展示的专利已在欧洲和美国失效[16],为自由使用噬菌体展示提供了巨大的机会。随着这种方法学成为一种商品,数家公司都在许可噬菌体展示抗体库,例如ALTHEA Gold Libraries™( https://www.globalbioinc.com/Services/ )。此外,Bio-Rad实验室还以相对较低的成本提供了基于HuCAL PLATINUM( https://www.bio-rad-antibodies.com/hucal-ordering-information.html )的发现服务。另一方面,剑桥大学或苏格兰生物学馆(https://www.abdn.ac.uk/sbf/about/sbf-libraries/ )的学术实验室可以使用2010年之前生成的许多噬菌体展示抗体库(此处未进行评论,但已得到广泛验证)。这些文库包括由英国医学研究理事会建立的称为Tomlinson的文库[86]的合成抗体文库,以及由John McCafferty和他的同事生成的10^10多种人类抗体的纯天然文库。汤姆林森(Tomlinson)的合成文库已针对许多靶标产生了特异性和高亲和力的抗体[87,88],而麦卡菲蒂(McCafferty)的naïve的文库已被用于选择,筛选和测序针对约300种抗原的约38,000种重组抗体。
关于改善当前文库的机会,理想的抗体文库应以最小的努力,即仅两到三轮淘选而产生多样的,高特异性和高亲和力的抗体,以及可发展的分子。构建抗体噬菌体展示文库时,一个重要的参数,也许是最简单的参数,就是它的大小,因为它与选择较高亲和力抗体的可能性相关,如Perelson所预测的[34]。表1中描述的文库达到了≈10^11抗体变体的最大可能大小。有了这些文库大小,几乎可以从几乎所有文库中获得多样化,高度特异性和亚nM的结合物,而不论是抗体naïve,合成的还是半合成的抗体库类型。库的有效大小平均为85%,这意味着如果可以达到100%的功能,则有15%的改进空间。 10^11个分子库中的15%的改进意味着可以选择1.5×10^10个其他抗体变体。
为了提高文库的有效大小,可以采用更好的DNA合成方法,通过减少目标靶位上的终止密码子,移码和多余和/或不需要的氨基酸来增加ORF的百分比,以实现多样化。当前的合成平台,例如Twist Bioscience的基于硅的DNA合成平台,通过下一代测序验证( https://www.twistbioscience.com/libraries_poster_precisionsynthesis ),可以在每个库中均匀整合变体,从而精确合成变体。可以将这种精确的合成方法与具有增强的developability的抗体分子文库结合使用。这可以通过在库的设计阶段结合更好的可开发性预测方法来完成。在这方面,新的计算机软件[89]可以识别与治疗性抗体相比具有异常可开发性值的抗体序列。这项称为治疗性抗体分析(TAP, therapeutic antibody profiling)的指标可建立抗体V序列的可下载结构模型,并针对可能与可开发性差有关的五种计算指标的指导性阈值进行测试。因此,通过计算机生成抗体库的每个可能变体并滤除TAP较差的那些,可以用可开发的变体代替不可开发的变体。
最后,值得一提的是,除scFv或Fab以外的其他格式,例如单域抗体(sdAbs)或纳米抗体,均已得到广泛测试,并成功用于构建噬菌体展示文库,以发现和优化基于抗体的药物[90, 91,92]。这些格式的灵感来自VHH骆驼科抗体[93]和鲨鱼的新抗原受体(NAR)[94]。由于其大小和高稳定性,纳米抗体已发现了多种治疗应用,并且治疗性分子,例如来自Ablynx的caplacizumab [95],最近已商业化。另一方面,已经开发了除噬菌体以外的展示平台,并用于发现治疗性抗体。这些平台包括核糖体[96],细菌[97]和哺乳动物[98]展示方法。这些平台在噬菌体展示方面各有利弊,酵母是最广泛使用的展示平台之一[99]。酵母展示已被证明是一种以非常高的亲和力(例如在低飞摩尔范围内)分离抗体的有效方法[100]。此外,尽管噬菌体限于展示抗体片段,例如sdAb,scFv或Fab,但是酵母能够展示具有糖基化的完整IgG抗体。由于最终的治疗产品通常是IgG,并且其功效和毒性是靶标表位,亲和力,Fc同种型和糖基化之间的相互作用,因此酵母展示已成为有效治疗发现和开发的合适平台[101]。实际上,基于酵母展示技术的抗体治疗发现公司Adimab最近宣布了在中国的生物制品许可申请(BLA)批准,该药物可抗PD-1的抗体来治疗霍奇金淋巴瘤( https://www.fda.gov/vaccines-blood-biologics/development-approval-process-cber/biologics-license-applications-bla-process-cber )
十、结束语 Concluding Remarks
自在三十年前,George Smith[1]和McCafferty等人的开创性工作以来,抗体工程领域在治疗性抗体的发现和开发中已取得了显着进展。该进展已经建立,并且部分是几代噬菌体展示抗体库的产物。在前面的部分中,我们回顾了过去十年中生成的九个库。目的是评估噬菌体展示抗体发现的最新技术,并概述各种文库之间的共性和差异。文库达到了10^10–10^11抗体变体,最大可能的大小。有效大小(理解为展示的百分比)为50-90%,大多数库达到85%。这样的文库产生nM或亚nM的结合物。有趣的是,文库的平均命中率相近(约10%),而与用于构建文库的底物的naïve,合成或半合成抗体性质无关。主要区别似乎在于抗体的多样性和可开发性。虽然naïve文库具有捕获抗体自然库的优势,但从这些文库中选择的某些抗体可能承担可开发性责任。为了避免这种限制,合成库和半合成库进行了多次迭代以改善其设计,尤其是在HCDR3上。这些新的库,加上该领域最近的专利到期,应该允许学术实验室和小型生物技术组织免费使用噬菌体展示方法。抗体噬菌体展示的广泛应用应能促进创新,进一步探索各种新颖的靶标,并在噬菌体展示方法方面产生新颖的改进。
参考资料
- Antibodies 2019, 8(3), 44. Phage Display Libraries for Antibody Therapeutic Discovery and Development ; https://doi.org/10.3390/antib8030044 。文献位置: https://www.mdpi.com/2073-4468/8/3/44/htm
