5.2 找影响系统发生的相关因素--UniFrac
Fast UniFrac是 UniFrac 软件的较新版本。是为了在你的样本中更好的寻找影响系统发生的相关因素。
物种的多样性指标共分为三种分别为:
- α多样性:引用于再特定地区、族群、生态系内的多样性,其测量系计算出生态系内种类的数量(通常以species (种)为单位,species 是生物分类中基本的分类单元和分类等级。微生物的种可以看作是:具有高度特征相似性的菌株群,这个菌株群与其他类群的菌株有很明显的区别。) 即我们通常所说的多样性,指一个集合体或者一个环境下的多样性,比如一个环境中含有多少物种,以及各物种的数目等
- β多样性:是生态系之间的种多样性,它包含分类单位的比较生态系内的每一独特性。即衡量群落之间的差别
- γ多样性:在一个地区内不同生态系的全部多样性的量测值
UniFrac该软件主要研究的是β多样性,侧重的是之间的比较。 该软件使用的是进化距离来对不同的样本进行分类。主要是通过进化树中的几点到上已节点的距离来进行分类。
β多样性基于OTU的群落比较方法和基于系统发生树比较:
- 基于OTU的群落比较方法:β多样性可以分为定性的度量和定量的度量两种。定性的度量,包括Jaccard指数,Dice系数等,只考虑每种OTU在群落中出现/不出现,而不考虑它们的丰度。而定量的度量, 包括Bray-Curtis距离,Canberra距离,欧氏距离等,考虑的是每种OTU在群落屮的丰度
- 基于系统发生树比较生物群落的方法:是指在比较群落的过程中,将序列之间的进化关系纳入考虑。基于OTUs的𝛽多样性度量有两个主要的不足:其一是很难保证OTUs被准确的定义,定义OTUs时发生的错误将直接导致比较群落关系时产生错误的结果;其二是所有的OTUs都被同等的对待,然而实际上有些OTUs相对更相近,而有些则差别很大。
UniFrac和加权UniFrac:
另外两个得到广泛应用的群落比较方法是UniFrac和加权UniFrac,它们也是基于系统发生关系的。UniFrac和加权UniFrac分别于2005年、2007年提出,可以用于衡量群落之间的距离,至今,已经应用于许多大型的研究项目,并得到一些重要结果。与P-检验类似,应用它们的先决条件是一个包含所有待比群落里所有序列的系统发生树(有根树,各枝长已知),每个序列根据其来自的排落进行标注。若比较多个群落,则往往两两比较,而每一次的比较,都在原始的 系统发生树的基础上,将其中不存在于这两个群落的叶节点去掉,形成一个只含这两个群落序列的子树。基于这样的子树,UniFrac这样定义两个群落之间的距离:对于系统发生树所有枝,考查其指向的叶节点是否只存于同一个群落,那些叶节点只存在于同一群落的枝的枝长和,占整个树的枝长和的比例,就定义为UniFrac距离。UniFrac的概念非常容易理解,直观来讲, 就是计算了仅被一个群落占据的进化历史的相对大小,这个量越大,说明两个群落中独立的进化过程越多,也就说明这两个群落的差别越大。若两个群落完全相同,那么它们没有各自独立的进化过程,UniFrac值为0;若两个群落在进化树中完全分开,即它们是完全独立的两个进化过程,那么UniFrac值为1.
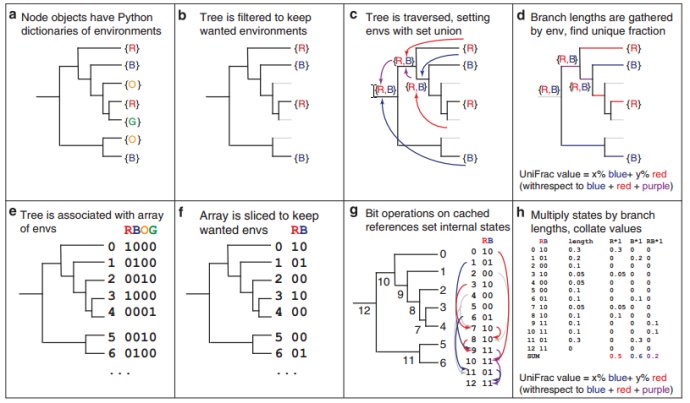
从UniFrac的定义中,可以看出它只考虑序列是否在群落中出现,而不考虑序列的丰度。若两个群落包含的物种完全相同,那么不管每个物种的丰度是否有差别或者差别的大小,UniFrac值为0。但在某些具体的情况下,研究者感兴趣的恰恰是群落中物种丰度的变化,例如研究人体肠道微生物分布在抗生素治疗下的变化情况,这时UniFrac就不能解决问题了。 W-UniFrac方法,就是在UniFrac的基础上,将序列的丰度纳入考虑,它能够区分物种丰度的差别。在计算中,W-UniFrac按照每条枝指向的叶节点中来自两个群落的比例,给每条枝加权重。W-UniFrac的计算公式如下:


总结一下,这个unifrac根据进化树的信息判断出了俩俩环境样品的距离,然后再根据PCoA即可作图
???下面这张图以及相应的几个分类没有搞明白,先留个悬念吧,过段时间再来补充 UniFrac can perform several different phylogenetic analyses on the tree and environments you uploaded. UniFrac Significance tells you which pairs of environments are significantly different using the UniFrac significance test. P Test Significance tells you which pairs of environments are significantly different using the P - test. Lineage-Specific Analysis allows you to break the tree up into the lineages at a specified distance from the root, and to test whether any particular group contributes to the overall differences between environments. Cluster Environments uses the UniFrac metric to cluster the environments based on the phylogenetic lineages they contain. Jackknife Environment Clusters performs statistical resampling to tell you which clusters you can be confident of. PCA uses the UniFrac metric to perform principal coordinates analysis on the environments, allowing you to see whether different types of environments are separated in different dimensions. Environment Counts tells you how many sequences are in each environment. Environment Distance Matrix shows you the UniFrac distances between each pair of environments. This distance matrix is what is used as input for environment clustering and PCA.
参考资料
- http://blog.sciencenet.cn/blog-491564-667282.html
- YucaiFan的博客 http://blog.sina.com.cn/s/blog_83f77c9401014kkd.html
- http://unifrac.colorado.edu/
- 文献:UniFrac: an effective distance metric for microbial community comparison
- 文献:UniFrac – An online tool for comparing microbial community diversity in a phylogenetic context
- 文献:宏基因组数据分析中的统计方法研究(赞一个)
- 文献: Fast UniFrac: facilitating high-throughput phylogenetic analyses of microbial communities including analysis of pyrosequencing and PhyloChip data
