【6.1.2】tRNA和tmRNA基因中精确定位的基因组岛的数据库(Islander)
基因岛是可移动的DNA,是细菌和古细菌进化的主要媒介。整合入原核染色体,通常在酪氨酸整合酶的催化下,特异地出现在tRNA或tmRNA基因(共同,tDNA)靶标上。这样就分裂了靶基因,但是岛内的序列恢复了被破坏的基因。再生的目标及其位移的碎片精确地标记了该岛的端点。我们采用这一原理来寻找基因组DNA序列中的孤岛(island)。我们的算法可以识别tDNA,在相同的复制子中找到这些tDNA的片段,并通过一系列过滤器去除不太可能的候选岛。对2168个完整原核基因组中的岛屿进行的搜索产生了3919个候选对象。 Islander网站( http://bioinformatics.sandia.gov/islander/ )显示了这些精确定位的候选岛,基因含量和岛序列( island sequence )。该算法还坚持认为,每个岛都编码一个整合酶,并仔细记录了连接位点序列的同一性。因此,该数据库还可用于整合酶位点特异性及其进化的研究。
一、安装与使用
1.1 安装
cd /home/sam/software/
wget -c https://bioinformatics.sandia.gov/software/IslanderV1.2.tgz
tar -xvf IslanderV1.2.tgz
chmod 775 install_dependencies.sh
./install_dependencies.sh
报错1:
pftools configure: error: Fortran 77 compiler cannot create executables
解决办法:
yum search gfortran
yum install gcc-gfortran.x86_64
报错2
Can't open perl script "/usr/local/bin/tRNAscan-SE": 没有那个文件或目录
解决经过:
尝试重新安装,尝试添加到相应的路径,
perl -p -i -e 's#\$\(HOME\)#/usr/local/src/tRNAscan-SE#' Makefile
cd /usr/local/src/tRNAscan-SE/lib
cp -fr tRNAscan-SE /usr/local/lib
cd /usr/local/src/tRNAscan-SE/bin
cp -fr * /usr/local/bin
BEGIN failed--compilation aborted at /usr/local/bin/tRNAscan-SE line 28.
echo 'PATH=$PATH:/usr/local/src/tRNAscan-SE/bin/' >> ~/.bashrc
echo 'PERL5LIB=$PERL5LIB: /usr/local/src/tRNAscan-SE/bin/' >> ~/.bashrc
source ~/.bashrc
。。。。 最后发现是权限的问题,普通用户不能读。。。
1) Add /usr/local/tRNAscan-SE/bin to your PATH variable
2) Add /usr/local/tRNAscan-SE/bin to your PERl5LIB variable
3) Add /usr/local/tRNAscan-SE/man to your MANPATH variable
报错3
./bin/hmmsearch --tblout example/NC_000913.tbl --domtblout example/NC_000913.domtbl --cpu 30 --cut_tc ./db/Pfam-A.hmm example/NC_000913.faa
Error: Unrecognized format, trying to open hmm file ./db/Pfam-A.hmm for reading
报错原因: hmm和pfam版本不匹配
[sam@localhost Islander_software]$ ./bin/hmmsearch -h
# hmmsearch :: search profile(s) against a sequence database
# HMMER 3.0 (March 2010); http://hmmer.org/
[sam@localhost db]$ head Pfam-A.hmm
HMMER3/f [3.1b1 | May 2013]
解决办法:
下载相对应的Pfam
wget -c ftp://ftp.ebi.ac.uk/pub/databases/Pfam/releases/Pfam25.0/Pfam-A.hmm.gz
安装相应版本的hmm
tar zxf hmmer-3.1b1.tar.gz
cd hmmer-3.1b1
./configure --prefix /usr/local/hmmer-3.1b1 # 安装到/usr/local/hmmer-3.1b1
make
make check
make install
cp /usr/local/hmmer-3.1b/bin/hmmsearch /home/sam/software/Islander_software/bin/
1.2 使用
----------------------
Command Line Arguments
----------------------
Example: perl Islander.pl example/NC_000913 --verbose --translate --trna --annotate --reisland --table 11 --nocheck
Input:
The only necessary file is a fasta file, labeled .fna
The relative or absolute paths can be provided, but the input filename will leave off the .fna
--verbose
(Optional) This option lists a number of additional output features
--translate
(Optional) This step runs prodigal on the original fna file and outputs a file of genes, .faa
If this is not called, the remaining programs will require a .faa file to be in the path of the .fna file
--trna
(Optional) This step identifies all the tRNAs and tmRNAs in the genome
It requires several additional perl scripts in the bin folder: tFind.pl trnarawFixer.pl
It also needs blastn and makeblastdb to be in the path
--annotate
(Optional) This step annotates the translated sequences and creates a .gff file
It requires additional perl scripts to be in the bin folder: dom_to_faa_gff.pl
It also needs hmmsearch to be in the path
It also requires an annotation source for the hmms: db/Pfam-A.hmm
This step is far and away the slowest part. If faa and gff files already exist, store them in the directory
and avoid this and the translate steps.
--reisland
(Optional) This step tells it to run islander, even it has been run previously
--table
(Optional) This will probably be deprecated
--nocheck
(Optional) This allows previous files to be overwritten
By default the program then determines all integrases
It requires perl scripts to be in the bin folder: integrase_finder.pl
It also requires database files:
db/PF00589_seed.hmm
db/famint9.hmm
db/xers.prf
The program then runs the island finding script
It requires perl scripts to be in the bin folder: island_finder.pl
Output:
Several files are output:
1) example.faa
2) example.trnSS
3) example.Aragorn
4) example.Bruce
5) example.trnaraw
6) example.trnaraw.fix.fna
7) example.trnaraw.fix
8) example.nsq
9) example.nin
一、介绍
基因岛(Genomic islands)是水平转移的DNA片段,整合到原核染色体中。当对第二个大肠杆菌基因组(O157:H7)进行测序并可以与大肠杆菌K-12基因组进行比较时,他们的视野特别清晰,从而发现了O157:H7独有的许多大岛,其中一些岛是K-在相同的染色体位点发现了12个甚至其他应变特异性岛(1,2)。许多这样的岛屿为它们的两个最基本特性提供了它们自己的机制的线索: 整合与流动(integration and mobility)。像原型(prototypical)基因组岛,噬菌体拉姆达(lambda)一样,许多岛屿包含:
- 包含整合酶基因的整合模块,以及
- 噬菌体结构和调控基因,可为细菌的跨细菌迁移提供现成的假设。
通过噬菌体颗粒在岛上。因此,可以将一类主要的基因组岛识别为特定地点的综合预言。第二大类岛屿显示了整合模块以及IV型分泌系统(T4SS)的基因,表明它们像许多质粒一样穿过缀合菌毛。这些被称为整合共轭元件( integrative conjugative elements)。
岛屿无需将自己的移动性或集成功能编码为可移动和集成的。既没有噬菌体也没有T4SS基因的岛屿可能仍含有动员信号,卫星噬菌体和可动员质粒也有动员信号,它们可进入由辅助元素编码的移动性载体。并非所有的孤岛都编码一个整数。有些依赖于整合酶家族宿主酶Xer,该酶通常负责解析二聚体染色体(5)。
除了具有移动性和整合性的孤岛功能外,孤岛还可以携带有益于宿主细菌的货物基因,从而促进例如致病性(如术语“致病岛”(6)所述),共生或分解代谢途径。载货岛屿(Cargo-bearing islands)是细菌进化的主要媒介。
如果可以确定岛的精确终点,则可以从基因组序列中重建基因组岛的假设的整合前或切除后的环形形式。这些圆圈有一个约250 bp的DNA片段,称为attP,其中整合酶的作用是促进与染色体中特定靶位点attB的重组。对于大部分整合岛(约30–50%)(9,10),attB位于tRNA或tmRNA基因(统称为tD-NAs)内。在这些情况下,孤岛attP包含tDNA靶标的片段,因此尽管破坏了原始tDNA,整合仍能恢复功能性tDNA。这在染色体上留下了一个生物信息学上可检测的岛标记:一个tDNA和一个tDNA片段,岛位于两者之间。尽管有些岛屿使用丝氨酸重组酶家族的整合体,但只有酪氨酸重组酶家族的那些靶向tDNA。在下面的内容中,我们将术语“整合”仅限制为酪氨酸重组酶家族的成员。
我们的算法Islander(11)搜索上述的岛签名,以发现包含整数基因(integrase gene)并靶向tDNA attB的基因组岛,我们的网站( http://bioinformatics.sandia.gov/islander )展示了其结果。 。 尽管这遗漏了一些孤岛,但发现的孤岛却以单核苷酸精度进行了映射。 它们还具有推定的活性,具有整合酶基因并且两个att位点均完整。 尽管古细菌与细菌有很多区别,但它们也带有基因岛,许多都符合我们的标准。
基因岛是在原核染色体中发现的多基因可移动DNA单元(12)。有些包含病毒结构基因集或结合基因集,表明它们在细胞之间的迁移方式。染色体位点特异性是由整合酶决定的,绝大多数是酪氨酸重组酶家族的基因,该基因通常在岛上发现。对于大约一半的岛,整合靶位点位于tRNA基因内(9,10)。尚不知道丝氨酸重组酶家族的整合子可指定tRNA基因,因此在下文中,术语“整合酶”仅限于酪氨酸重组酶家族的成员。
除了活动性和整合性的选择基因外,岛屿还可以携带有益于宿主的货物基因,从而促进诸如致病性和代谢谱等表型(8)。在整合进化过程中,由整合指定的靶DNA基因已频繁转换(10),从而促进了在给定宿主基因组中各种岛的组合积累。我们描述了用于识别tDNA整合,整合酶编码基因组岛的当前算法,并在Islander网站上从文献中介绍了其结果以及其他此类岛。
二、搜索策略
Islander网站收集靶向t(m)RNA的基因岛,使用Islander.pl软件包( http://bioinformatics.sandia.gov/software/ )识别的。 对先前描述的算法(13)的更改包括:六帧转换(six-frame translation)和Pfam域注释; 更精确地识别岛屿整合; 并将编码DNA序列(CDS)从使用RefSeq调用的所有蛋白质更改为仅编码Pfam域的蛋白质(图1)。

该算法通过每个复制子(replicon)进行以下操作:
-
查找tDNA。使用tRNAscan-SE(tRNA),BRUCE(tm-RNA),ARAGORN(两者)和rFind.pl(两件式tmRNA)鉴定tRNA和tmRNA基因(14-17)。 tFind.pl协调了这些工具的实现,根据需要更正了端点,并将带有CAT反密码子的tDNA分为异亮氨酸,引发剂和延伸剂蛋氨酸(18)。
-
查找integrases,不包括Xer和integron子类。复制子是六帧(six-frame)翻译的,氨基酸序列从终止密码子延伸到终止密码子。使用HMMER和来自Pfam的隐式马尔可夫模型(HMM)PF00589来识别候选整合基因(19)。 Xer蛋白在不同位点起作用,以解析二聚体染色体。它们也可以作为岛屿的整合体,但是在这些情况下,它们不会在岛屿中进行编码,并指定整合到dif部位而不是tDNA部位。排除了Xer蛋白,使用pfscan(20)并严格限制了HAMAP(21)文件中XerC,XerD,XerS和类似XerD的亚家族的蛋白。Integron integrases mobilize cassettes within integrons, but not canonical islands。使用由ACLAME系列Famint8(23)的MUSCLE(22)比对制备的HMM排除了它们。通过对ACLAME数据库中所有积分的测试,发现1.2e-24的阈值足以将整合子积分( integron integrases )与其他integrases分开。
-
找到tDNA片段。每个tDNA在其源复制子的BLASTN搜索中用作查询(参数:task,blastn; gapopen,0; gapextend,2.5;word size,7)。命中和查询基因定义了候选岛的端点,该候选岛顺序地经过了多个滤波器。
-
Integrase filter。不包含或不整合开放阅读框的候选岛将被拒绝。
-
CDS过滤。由于真实的tDNA及其岛状分裂片段不应与蛋白质编码基因的保守部分重叠,因此在六帧翻译序列中发现了Pfam-A结构域,而与CDS的结构域部分重叠的候选tDNA片段被排除在外。 作为例外,由于某些整合(例如,来自病毒Mx8和SSV的整合)延伸到了attP,因此允许整合域重叠(24,25)。
-
tDNA过滤。落入具有已知全长tRNA基因的BLAST片段。
-
长度过滤。短于2 kb或长于200 kb的候选人将被拒绝。一些合法的岛屿,例如通过此过滤器,失去了611 kb的中生根瘤菌共生岛(26)。
-
内部tDNA片段过滤。整合分离出tDNA末端片段。因此,拒绝了tDNA片段位于完整tDNA内部的候选岛。在3’端有一个例外,因为某些岛在tDNA片段中有一个小缺失,位于鉴别位点上游3 bp(27)。为了检测这种受损的tDNA片段,我们允许BLAST命中仅扩展到该缺失位点。
-
Configuration filter。整合将tDNA片段移至一侧。 tDNA下游5’片段或上游3’片段的候选岛被拒绝。
-
Orientation filter。带有与片段相反方向的tRNA基因的候选岛被拒绝。在分析的最后阶段,该拒绝步骤可用来衡量应在每个方向上以相同频率出现的候选人中的误报率。
-
Resolve。最后,将多个剩余候选岛共享相同tDNA的情况解析为单个候选岛,除非可以辨认串联阵列,其中每个阵列成员都有自己的整合酶和tDNA片段。
三、ISLANDER WEBSITE DESCRIPTION
Islander网站是根据2031个完整细菌基因组(包括1640个质粒和6个噬菌体)和137个完整古细菌基因组(包括75个质粒)以及1711个附加细菌质粒和543个噬菌体以及44个附加古细菌产生,截止2012年11月,从RefSeq的四个目录(古细菌,细菌,质粒和病毒)下载了38种弓形病毒,它们拒绝了真核病毒和质粒。 Islander算法产生了3927个独特的岛,尽管在发现每个都是假阳性的证据后,病毒中发现的所有七个岛和最小的岛上标记的质粒中的一个都被排除在外。表1列出了3919个最终基因组岛的一些统计数据。在大多数基因组中,至少发现了一个岛。在任何基因组中发现的最高数量是19(Desulfovibrio magneticus RS-1)。如前所述(11),tmRNA基因在整合目标中的富集程度高于任何tRNA异源受体类型。
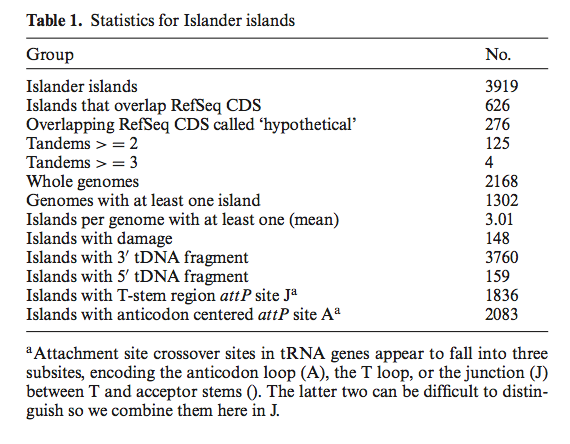
下拉菜单显示成千上万的基因组和孤岛,显得很尴尬。我们使用搜索文本框,还为交互式元基因组分类法查看器Krona(28)修改了javascript,以便能够导航到各个岛页面,同时还提供了数据库中岛的系统发育分布的可视化描述。 (图2)。
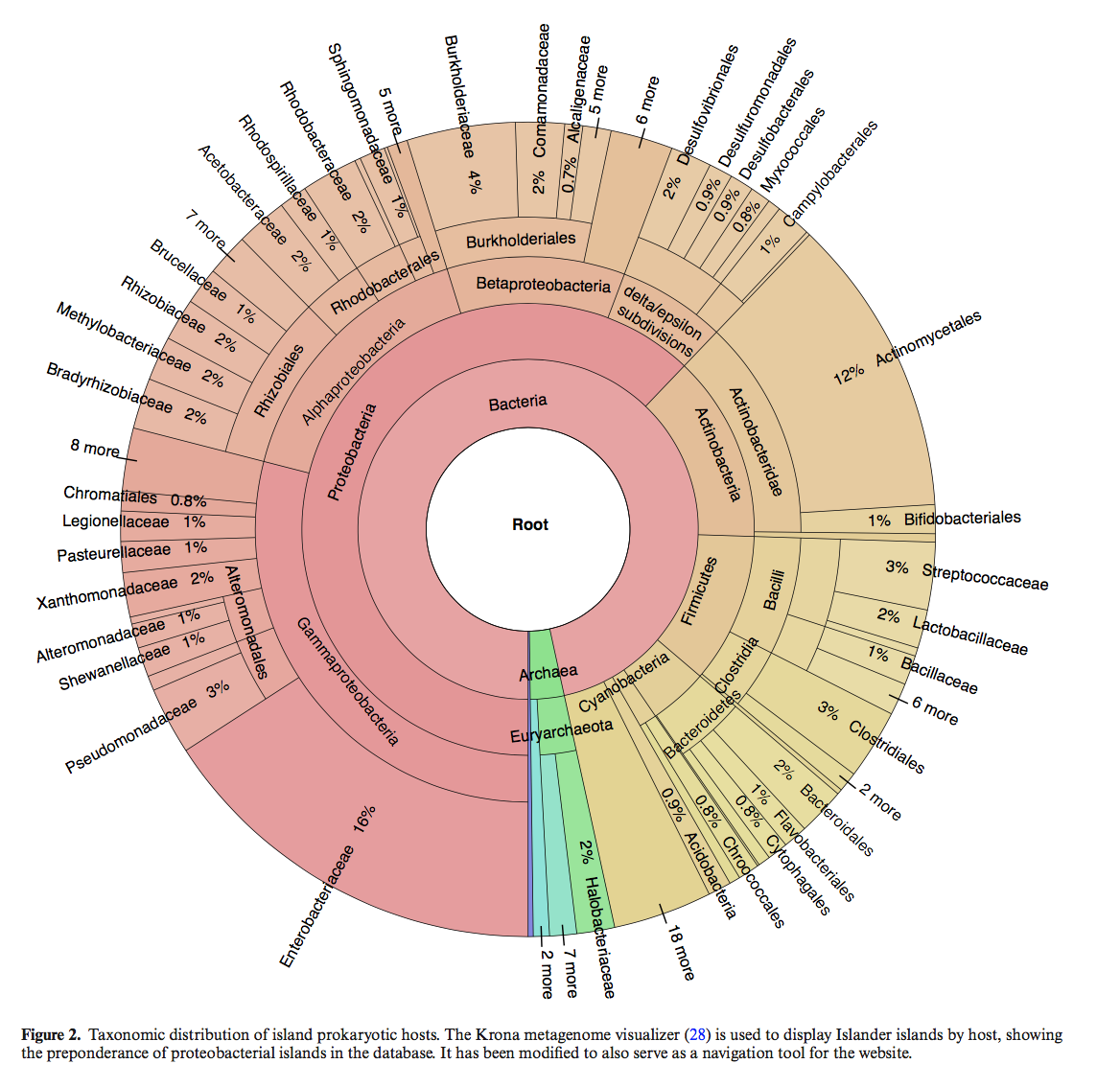
四、与正交方法的比较 COMPARISON WITH ORTHOGONAL METHODS
我们的算法主要通过目标岛来检测岛屿,而其他正交方法则寻找噬菌体样的基因组含量或异常的核苷酸组成。我们在研究中使用PHAST(29)评估了整个基因组,以识别类似噬菌体的区域,并使用Alien hunter(30)评估了组成偏向的nd区域。图3A显示了Islander(占总基因组长度的2.34%),PHAST(占1.70%)和Alien hunter(占19.3%)对基因组的碱基对覆盖率,以及它们的重迭之处。一个关键的重叠是PHAST呼叫涵盖了Islander岛的DNA的17.6%;其余的PHAST区可能表明在tDNA以外的位点有噬菌体,或者丢失了整合酶基因或tDNA片段。尽管Alien hunter 地区的基因组覆盖率很高,但在Islander islands却很丰富。
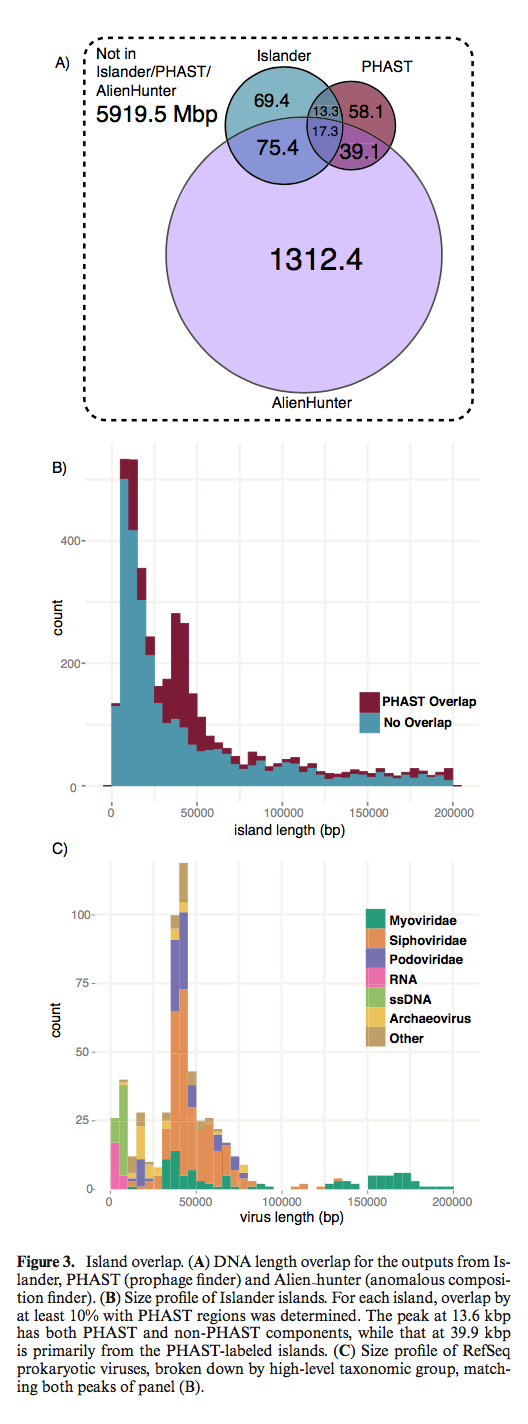
图3B示出了Islander islands分布。有明显的峰值集中在13.6 kbp和39.9 kbp,主要是由于PHAST重叠的岛。这些峰均出现在RefSeq原核病毒的大小文件中,我们在图3C中显示了被病毒系统发育分解的趋势,并具有有趣的趋势。 (请注意,大小文件都不一定代表自然世界中岛屿或病毒的频率;每种大小都受研究人员对基因组测序的选择所影响。)RefSeq文件最适合使用PHAST的岛屿文件中的部分重叠(图3B中的红色部分)。 40 kbp的峰富含Podoviridae和Siphoviridae。在非PHAST岛中,40 kbp的峰值可忽略不计(图3B中的蓝色部分),表明PHAST至少是在这个大小范围内,发现了大多数 Islander islands被预言。如果Islander islands中几乎所有的自发性噬菌体都具有引人入胜的优势,那就是PHAST发现的1119个岛上的遗留物,其中约有71%的岛民群岛的机动性必须通过其他方式加以解释,也许是PHAST不可检测的卫星噬菌体或通过缀合。 PHAST重叠岛在13.6 kbp峰中也富集,但比40 kbp峰中的富集少,并且该峰内PHAST和非PHAST成分之间可能存在相对移动。该峰不能由图3C所示大小范围内的单链DNA病毒解释,因为它们均未编码整合子。
五、网站更新
自该网站上次发布以来(13),已处理的islands数目从143个增加到3919个,全基因组从106个增加到2168个。我们的算法已如上所述进行了更改。我们打算让Islander成为准确映射基因组岛的黄金标准存储库,因此目前正在与一些误报率作斗争,这主要是由于放宽了我们的CDS过滤器。
我们的Islander命名约定采用属名的第一个字母(不包括念珠菌)和物种名称的前两个字母,在具有相同的三个字母昵称的可区分菌株中添加序列号,然后添加岛长以kbp为单位,并为集成站点指定一个字母名称。例如,E. coli O157:H7 str. Sakai (Eco661) 中的49 591 bp岛,整合到tRNA-Ser基因中被称为Eco661 50S。
此外,更新后的网站还标记了与PHAST调用重叠的推定孤岛,报告了复制子中的所有整合,附加的匹配tDNA片段,孤岛的基因列表和孤岛序列。
更多资料
- http://www.pathogenomics.sfu.ca/islandviewer2/download.php
- https://bioinformatics.sandia.gov/islander/
- https://bioinformatics.sandia.gov/software/index.html
- https://card.mcmaster.ca/
- http://www.pathogenomics.sfu.ca/islandviewer/browse/
参考资料
- Hudson, C.M., B.Y. Lau, and K.P. Williams, Islander: a database of precisely mapped genomic islands in tRNA and tmRNA genes. Nucleic Acids Res, 2015. 43(Database issue): p. D48-53.
