【7.1.2】 微生物天然产物数据库
数字革命正在推动人们存储、分发和使用信息的方式发生重大变化。随着关联数据、机器学习和大规模网络推理等新技术的出现,天然产物研究领域开始拥抱数字化实验数据的实时共享和大规模分析。数据库在这方面起着关键作用,因为它们允许对基本和高级应用程序的数据进行系统注释和存储。这些数据库的内容、结构和可访问性的质量都有助于它们在实践中对科学界的有用性。本综述涵盖了过去十年(2010-2020 年)与微生物天然产物发现相关的数据库的开发,包括化学结构/特性、代谢组学和基因组数据(生物合成基因簇)的存储库。它概述了最重要的数据库及其功能,重点介绍了使用此类数据库的一些早期元分析,并讨论了实现数据库之间广泛互操作性的基本原则。此外,它还指出了天然产物数据库的管理和使用中的概念和实践挑战。最后,评论结束时讨论了该领域向前发展所需的关键行动点,不仅适用于数据库开发人员,也适用于活跃在该领域的任何科学家。
一、 简介
1.1. 天然产物数据管理简史

- 一个代表性示例是制药公司 Lederle Laboratories 于 1960 年代初开发的系统.
- 在 80 年代,一些重要的电子资源开始出现。CAS 和 Beilstein 开始开发已成为 Scifinder 和 Reaxys 的大型文献数据库
- Hartmut Laatsch 教授创建了 AntiBase,8微生物天然产物数据库,而约翰·布朗特教授和默里·芒罗教授创建了 MarinLit,这是一个关于海洋天然产物的文章数据库
1.2. 天然产物发现的新时代
2010 年代初期的标志是新工具的出现,使“普通”天然产物科学家可以使用以数据为中心的方法;一个没有接受过专门的编程或计算机科学培训的人。此类工具的示例包括 NaPDoS 和 eSNaPD,用于评估微生物菌株的生物合成多样性,FuSiOn 用于从头预测复合作用模式,以及 iSNAP ,用于从质谱数据中去除非核糖体肽。
对采用新数据技术产生重大影响的一种工具是 antiSMASH。antiSMASH 于 2011 年首次发布,提供了一个简单、可免费访问的 Web 界面,用于从基因组序列数据中识别生物合成基因簇 (BGC)。天然产物社区很快认识到这种分析可以为其研究计划的许多方面带来的力量,antiSMASH 成为许多天然产物计划的主要工具。antiSMASH 不需要学科专家手动扫描原始序列数据,而是为用户提供了一种简单的机制来生成初始自动注释,然后可以优先考虑进一步调查。这种新资源的可访问性和功能为天然产物工具的开发奠定了基调,并产生了对新工具的直接需求,这些工具将在天然产物的其他领域提供相同水平的功能。
二、 微生物天然产物研究数据库
2.1. 化学结构和性质数据库
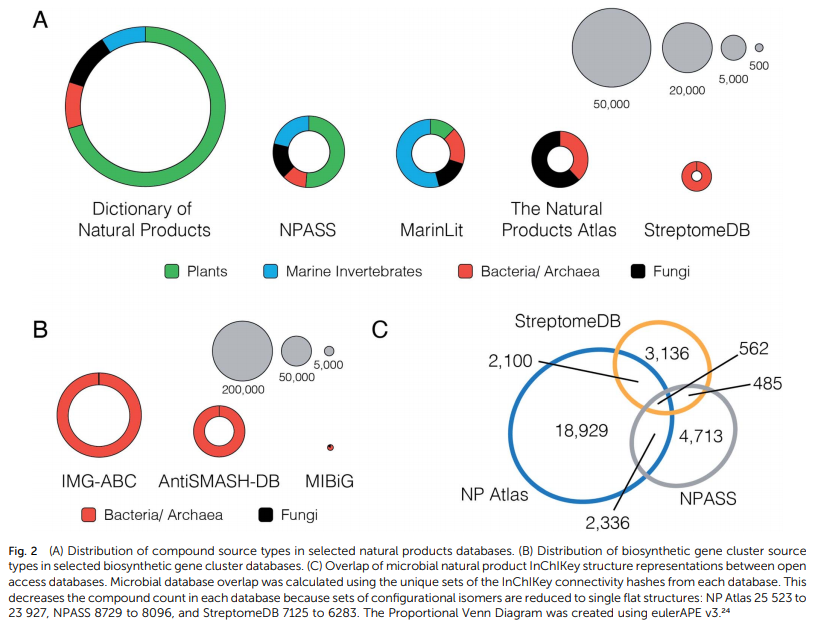
天然产物结构数据库的当前格局高度分散。Sorokina 和 Steinbeck 最近的综合评论23列出了自 2000 年以来开发的 122 种天然产物结构资源,数量惊人。该列表包括商业和非商业资源库,涵盖范围广泛的生物源和地理位置。然而,尽管可用的天然产物数据库很广,微生物天然产物科学家的选择却出人意料地有限。在 122 个资源中,有 50 个允许访问完整的结构集。其中,11 个包含细菌天然产物的条目,只有三个(NPASS、StreptomeDB 和 Natural Products Atlas)允许按分类来源过滤以仅提取微生物衍生的化合物。因此,这三种资源目前代表了有关微生物天然产物结构的最佳免费信息来源
-
NPASS ( http://bidd.group/NPASS/ ) 是最近开发的天然产物数据库 (2018),旨在提供天然产物的来源生物和生物活性。它部分涵盖了来自多种分类来源的天然产物的化学空间,包括植物、无脊椎动物和微生物。它总共包含 35 032 种化合物,其中大约 9000 种源自微生物
-
StreptomeDB ( http://www.pharmbioinf.uni-freiburg.de/streptomedb3/ ) 是一个专门针对细菌属链霉菌属的目标数据库。最近于 2020 年更新,它包含 7125 种化合物和源生物信息,以及一些生物活性和光谱数据。
-
Natural Products Atlas ( https://www.npatlas.org/ ) 是一项新资源 (2019),旨在全面覆盖所有微生物衍生的天然产品结构。它目前包含 25 523 种化合物 ( v2019_12 ) 并且正在积极开发中。它具有与其他两种天然产品资源的双向链接;生物合成基因簇的 MIBiG 数据库和天然产物质谱的 GNPS 数据库。 除了开源数据库,还有一些高质量的商业平台。其中,天然产物词典 (DNP)、MarinLit 和 AntiBase 是最完善的,尽管 AntiBase 上次更新是在 2014 年。这三个数据库都很大(> 30 000 种化合物)并且包含丰富的元数据。它们对已发表的文献有广泛的覆盖,并且通常非常准确。但是,它们的年度订阅成本很高,并且不允许将结构数据或其他信息批量导出到外部应用程序。这限制了它们对个人搜索的效用,并排除了它们与其他基于天然产物的数据资源的集成。
-
DNP ( http://dnp.chemnetbase.com/ ) 包含超过 290 000 个条目(2020 年 2 月访问),包括来自所有主要来源生物群的天然产物,以及物理化学和生物数据。该数据库通过学科专家的广泛手动管理过程不断更新,以确保高数据质量标准。然而,基于化合物名称对数据集的抽查表明覆盖率并不普遍,即使对于一些众所周知的化合物类别(例如abyssomicins)。
-
MarinLit ( http://pubs.rsc.org/marinlit/ ) 是海洋天然产物的文献数据库,包括结构、分类学和 35 015 种化合物的全合成报告(2020 年 2 月访问)。它包括来自无脊椎动物和藻类的化合物,以及来自海洋微生物的 8082 种化合物。令人印象深刻的是,该数据库几乎每天更新,使其成为该领域最现代的资源。
-
抗生素及相关物质辞典 抗生素及相关物质词典28是一份超过 2000 页的参考文本,列出了所有已知的天然抗生素物质 (>10 000)。它最近从 1980 年代的原始版本更新(2013 年),现在包括来自 BMIC 数据库的许多条目,该数据库由 Janos Berdy 博士维护多年,并且是抗生素化合物手册的基础数据库。随附一张可搜索的 CD-ROM。
如第 1.1 节所述,还有许多来自生物技术和制药公司的天然产物数据库。不幸的是,其中许多是很难获得的,如果不是不可能的话。大多数都没有在积极开发中,并且仅以物理格式或遗留数据库结构存档。尽管一些公司愿意向更广泛的社区发布这些数据,但实际挑战可能会阻止访问,例如完成责任免除文件;法律部门通常优先级较低的任务。
最后,值得一提的是两个最大的化学文献数据库的天然产物覆盖率;Scifinder 和 Reaxys。这两个平台都包含来自天然产物文献的大部分化合物。然而,除了简单的结构搜索之外,它们都不是特别适合基于天然产物的查询。Scifinder 不包含任何将化合物识别为天然产物的标志,因此无法将天然产物与合成化合物分开。Reaxys 确实包含术语“从天然来源中分离出来”,但许多已知的天然产品没有标注此标志,这意味着使用此过滤器执行的搜索并不全面。
2.2 生物合成基因簇数据库
随着 BGC 发现的速度在 2000 年代初期开始加快,生物合成界面临着许多与 30 年前天然产物结构阐明界遇到的相同挑战。特别是,关于 BGC 发现的信息越来越分散在科学文献中,或者以结构化程度较低的方式存储在 NCBI GenBank 等基因组数据库中。与基于结构的发现一样,这限制了资源之间交叉链接的可能性,并阻止了利用内部知识的可编程访问。为了解决这个问题,已经开发了几个 BGC 数据数据库。
-
ClusterMine360 ( http://clustermine360.ca )于 2013 年推出, 是首批冒险从事对已知产品经过实验验证的 BGC 信息进行编目任务的平台之一。专注于非核糖体肽 (NRP) 和聚酮 (PK) 类,它包含 300 个与其化学产品相关的 BGC。虽然最初准备通过用户提交的注释进行持续扩展,但似乎数据库覆盖的 BGC 总数自最初发布以来并没有显着增加。
-
ClusterMine360 大约同时发布,DoBISCUIT 30 ( https://www.nite.go.jp/en/nbrc/genome/dobiscuit.html ) 发布了 72 个已知 PK BGC的初始集合。不幸的是,该数据库已无法访问,尽管其主页仍处于活动状态,并显示了 2016 年 12 月 27 日记录的 108 个 BGC 的最终日志。
-
MIBiG 在2015年,有超过150名的天然产物的科学家协同努力导致最小信息的有关生物合成基因簇(MIBiG)数据标准和信息库的出版物( https://mibig.secondarymetabolites.org )和实验表征的 BGC。MIBiG 拥有超过一千个特征 BGC 的信息,很快被社区用作 BGC 数据的中央参考数据库。值得注意的是,antiSMASH 自动将每个检测到的 BGC 与来自 MIBiG 的所有参考基因簇进行比较。首次发布四年后,即 2019 年,宣布了数据库和模式的第二次迭代,突出了累计 2021 个 BGC 条目及其在线存储库基础设施的重大改革。MIBiG 仅包含经实验验证负责生产一种或多种已知天然产物的 BGC。MIBiG 条目还受到开发人员和科学界的广泛手动管理和注释,进一步提高了该存储库中的信息内容和数据质量。
-
IMG-ABC 利用联合基因组研究所 (JGI) 广泛的细菌基因组平台 IMG/M,IMG-ABC 33 ( https://img.jgi.doe.gov/cgi-bin/abc/main.cgi ) 集是已知(间接来自 MIBiG)和计算预测的细菌 BGC 的最全面和功能最丰富的数据库。在 IMG-ABC v5 之前,该数据库包含总共超过一百万个使用 antiSMASH 和 ClusterFinder 算法预测的 BGC。34后一种方法已被弃用,取而代之的是对 antiSMASH 5 进行更严格但更“高可信度”的 BGC 类检测。这导致 IMG-ABC 提供的 BGC 总数下降,截至目前有 410 558 个 BGC 可用2020 年 6 月 29 日。需要注意的一个重要细节是,由于 JGI 的数据使用政策 ( https://jgi.doe.gov/user-programs/pmo-overview/policies/ ),不建议对数据进行批量分析和发布IMG-ABC 的一些基因组数据可能仍处于禁运状态。在未来,我们建议 IMG/M(和 IMG-ABC)应该跟随他们的真菌基因组数据库对应物 MycoCosm ( https://mycocosm.jgi.doe.gov/ )的脚步,35提供一个简单的过滤禁止基因组,从而能够“安全”批量下载和分析其数据。
-
antiSMASH 数据库 (antiSMASH-DB) ( https://antismashdb.secondarymetabolites.org ) 最初由开发 antiSMASH 的同一团队于 2016 年发布,用作预计算 antiSMASH 运行的中央存储库。与 IMG-ABC 相比,antiSMASH-DB 旨在提供一个有限的、去重复的、来自最高质量细菌基因组的推定 BGC 列表。对于一组高度相似的基因组(例如,只有少数单核苷酸多态性的数千个大肠杆菌基因组),已经挑选了代表,而不是单独提供所有菌株的结果。一个关键的理由这样做是提供antiSMASH无缝集成通过它的“ClusterBlast”模块对每个检测到的 BGC 与数据库中的 BGC 进行序列比较。继 2018 年第二次发布后,antiSMASH-DB 包含从 NCBI RefSeq 数据库的 24 776 个细菌基因组(其中 32 548 个 BGC 来自 6200 个完整基因组)预先计算的总共 152 106 个 BGC。38即将发布的第三个版本也将包括来自高质量真菌基因组的 BGC
2.3. 代谢组学和分析化学数据库
过去十年中出现了许多用于共享和分析代谢组学数据的资源。其中许多资源集中在数据的公平共享上,以实现更高效的天然产物发现,并且不限于微生物天然产物科学的范围。
-
GNPS (The Global Natural Products Social molecular network) ( https://gnps.ucsd.edu/ ) 系统是一个用于共享和分析串联质谱数据的生态系统。它建立在 MassIVE 平台上,并具有令人印象深刻的内部连接工具套件。它还提供完整的数据生命周期管理功能,从数据采集到发布。最流行的功能之一是分子网络,它可以实现 MS/MS 实验光谱之间的可视化关系。在 GNPS 中提交用于分析的数据被组织成数据集,这些数据集可以是私有的,也可以是公开的。迄今为止,在线提供了 1413 个公共数据集(访问时间为 2020 年 2 月 24 日)。此外,GNPS 拥有许多公共 MS/MS 谱库,其中包含 74 130 个带注释的谱图。
-
MetaboLights ( https://www.ebi.ac.uk/metabolights/ ) 是 EMBL-EBI 运行的数据库,最初创建于 2012 年,并于 2019 年进行了大修。它是一个代谢组学数据的数据库,具有存储和报告各种数据类型,包括 NMR、GC/MS、LC/MS,以及代谢物结构、它们的参考光谱和生物学作用。MetaboLights 是基于 FAIRsharing 计划 [ https://fairsharing.org/biodbcore-000168/ ]的许多期刊的代谢组学数据的推荐存储库。
2.4. 核磁共振代谢组学
McAlpine等人最近的综合评论。确立了天然产物领域的核磁共振去复制状态。审查表明,仍然迫切需要对天然产物的 NMR 数据进行全面和开放的数据交换。这篇综述发表后,美国国家补充和综合健康中心和美国 NIH 膳食补充剂办公室发起了一项开发此类资源的提案征集。这一呼吁导致在 2020 年建立了天然产物磁共振数据库 (NP-MRD;www.np-mrd.org ),旨在为天然产物结构的实验和计算光谱创建一个开放访问存储库。
除了这项新举措之外,还有许多当前的数据库和工具已经通过实验和预测 NMR 光谱解决了这个问题。
-
NAPROC-13 ( http://c13.usal.es/ ) 是一个包含超过 6000 种天然产物化合物的13 C NMR 谱的数据库。该数据库有一个网络界面,可以快速识别复杂混合物中存在的化合物,并提供对新结构阐明有用的结构信息。
-
NMRshiftDB ( https://xn--nmrshidb-vs49b.nmr.uni-koeln.de/ ) 包含许多与 NAPROC-13 相似的特征以及来自其他核的 NMR。然而,它并不是天然产物化学所独有的。
-
BMRB (Biological Magnetic Resonance Data bank) ( http://www.bmrb.wisc.edu/ ) 包含来自蛋白质、肽、核酸和其他生物分子的各种实验和模拟 NMR 数据。BMRB 不仅限于微生物天然产物,还包含来自天然产物和代谢组学所有领域的数据。BMRB 还维护着一个 NMR 脉冲序列库和用于生物分子 NMR 的计算软件。
-
HMDB (Human Metabolome Database ) ( https://hmdb.ca/ ) 是一个开放存取的数据库,提供有关人体代谢物的详细信息,因此包括对人体微生物组至关重要的代谢物。许多代谢物还包含实验性 1D 和 2D NMR 谱,可免费下载。
-
CH-NMR-NP ( https://www.j-resonance.com/en/nmrdb/ ) 是由 JEOL 托管的数据库,其中包含从 2000 年到 2014 年的期刊列表中汇编的 NMR 数据。它包含来自大约 35 500 种天然产物的1 H 和13 C NMR 数据,并且不限于微生物天然产物。CH-NMR-NP 可在线搜索,并允许在逐个化合物的基础上下载 JEOL Delta 数据格式的 NMR 数据。
三、数据库管理和使用
3.1. 数据库用户面临的实际挑战
令人惊讶的是,在这一领域的资源之间进行数据比较仍然非常困难。化学结构和化合物名称是连接许多这些数据库的常用术语。原则上它应该是可能的关联数据从一个资源(例如生物合成基因簇)与来自另一(数据例如NMR或MS数据)经由化学结构。然而,在实践中,没有就化学结构提供一种独特的、机器可读的结构表示而不会丢失信息的标准化方法。例如,单个结构可能有多个 SMILES 字符串,标准 InChI 表示不保留有关首选互变异构体的信息,并且 MOL 文件是大块文本,难以以大多数数据库格式存储。这些问题意味着数据库通常在结构上很难对齐,而没有大量额外的手动管理。
复合名称同样具有挑战性。标点符号的微小变化、特殊字符的包含和编码,或者文献中许多化合物没有琐碎的名称,都会导致资源之间的重叠不足。由于为现有化合物分配新的同义词,以及偶尔将同一名称错误分配给多个结构,这进一步复杂化。更复杂的是,一些复合类收到几个不同的父名称,通常是为了增加新发现的可见性。相反,一些研究人员对从给定生物体中分离出的所有化合物使用相同的母体名称,而不管结构相关性如何。这两个问题都使基于琐碎名称的相关结构的分组复杂化。
一些资源已投入大量精力来提高互操作性。例如,天然产品图集和 MIBiG 团队手动审查了 MIBiG 数据库中的每个条目,并在每种情况下确定了适当的天然产品图集条目。这两个资源现在包括数据页面之间的双向链接,并提供列出每个平台中主键之间链接的可导出表。GNPS 平台也建立了类似的链接。
投资类似的努力以按结构调整其他关键资源可能会对新的跨学科发现工具的开发产生重大影响。UniChem 提供了一个有效的交叉引用系统的例子,48由 EMBL-EBI 建立的系统通过为每个唯一的化学结构分配一个 UniChem 标识符并将该标识符链接到所有数据库来连接多个数据库中的化学结构隶属于 UniChem 系统。
3.2. 数据库创建和管理的实际挑战
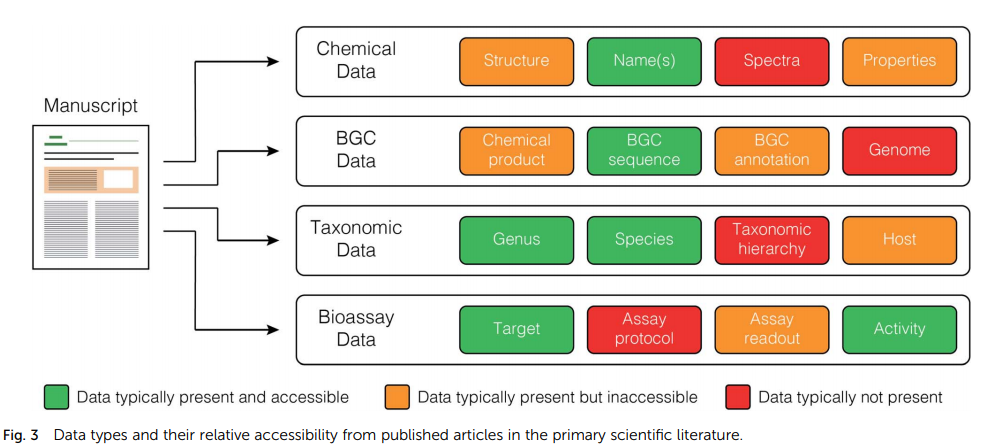
当前的发布模型不太适合大规模数据库的创建和维护。每个期刊都有自己的格式和数据要求,没有期刊产生包含关键原始数据的标准化、机器可读的文件(图 3)。相反,这些数据通常以各种格式作为补充材料提供。将数据保存到公共资源(例如使用 NCBI 保存生物合成基因簇)是有价值的,但仍然必须从论文的方法或数据可用性部分手动提取登录号,从而减慢了数据管理的速度。
.。。
3.3. 2020年微生物天然产物数据整理
从原始文献中挑选天然产物数据仍然主要是手动过程。它需要三个主要步骤;识别与微生物天然产物发现有关的文章,从每篇文章中提取结构、基因簇和其他数据,并将这些数据组织成结构化格式。其中最具挑战性的是相关文章的识别。传统上,超过 50% 的微生物天然产物发现发表在《抗生素杂志》或《天然产物杂志》上. 然而,随着天然产物研究范围的扩大,报告天然产物发现的场所数量有所增加。这给数据管理带来了挑战。手动检查所有已发表文章的标题和摘要现在是一项不可能完成的大任务。相反,策展工作必须依靠关键期刊的有针对性的策展,或使用关键字的文本挖掘策略从公共数据源(如 PubMed)中查找相关文章。这两种方法都有局限性,会影响策展工作的覆盖范围。专注于有针对性的期刊列表可以排除外围相关领域的报告(例如海洋化学生态学或微生物组研究),而文本挖掘方法可能会遗漏核心文章,并且容易产生偏差,具体取决于用于过滤的算法。作者可以通过确保在摘要中突出描述新的天然产物或 BGC 的发现来协助这项工作。在大多数情况下,策展人无法批量访问文章的全文版本,这意味着标题和摘要是唯一可用于文章优先级排序的信息。因此,在摘要中明确描述新化合物或 BGC 发现是确保新数据包含在管理工作中的最有效方法。
四、 数据库之间的集成和互操作
4.1.3. BGC 数据库的新用途 BGC
数据库最明显的用途之一是去复制过程:确定在一组(元)基因组序列中检测到的 BGC 是否可能编码已知的生物合成途径。例如,Crits-Christoph等人。73使用 MIBiG 数据库表明,他们在来自未培养的酸杆菌、疣状杆菌、Gemmatimonadetes 和 Rokubacteria 的宏基因组组装基因组中鉴定的 >90% 的 BGC 可能编码新途径。现在也可以使用 BiG-SCAPE 算法为大型基因组数据集自动化这种去复制过程。74 BiG-SCAPE 根据用户指定的 antiSMASH 结果和所有 MIBiG 数据库 BGC 计算序列相似性网络,并重建基因簇家族 (GCF),从中可以评估哪些 BGC 与来自 MIBiG 的已知 BGC 相似,哪些不相似。 BGC 数据库的另一个明显用例是注释功能,例如微生物组研究,并使用这些注释来推断生态相互作用。例如,巴赫拉姆等人。75使用一组 MIBiG 条目与具有经证实的抗菌功能的产品相关联,以评估真菌抗生素生产潜力是否与表土宏基因组中细菌抗生素抗性基因的频率相关。
此外,人们一直在使用 antiSMASH-DB 等 BGC 数据库来识别包含特定感兴趣基因组合的 BGC。例如,克劳斯等人。76进行了模式匹配,以绘制 antiSMASH-DB 中 PapR2 样调节剂(SARP 型 DNA 结合蛋白,具有作为沉默 BGC 的通用激活剂的潜力)的发生率和多样性,这揭示了其在放线菌基因组中的广泛分布。
BGC 数据库的另一个直接用途是绘制更大分类群内生物体的生物合成多样性。77 antiSMASH-DB 等数据库通过提供可通过应用程序编程接口 (API)访问的即用型、预先计算的 BGC 数据和元数据(例如,关于它们的分类学起源)使这些分析变得简单明了。
最后,BGC 数据库也有潜力用作使用合成生物学进行通路工程的“部分目录”。例如,ClusterCAD 软件78允许用户通过从 MIBiG 采购聚酮 BGC 和聚酮合酶模块来设计新的模块化聚酮合酶装配线,并提供图形界面来混合和匹配这些以构建感兴趣的新型聚酮结构。原则上,这种类型的计算机辅助设计可以通过多种方式扩展,例如通过从 IMG/ABC 或 antiSMASH 数据库中的公开可用数据中获取和搜索任何 BGC,或者通过例如包括搜索编码定制酶的基因.
五、未来展望
以数据为中心的方法从根本上改变了自然科学许多领域的格局。例如,从 1960 年代费力地早期确定蛋白质晶体结构,蛋白质生物化学已经发展到一个复杂的领域,即使是非专家也可以针对几乎任何生物目标对虚拟文库进行大规模的自动对接研究。同样,长期以来将 KEGG 创建为基因功能百科全书83的努力使得能够开发用于跨基因组和宏基因组(例如BlastKOALA 和 GhostKOALA 84)自动注释基因功能的工具。
天然产物科学尚未充分利用这一不断变化的科学发现格局。许多发现计划仍然专注于手动方法,没有有效地利用该领域的先验知识。化合物重新发现的高比率和“不寻常”BGC 的异源表达证明了这一点,这些 BGC 产生了众所周知的化合物类别。虽然这不能总是避免,但更好的化学结构数据、基因组数据和代谢组学数据的数据集成具有明显的潜力,可以提高研究工作的优先级。
开发新的数据驱动发现方法提供的机会是显而易见的。然而,期望参与工具开发的研究人员也将创建支持这些工具所需的基础数据集是不合理的。相反,我们必须投入资源来创建大型、结构良好的关键信息存储库,并且必须发展一种文化,将新结果的数据存储作为发现工作流程的标准和预期部分。如果我们能够实现这些目标,自然产物科学的每个角落都会强烈感受到这项投资的回报
参考资料
- Microbial natural product databases: moving forward in the multi-omics era。 https://pubs.rsc.org/en/content/articlelanding/2021/NP/D0NP00053A
