【7.5.2】微生物网络构建原理--SparCC/MENA/LSA/CoNet/SPIEC-EASI
原文链接: Microbial association network construction tutorial。 https://psbweb05.psb.ugent.be/conet/microbialnetworks/index.php
下面对PPT的内容进行简单介绍。核心内容如下:

一、背景
微生物之间的共现(Co-occurrence)可能有以下几种原因,他们可能具有一定的生态关系,或者在生态位上有重叠。
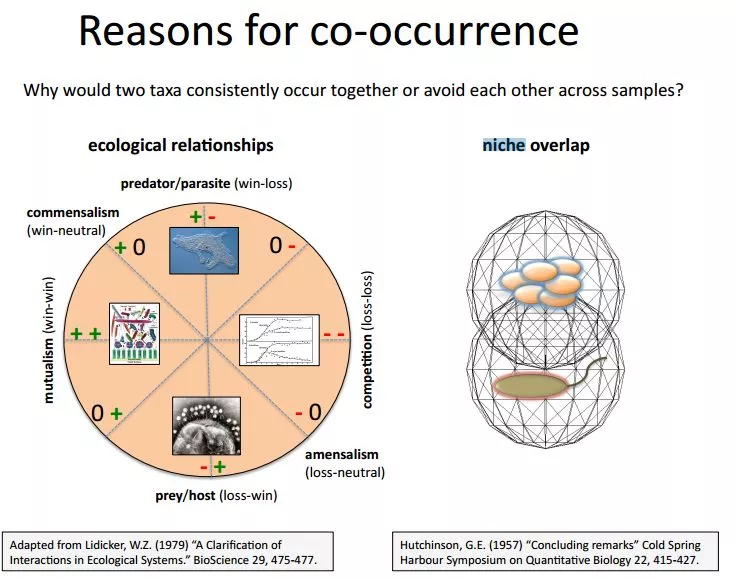
物种的关系可能是此消彼长、或者共增共减。
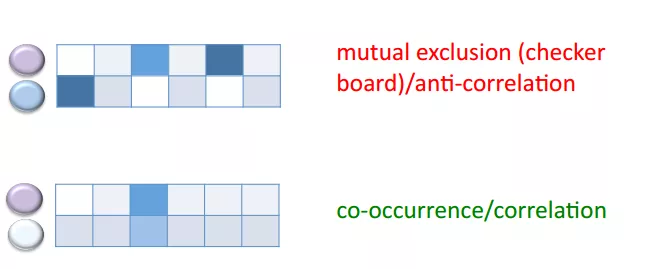
二、实现网络的技术及存在的问题
2.1 基于相似性
目前有两种实现网络的技术,第一种是基于相似性。
不管是基于abundance还是incidence的数据,都可以计算成对物种之间的相似度矩阵,并随机化数据反复计算。 考察实际相似度与打乱后相似度的显著性差异。 最后保留具有意义的相似度并可视化。

其存在的问题包括:
1.双零问题(double zeros):
微生物数据存在很多的0值。但是我们不知道该物种是低于检测限还是真的不存在。
因此当存在很多0,就会得到很高的相似度。
如下表所示,增加了0值后相似度显著的升高了。
因此对于双零值,算法中要避免得到很高的相似度。
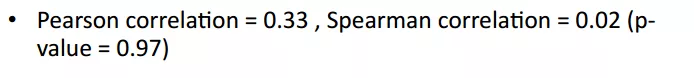
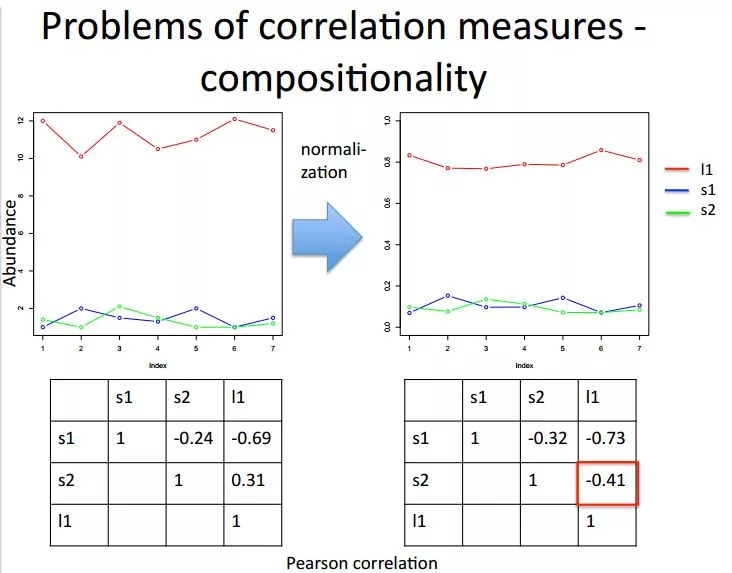
2. 群落组成的问题:
由于每个样本测序深度不同,因此即使物种个数相同,所占比例也不同。因此需要标准化,可以用每个样本物种的个数除以总样本物种的个数;或者重抽到相同深度。
另外pearson和 spearman考虑的是绝对值,因此标准化后会带来很大的偏差。而基于比例或者对数比例(log-ratio)的方法不受数据组成的影响,因为标准化后数据之间的比例不会变。具体如下图所示,标准化后pearson相关性改变了。
2.2 基于回归
第二种实现网络的技术是基于回归。将物种划分为source和target,使用多元回归计算物种之间的关系。
也是要随机化数据重复计算。根据实际回归系数与随机得到的回归系数的关系进行判定。
其优点在于可以检测多个物种的关系;并可以预测不对称的关系(如偏利共生)。
缺点在于会出现假阳性、过拟合,且难以可视化。

三、实现网络的工具
3.1 SparCC
SparCC使用对数比例的方差来计算物种之间的相关性。
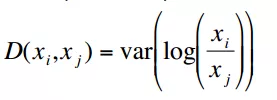
SparCC 对观测到的数据拟合狄利克雷分布,对物种的比例及相关性计算迭代计算多次。最后得到的相关性为分布的中位数。P值采用bootstrap方法计算。
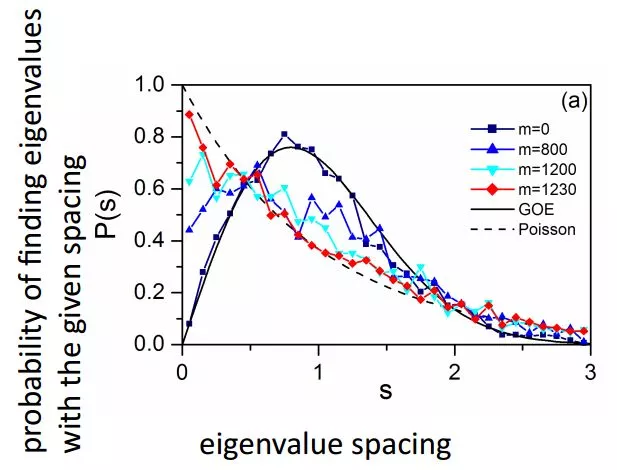
3.2 MENA
Molecular ecological network analysis pipeline

其核心在于随机矩阵理论(Random matrix theory, RMT)。
计算给定阈值的pearson相关矩阵的特征值间距分布;
对于整个阈值范围都进行计算;
保留分布由高斯分布变为泊松分布的阈值;
保留阈值以上的所有相关性。
3.3 LSA
Local similarity analysis
计算时间序列之间的相似性。由于考虑了滞后效应,可以得到直接连接和不直接连接的边。这种方法在海洋和湖泊宏基因组中很受欢迎。基本计算原理和基于相似性的网络相同,只是将相似性按照时间进行了分割。
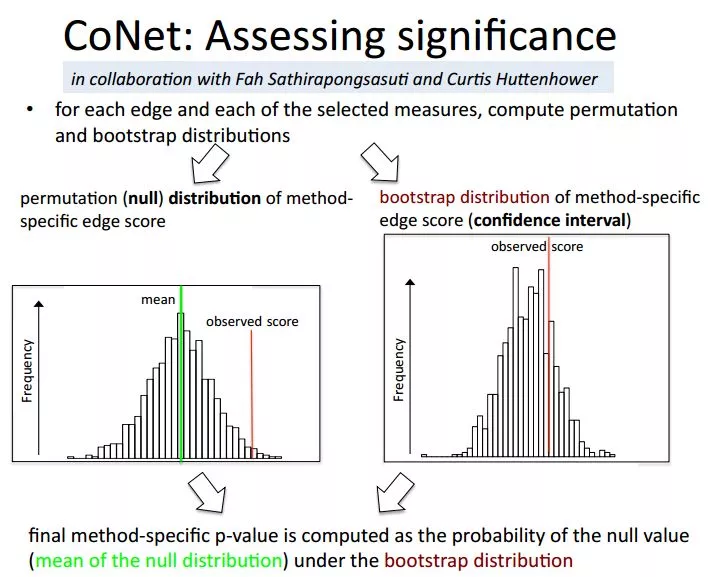
3.4 CoNet
基于组合效应(Ensemble-based)的网络
不同相关性计算方法(pearson,spearman,bray-curtis)可表达不同的关系,但是随着阈值的增加彼此的结果会趋同。
不同方法也会存在不同的错误,但是对于正确的结果却是一致的。
因此可将不同方法结合起来。

对于每条边和每种方法,分别计算permutation和bootstrap分布。两者相比较得到最终的P值。
四、讨论
最后总结一下,四种方法各有特色:
MENA强项在于阈值的算法,且不需要人为设定阈值。
Sparcc特色在于相关性的计算方法。
LSA引入了时间序列。
CoNet将多种相关性综合考量。
参考资料
