【7.2.1】 天然产物域搜索器--NaPDoS
Natural Product Domain Seeker (NaPDoS)
在线工具; https://npdomainseeker.sdsc.edu/
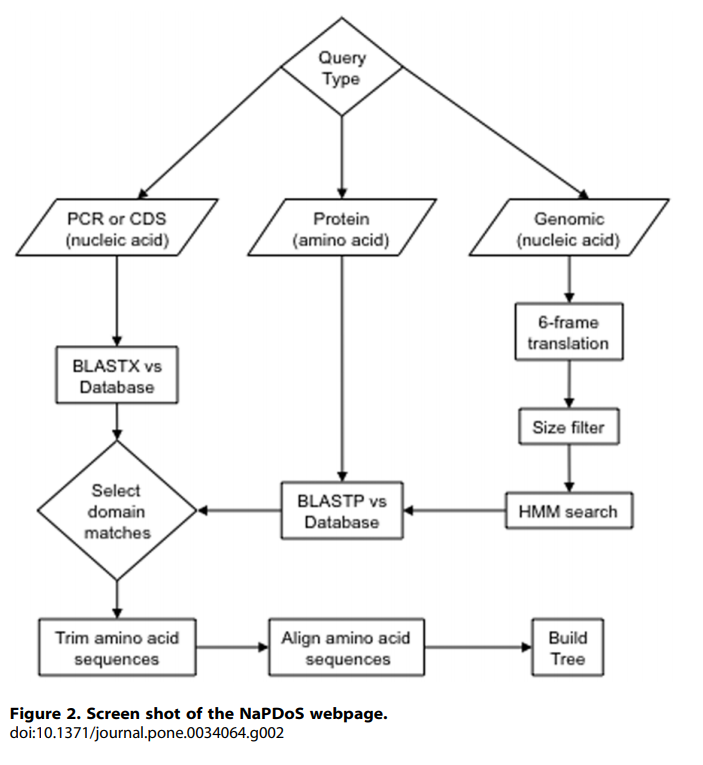
NaPDos 是一种用于快速检测和分析次级代谢产物基因的生物信息学工具。 该工具旨在从 DNA 或氨基酸序列数据中检测和提取 C 域和 KS 域,包括 PCR 扩增产物、单个基因、全基因组和宏基因组数据集。
候选次级代谢物结构域是通过与来自充分表征的化学途径的大量人工策划的参考基因进行序列比较来确定的。 候选基因序列被提取、修剪、翻译(如有必要)并进行域特异性系统发育聚类以预测它们的推定产物可能是什么,并确定这些产物是否可能产生与先前已知的生物合成途径相似或不同的化合物 .
- 它提供了一种自动化方法来评估次级代谢物生物合成基因的多样性和菌株或环境的新颖性
- NaPDoS 分析基于分别源自聚酮化合物合酶 (PKS, polyketide synthas) 和非核糖体肽合成酶 (NRPS,non-ribosomal peptide synthetase ) 基因的序列标签的系统发育关系。序列标签对应于 PKS 衍生的酮合酶域和 NRPS 衍生的缩合域,并与实验表征的生物合成基因的内部数据库进行比较
- NaPDoS 提供了一种从 PCR 产物、基因组和宏基因组数据集中提取和分类酮合酶和缩合域的快速机制。紧密的数据库匹配提供了一种推断次级代谢物广义结构的机制,而新的系统发育谱系为发现新的酶结构或次级代谢物组装机制提供了目标
三、背景介绍
3.1 前言
基因组测序表明,即使是经过充分研究的细菌,其次级代谢产物的潜力也被严重低估[1]、[2]。这一发现引起了人们对基因组挖掘作为天然产物发现方法的兴趣的爆炸式增长[3]、[4]、[5]、[6]、[7]、[8]。考虑到天然产物仍然是治疗剂的主要来源之一[9] , [10],序列分析提供了在化学分析之前识别具有最大遗传潜力的菌株的机会,以产生新的次级代谢物,从而提高发现新药物先导物的速度和效率。此外,群落或宏基因组分析可用于识别具有最大次生代谢潜力的环境,并解决与次生代谢相关的生态问题。为了利用这些机会,开发新的生物信息学工具来处理由下一代测序技术产生的大量序列数据至关重要[11]。
聚酮化合物合成酶 (PKS) 和非核糖体肽合成酶 (NRPS) 是大型酶家族,可用于许多临床重要的药物制剂。这些酶采用互补策略,使用装配线工艺从相对简单的羧酸和氨基酸构建块中依次构建多种天然产物[12]、[13]。PKS 和 NRPS 基因的分子结构已被详细审查,最低限度包括激活(AT 或 A,activation )、硫醇化(ACP 或 PCP,thiolation )和缩合(KS 或 C,condensation)结构域,分别[14]、[15]、[ 16]、[17]、[18]. 这些基因是微生物基因组中发现的最大基因之一,并且可能包含高度重复的模块,这对准确组装和随后的生物信息学分析造成了相当大的挑战[8]。
当与 PKS 和 NRPS 基因组装相关的挑战可以克服时,已经开发了许多有效的生物信息学工具用于域解析[19]、[20]和域字符串分析[21]、[22]。在模块化 I 型 PKS 和 NRPS 的情况下,域字符串遵循“共线性规则”(co-linearity rule),以便根据在途径中观察到的精确域组织来合并和处理底物,生物信息学已被用于对代谢进行准确的结构预测这些途径的产物[23]。然而,越来越多的共线性异常,例如模块跳跃和口吃(stuttering )[24],为精确的、基于序列的结构预测创造了限制。目前可用于次级代谢的生物信息学工具已经过审查[25] , [26]并得到最近发布的 antiSMASH 的补充,该工具能够准确识别和提供与所有已知次级代谢产物相关的基因簇的详细序列分析班级[27]. 虽然所有这些工具都有有用的应用,但 NaPDoS 采用基于系统发育的分类系统,可用于从各种数据集中量化和区分 KS 和 C 域类型,包括通常使用下一代测序技术获得的不完整基因组组装。之所以选择这些特定域,是因为它们高度保守,并且已被证明是系统发育背景中信息量最大的域之一[28]、[29]。
系统基因组学提供了一种基于系统发育关系而非序列相似性来推断基因功能的有用方法[30]、[31]。虽然 PKS 和 NRPS 基因的进化历史由于其大小和复杂性而在很大程度上缺乏信息,但 KS 和 C 域系统发育揭示了高度支持的聚类模式。这些模式已被用于区分与孢子色素和抗生素生物合成相关的 II 型 PKS [32]、I 型模块化和混合型 PKS [33],并随后识别出许多不同的 PKS 类型[34]。KS 系统发育也被用于预测通路关联[26] , [35]在某些情况下,这些途径的次级代谢产物[28]、[36]、[37]。系统发育学也已被用于从复杂的宏基因组数据集中成功识别 PKS 序列[38]。同样,C 域系统发育清楚地描绘了功能亚型,而不是物种关系[39],并已被用于识别新的功能类别,例如“起始”C 域[29]。综上所述,已建立的 KS 和 C 结构域的系统发育关系提供了一个有效的框架,可在其中评估次级代谢物基因的丰富度和多样性,并确定可能与未表征的生物合成机制相关的新功能类别。
在这里,我们介绍了 Web 工具 Natural Product Domain Seeker (NaPDoS),它从广泛的序列数据中提取并快速分类 KS 和 C 域。结果可用于评估生物体或环境中 PKS 和 NRPS 次级代谢物生物合成的潜力,并确定新的系统发育谱系,随后可将其作为新机制生物化学的来源进行研究。我们在四个细菌基因组草图和两个宏基因组数据集上测试了 NaPDoS。结果揭示了密切相关菌株之间次生代谢物基因多样性的显着水平,并提供了一种从组装不良的基因组数据中评估次生代谢的机制。
3.2 原理
- 所有参考 KS 和 C 域的氨基酸序列使用 MUSCLE [40]或 ClustalX(版本 1.83)[41]与 BLOSUM 62 蛋白质重量矩阵进行比对。
- 使用 Mesquite [42]手动调整对齐。
- 最大似然、简约和相邻连接的系统发育树是在 Phylogeny.fr 网站 ( http://www.phylogeny.fr/ ) [43] 上使用“点菜”模式构建的。
- 最终的最大似然树是使用 PHYML [44]程序从参考数据集构建的。基于在这些树中观察到的系统发育关系进行最终域分类。
参考资料
- 2012,The Natural Product Domain Seeker NaPDoS: A Phylogeny Based Bioinformatic Tool to Classify Secondary Metabolite Gene Diversity。 https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0034064
