【5.1.2.3】Tat信号肽的预测
一 Tat的简介
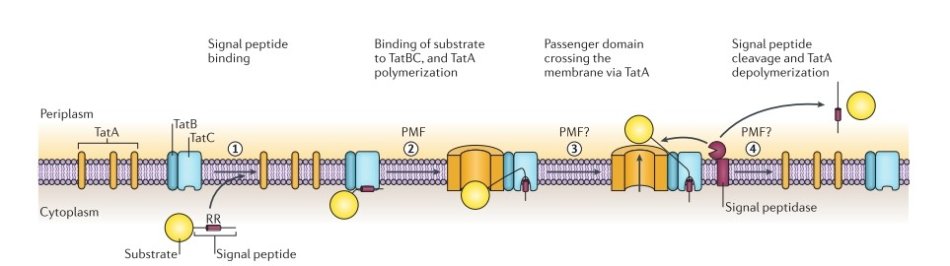
在细菌和古菌中的蛋白要想跑到周质空间,一般都会具有Sec或Tat(twin-arginine translocation))信号肽。而Sec模式转移的未结构化的蛋白,而Tat模式转移的是已经折叠好的蛋白。
Sec途径一般保守,必须,也是抓哟的蛋白跨膜转移途径。而Tat途径在一部分细菌和古菌中得到证实是必须的途径。通过Tat跨膜机制的蛋白一般都会含有保守的twin-arginine基序的信号肽。而实现这个转运的过程需要依靠膜结合蛋白TatA, TatB, TatC蛋白家族(有的物种中,仅仅TatA和TatC就可以实现)。TatA与TatBj进化距离近,差距小,容易分不清。
为什么需要Tat机制
the need for theinsertion of complex cofactors,
the avoidance of metal ions that compete for insertion into the active site of the proteins, transport as a hetero-oligomeric complex
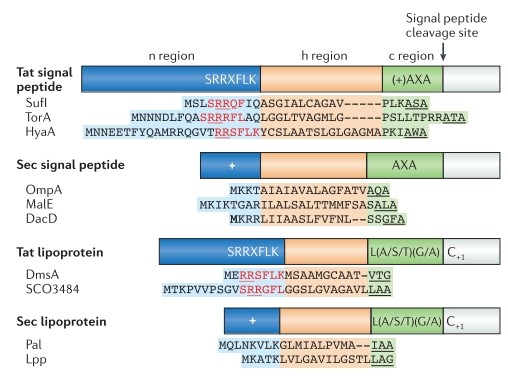
Tat信号肽的特点
信号肽三段式结构,n区,中间的疏水性h区,C端极性c区
Tat转运机制
二 Tat预测
在refseq注释的结果中搜索twin arginine translocase protein A或者Sec-independent protein或Twin-arginine translocation或twin,这里面找到的是tat蛋白. 也可以先在pfam和Interpro注释结果中搜素twin或tat,这里找到的是具有tat信号结构域或者tat结构域
再用TatP 1.0 server对感兴趣的氨基酸序列上传,进一步的确认。
TatP 1.0 server用来预测Twin-arginine signal peptide cleavage sites。 predicts the presence and location of Twin-arginine signal peptide cleavage sites in bacteria(细菌). The method incorporates a prediction of cleavage sites and a signal peptide/non-signal peptide prediction based on a combination of two artificial neural networks. A postfiltering of the output based on 正则表达 is possible.
这个工具也是可以本地使用了
预测原理
先用正则表达式来初步鉴定出可能的Tat区域,然后用神经网络的方法来确定轻微疏水性的h区域。Tat的h区的疏水性比Sec或SRP的更弱一些。
预测结果的解读
STANDARD OUTPUT
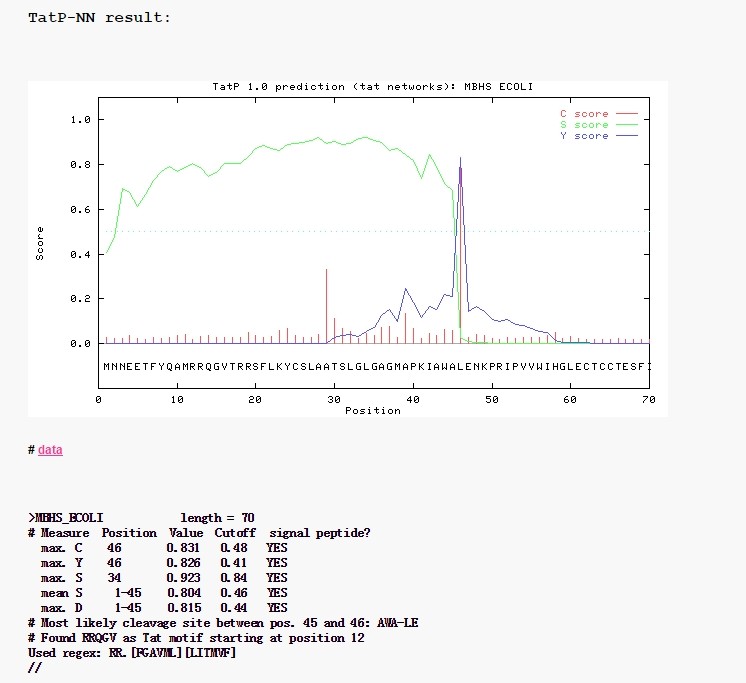
得分和图形结果的输出跟SignalP server相似,计算的方式也跟他相似。
每一个预测,两种不同神经网络被使用,一个用来预测真实的信号肽,一个用来预测信号肽切割点的位置。
- S-score用来预测序列中每个aa的信号肽可能性,分值越高,越有可能是信号肽,得分越低越有可能是非信号肽的蛋白序列。
- C-score 用来评价每个aa为信号肽与蛋白序列分割点的可能性。分值越高,那个aa越有可能是分割位点。
- Y-max 也是显示切割位点,但是结合的是C-score和S-score。这是为了纠正一条序列中发现好多个高分的C-score。
- S-mean 是S-score的平均值,起点为N-terminal到highest Y-max这段aa。
- D-score 是The D-score is introduced in SignalP version 3.0 and is a simple average of the S-mean and Y-max score.
The score shows superior discrimination performance of secretory and non-secretory proteins to that of the S-mean score which was used in SignalP version 1 and 2.
In TatP the D-score is used for final discrimination of secretory vs. non-secretory。
SHORT OUTPUT
>MBHS_ECOLI length = 70
# Measure Position Value Cutoff signal peptide?
max. C 46 0.831 0.48 YES
max. Y 46 0.826 0.41 YES
max. S 34 0.923 0.84 YES
mean S 1-45 0.804 0.46 YES
max. D 1-45 0.815 0.44 YES
# Most likely cleavage site between pos. 45 and 46: AWA-LE
# Found RRQGV as Tat motif starting at position 12
Used regex: RR.[FGAVML][LITMVF]
预测结果的争议
TatP 1.0 server预测的结果一般根据是否有
# Most likely cleavage site between pos. 45 and 46: AWA-LE
# Found RRQGV as Tat motif starting at position 12
这样的两句来判断是否是。如果这两句有,肯定就是了。
>117_8 length = 100
# Measure Position Value Cutoff Tat signal peptide?
max. C 22 0.250 0.51 NO
max. Y 22 0.328 0.35 NO
max. S 15 0.864 0.75 YES
mean S 1-21 0.691 0.30 YES
max. D 1-21 0.510 0.36 YES
# Most likely cleavage site between pos. 21 and 22: AGA-AI
# Found RRSFL as Tat motif starting at pos. 11
# Used regex: RR.[FGAVML][LITMVF]
117_8 2c795eab832be2025421488eeb8918be 333 ProSiteProfiles PS51318 Twin arginine translocation (Tat) signal profile. 1 35 7.605 T 18-07-2014 IPR006311 Twin-arginine translocation pathway, signal sequence
>125_3 length = 100
# Measure Position Value Cutoff Tat signal peptide?
max. C 31 0.590 0.51 YES
max. Y 31 0.273 0.35 NO
max. S 2 0.828 0.75 YES
mean S 1-30 0.366 0.30 YES
max. D 1-30 0.320 0.36 NO
# Most likely cleavage site between pos. 30 and 31: SFA-YY
# Used regex: RR.[FGAVML][LITMVF]
//
125_3 7c754dec20e92e9d6a4a2640a91758ab 316 TIGRFAM TIGR01409 TAT_signal_seq: Tat (twin-arginine translocation) pathway signal sequence 3 19 7.2E-4 T 18-07-2014 IPR019546 Twin-arginine translocation pathway, signal sequence, bacterial/archaeal
上面两段aa分别用TatP 1.0 server和Interpro来预测,interpro显示是,但是TatP 1.0 server给的结果中证据又似乎证据不那么确定。到底是不是呢?
ps
上面的两段用不同方法预测出来有争议的aa序列,到底具有tat吗? 我现在算他是
参考资料
- 文献: The twin-arginine translocation (Tat) protein export pathway
- Prediction of twin-arginine signal peptides
- 官网结果说明:http://www.cbs.dtu.dk/services/TatP/output.php