【3.1】宏基因组聚类-LMAT
-
下载地址 :https://computation-rnd.llnl.gov/lmat/software.php
-
参考序列数据库下载地址:ftp://gdo-bioinformatics.ucllnl.org/pub/lmat/
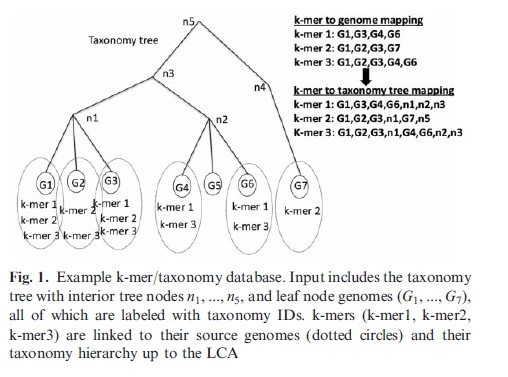
LMAT不同于序列组成分类方法,因为他用的k-mer为17到20,不同于比对的方法,每个k-mer。 参考序列的数据库中的每条基因组序列都会有一个分类上的标记。所以第一步就是把原始的参考基因组数据库变成一个可以搜索的K-mer分类数据库,通过保存带有物种信息的每个重叠的K-mer。一张图说明问题,例如包含kmer1,kmer2,kmer3的序列我就就默认为他为物种G1。The reference set includes 1272 bacterial species, 121 archaeal species, 3048 viral species and 335 eukaryotic species. Microbial genome segments range in length from a small number of single read contigs of length less than 100 bases up to a 13 033 779-base chromosome (for Sorangium cellulosum).
这里是一个广告位,,感兴趣的都可以发邮件聊聊:tiehan@sina.cn
![]() 个人公众号,比较懒,很少更新,可以在上面提问题,如果回复不及时,可发邮件给我: tiehan@sina.cn
个人公众号,比较懒,很少更新,可以在上面提问题,如果回复不及时,可发邮件给我: tiehan@sina.cn

