【3.2.1】sam格式
SAM是一种序列比对格式标准,由sanger制定,是以TAB为分割符的文本格式。主要应用于测序序列mapping到基因组上的结果表示,当然也可以表示任意的多重比对结果。当测序得到的fastq文件map到基因组之后,我们通常会得到一个sam或者bam为扩展名的文件。SAM的全称是sequence alignment/map format。而BAM就是SAM的二进制文件(B取自binary)。
一、Sam详解
HWI-ST1001:137:C12FPACXX:7:1115:14131:66670 0 chr1 12805 1 42M4I5M * 0 0 TTGGATGCCCCTCCACACCCTCTTGATCTTCCCTGTGATGTCACCAATATG CCCFFFFFHHGHHJJJJJHJJJJJJJJJJJJJJJJIJJJJJJJJJJJJIJJ AS:i:-28 XN:i:0 XM:i:2 XO:i:1XG:i:4 NM:i:6 MD:Z:2C41C2 YT:Z:UU NH:i:3 CC:Z:chr15 CP:i:102518319 XS:A:+ HI:i:0
<QNAME> <FLAG> <RNAME> <POS> <MAPQ> <CIGAR> <MRNM> <MPOS> <ISIZE> <SEQ> <QUAL> [<TAG>:<VTYPE>:<VALUE> [...]]
ST-E00144:375:H32VMALXX:2:1215:9912:44890 147 17 41246523 60 150M = 41246497 -176 AGATCTACCTTTTTTTCTGTGCTGGGAGTCCGCCTATCATTACATGTTTCCTTACTTCCAGCCCATCTGTTATGTTGGCCCCTTGCTAAGCCAGGCTGTTTGCTTTTATTACAGAATTCAGCCTTTTCTACATTCATTCTGTCTTTAGTG JJFJJFAFJJJJJJJJJJJJJFJJJJJJJJFJJJJJJJJJJJJJJJJJJJJJJJJJJJJFJJFJJJJJJJJFJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJFJJFJJJJJJFFFAA MD:Z:79T70 RG:Z:GWA5720199 NM:i:1 AS:i:145 XS:i:20
SAM分为两部分:
- 注释信息(header section)
- 比对结果部分(alignment section)
注释信息可有可无,都是以@开头,用不同的tag表示不同的信息,主要有
- @HD,说明符合标准的版本、对比序列的排列顺序;
- @SQ,参 考序列说明;@RG,比对上的序列(read)说明;
- @PG,使用的程序说明;
- @CO,任意的说明信息。
SAM文件由两部分组成,头部区和主体区,都以tab分列。

-
头部区:以’@‘开始,体现了比对的一些总体信息。比如比对的SAM格式版本,比对的参考序列,比对使用的软件等。
-
主体区:比对结果,每一个比对结果是一行,有11个主列和一个可选列。
主体区部分:
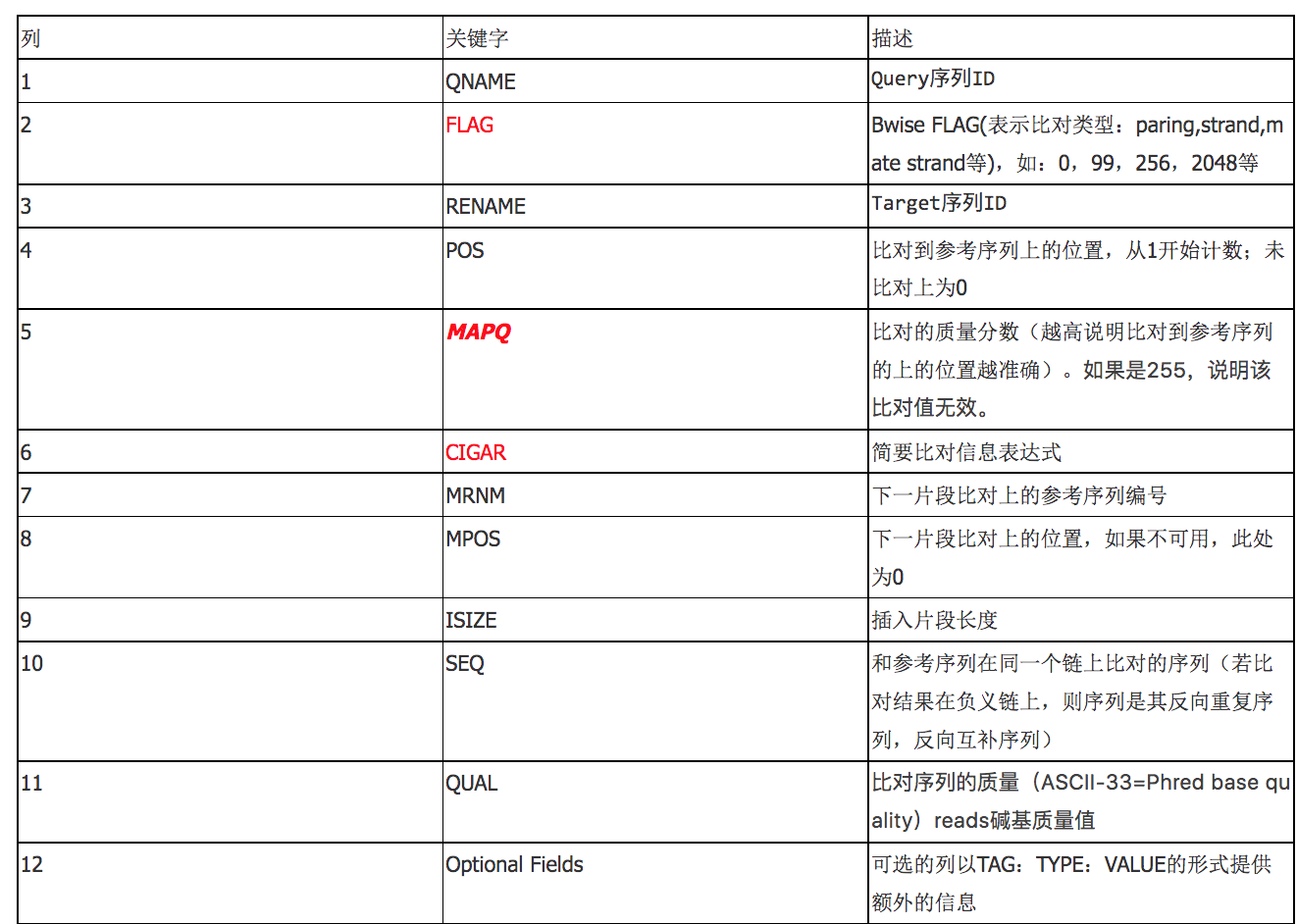
比对结果部分(alignment section),每一行表示一个片段(segment)的比对信息,包括11个必须的字段(mandatory fields)和一个可选的字段,字段之间用tag分割。必须的字段有11个,顺序固定,不可用时,根据字段定义,可以为’0‘或者’*‘,这是11个字 段包括:
1.1 QNAME
比对片段的(template)的编号;read的名字通常包括测序平台等信息
1.2. FLAG
位标识,template mapping情况的数字表示,每一个数字代表一种比对情况,这里的值是符合情况的数字相加总和;
Flag标识对应的情况说明:
| flag | details |
|---|---|
| 1 | The read is one of a pair read是pair中的一条(read表示本条read,mate表示pair中的另一条read) |
| 2 | The alignment is one end of a proper paired-end alignment pair一正一负完美的比对上 |
| 4 | The read has no reported alignments 这条read没有比对上 |
| 8 | The read is one of a pair and has no reported alignments mate没有比对上 |
| 16 | The alignment is to the reverse reference strand 这条read反向比对 |
| 32 | The other mate in the paired-end alignment is aligned to the reverse reference strand mate反向比对 |
| 64 | The read is the first (#1) mate in a pair 这条read是read1 |
| 128 | The read is the second (#2) mate in a pair 这条read是read2 |
| 256 | 第二次比对 |
| 512 | 比对质量不合格 |
| 1024 | read是PCR或光学副本产生 |
| 2048 | 辅助比对结果 |
通过这个和可以直接推断出匹配的情况。假如说标记不是以上列举出的数字,比如说83=(64+16+2+1),就是这几种情况值和。

1.3.RNAME
reference sequence name,实际上就是比对到参考序列上的染色体号。若是无法比对,则是*;
1.4.POS
比对上的位置,注意是从1开始计数,没有比对上,此处为0;
1.5.MAPQ
mapping的质量;比对的质量分数,越高说明该read比对到参考基因组上的位置越唯一;
1.6.CIGAR
简要比对信息表达式(Compact Idiosyncratic Gapped Alignment Report),其以参考序列为基础,使用数字加字母表示比对结果,比如3S6M1P1I4M,前三个碱基被剪切去除了,然后6个比对上了,然后打开了一 个缺口,有一个碱基插入,最后是4个比对上了,是按照顺序的;
M”表示 match或 mismatch;
“I”表示 insert;
“D”表示 deletion;
“N”表示 skipped(跳过这段区域);
“S”表示 soft clipping(被剪切的序列存在于序列中);
“H”表示 hard clipping(被剪切的序列不存在于序列中);
“P”表示 padding;
“=”表示 match;
“X”表示 mismatch(错配,位置是一一对应的);

1.7.MRNM(chr)
下一个片段比对上的参考序列的编号,没有另外的片段,这里是’*‘,同一个片段,用’=‘;
1.8.mate position
下一个片段比对上的位置,如果不可用,此处为0;
1.9.ISIZE
Template的长度,mate position大于POS为正,否则为负,中间的不用定义正负,不分区段(single-segment)的比对上,或者不可用时,此处为0;Inferred fragment size.详见Illumina中paired end sequencing 和 mate pair sequencing,是负数,推测应该是两条read之间的间隔(待查证),若无mate则为0;
ISIZE为正,说明amplicon的start 为POS,amplicon 的end为 pos + isize-1
ISIZE为负,说明amplicon的start为mate position,amplicon的end为mate position -isize-1
ISIZE为0,说明无法计算出amlicon的大小
ST-E00144:375:H32VMALXX:2:2117:4878:44152 65 17 41243501 60 150M 1 32799454 0 CGTTCCTCTTTCTTCATCATCTGAAACCAATTCCTTGTCACTCAGACCAACTCCCTGGCTTTCAGACTGATGCCTCATTTGTTTGGAAGAACCAATCAAGAAAGGATCCTGGGTGTTTGTATTTGCAGTCAAGTCTTCCAATTCACTGCA -AFFFAJFJA<AJAAAJ7JA77AJFFFJJJF-F<AAA-F<7-7<7F<JJJJJJFFJJFJJJJ7<-<7FJ<FJ7-<7<J7FJJJJFFAAF-AFAAA<FF<JAJ<FJAJJF-77-AJJF7-<AAFJJ-A-FFAFAFFJJ--F<J<JJJJJ77 MD:Z:150 RG:Z:GWA5720199 NM:i:0 AS:i:150 XS:i:0
1.10.Sequence
序列片段的序列信息,如果不存储此类信息,此处为’*‘,注意CIGAR中M/I/S/=/X对应数字的和要等于序列长度;就是read的碱基序列,如果是比对到互补链上则是reverse completed
1.11.ASCII
read质量的ASCII编码。
1.12.Optional fields
可选的区域
格式如:TAG:TYPE:VALUE,其中TAG有两个大写字母组成,每个TAG代表一类信息,每一行一个TAG只能出现一次,TYPE表示TAG对应值的类型,可以是字符串、整数、字节、数组等。
二、更多资料
- http://samtools.github.io/hts-specs/SAMv1.pdf
- http://biobits.org/samtools_primer.html
- http://blog.csdn.net/u014182497/article/details/51691743
参考资料
- 官方使用说明手册 http://computing.bio.cam.ac.uk/local/doc/bowtie2.html
- 糗世界 http://pgfe.umassmed.edu/ou/archives/3050
- 阿飞与安达:http://liuwei441005.blog.163.com/blog/static/135705811201322331740144/
- https://blog.sciencenet.cn/home.php?mod=space&uid=994715&do=blog&id=1341509
- https://mp.weixin.qq.com/s?__biz=MzI2MjA1MDQxMg==&mid=2649709965&idx=2&sn=a5a7b2de70ad0fac61eb541946a1a8f9&chksm=f24afa8ec53d739824702496dc642693c05c2af40a85a590921c46ee403c14d90b0abdd0ef67&scene=21#wechat_redirect
