【2.1.2】AMBER分子动力学模拟
适用于amber18
一、前言
本教程旨在提供Amber分子动力学模拟的简介。它是围绕AMBER Tools v14设计的,并且假定您以前从未使用过Linux或Amber。它是为想要学习如何运行分子动力学模拟的新用户而设计的。但是,它确实假定您有一台正确安装了AmberTools v15,VMD和xmgrace的计算机。
AMBER代表辅助模型构建和能量提炼。它不仅涉及分子动力学程序,还涉及描述力势函数和生物分子相互作用参数的一组力场。
为了在Amber中运行分子动力学模拟,每个分子的相互作用都由分子力场描述。力场具有为每个分子定义的特定参数。
- sander是Amber的基本MD引擎。
- pmemd是MD引擎的高性能实现,其中包含sander功能的子集。 pmemd也可以通过图形处理单元(GPU)加速运行。
为了使用sander或pmemd运行MD模拟,需要三个关键文件:
- parm7 描述系统中分子的参数和拓扑的文件
- rst7 描述系统初始分子坐标的文件
- mdin 描述Amber MD引擎设置的文件
二、Linux入门
Amber MD是完全基于Linux的计算机上的命令行界面(CLI)的软件。 要运行Amber,您需要打开一个终端。
2.1 现在在Linux计算机上打开一个终端。
在大多数Linux机器上,您的终端将如下所示:
本教程的大部分工作将主要通过终端完成。
首次登录并启动终端时,当前的工作目录(或文件夹)就是您的主目录。它与您的用户名同名,是文件和目录的存储位置。在大多数情况下,这是/home/username
2.2 ls (list)
使用ls命令列出当前目录中的内容。
请注意,“ $”是终端中的命令行提示符。
$ ls
此时,您的主目录中可能会有一些文件和目录是使用您的帐户自动创建的。
2.3 mkdir (make directory)
您需要一个新目录来存储本教程中创建的文件和文件夹。
使用名为Tutorial的mkdir命令为本教程创建一个新目录。
$ mkdir Tutorial
现在,当您执行ls时,您应该看到新目录已创建。
$ ls
Tutorial
2.4 cd (change directory)
此时,您将需要进入Tutorial目录,以便将所有工作文件保存在那里。
使用cd命令更改为其他目录。
$ cd Tutorial
$ ls
有一个名为“ ..”的特殊目录。这意味着当前目录的父目录。因此,要返回到Tutorial目录的父目录,请使用cd ..
$ cd ..
$ ls
Tutorial
如果您需要返回主目录,请仅使用cd命令。连字符“〜”是主目录的快捷方式。以下命令都将目录更改为您的主目录。
$ cd
$ cd ~
2.5 pwd (print working directory)
路径名描述相对于整个计算机文件系统的目录。您的主目录在整个文件系统中都有位置。
使用pwd打印主目录的工作目录路径名。
$ cd
$ pwd
/home/username
这是您当前所在的工作目录。在这种情况下,目录用户名位于/(根)目录中的目录主目录中。
三、准备拓扑和坐标文件 Prepare topology and coordinate files
对于本教程,您将在名为xLEaP的准备程序中构建以下分子,以在AMBER中进行模拟。

为了构建和溶解(solvate)该分子,您将需要启动xLEaP。 xLEaP具有另一个命令行界面和简单的分子图形,用于构建系统拓扑并定义分子的参数。
3.1 立即使用xleap命令启动xLEaP。
$ xleap
-I: Adding /usr/local/amber_14/amber/dat/leap/prep to search path.
-I: Adding /usr/local/amber_14/amber/dat/leap/lib to search path.
-I: Adding /usr/local/amber_14/amber/dat/leap/parm to search path.
-I: Adding /usr/local/amber_14/amber/dat/leap/cmd to search path.
您应该看到一个这样的窗口:
警告: 不要在任何LEaP窗口上单击“ X”。 它将完全退出LEaP。
注意: 此时,最好关闭Num Lock,以使菜单起作用。
3.2 加载蛋白质和核酸力场
MD力场是哈密顿量(Hamiltonian,势能函数)和相关参数,它们描述了系统中分子之间的分子内和分子间相互作用。 在MD中,哈密顿量被整合为描述分子的力和速度。
amber哈密顿量的基本形式为:
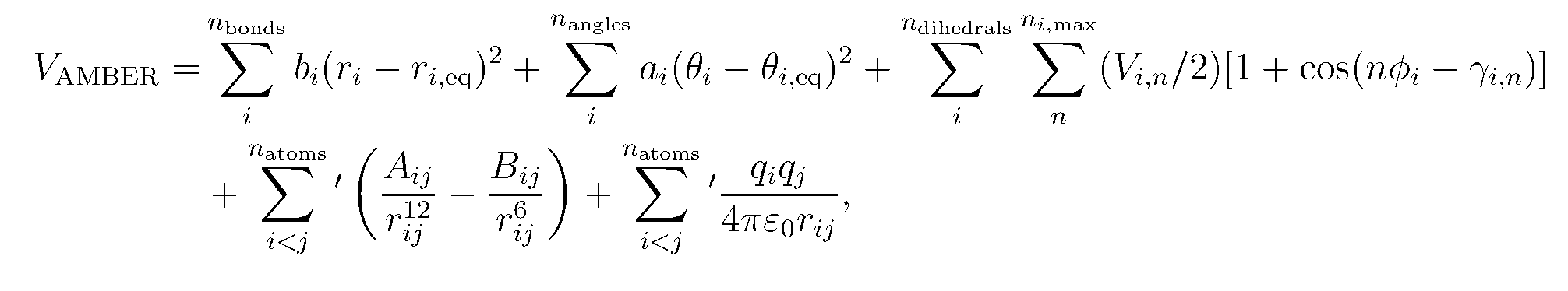
为了进行分子动力学模拟,我们需要加载一个力场来描述丙氨酸二肽的势能。 我们将对蛋白质和核酸使用AMBER力场FF14SB。 FF14SB基于FF12SB(FF99SB的更新版本),而FF12SB则基于原始 Amber Cornell et al (1995) [ff94]力场。 FF14SB力场的最重要变化包括更新了蛋白质Phi-Psi角的扭转项以及对侧链的扭转项的修正。 这些共同改善了这些分子中α螺旋的估计。
现在获取(加载)FF14SB力场。
> source leaprc.protein.ff14SB
Loading parameters:
/usr/local/amber14/dat/leap/parm/frcmod.ff14SB
Reading force field modification type file (frcmod)
Reading title:
3.3 构建丙氨酸二肽 Build alanine dipeptide
我们可以构建丙氨酸二肽,将其作为丙氨酸氨基酸,在N端盖一个乙酰基,在C端盖一个正甲基酰胺。 在加载力场ff14SB之后,xLEaP现在具有可用于构建分子的这些“单位”(units)。 sequence命令将根据可用单元创建一个新单元,并将它们连接在一起。
使用sequence从ACE,ALA和NME单元中创建一个名为foo的新单元。 请注意,“>”表示xLEaP中的命令行。
> foo = sequence { ACE ALA NME }
现在,您在foo单元中存储了一个丙氨酸二肽分子。 xLEaP提供了一个非常基本的编辑器来检查和更改单元和分子。
检查丙氨酸二肽分子的结构。 使用edit命令查看结构。
> edit foo
编辑窗口将如下所示:
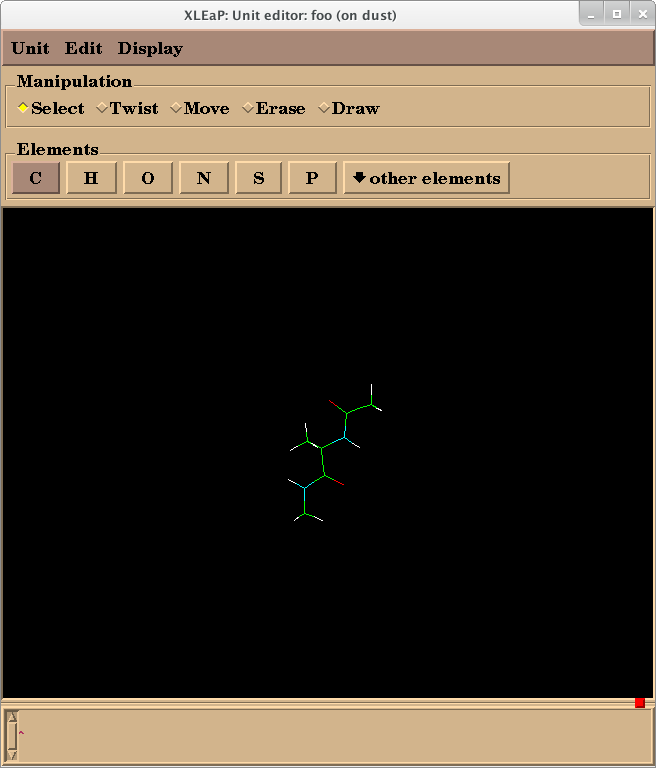
从这里,您可以检查分子的拓扑结构,结构,原子名称,原子类型和部分电荷。 分子的基本编辑也是可能的。
警告: 不要单击“ X”关闭此窗口。 它将退出xLEaP。 要关闭此窗口,请使用Unit -> Close。
3.4 溶剂化丙氨酸二肽 Solvate alanine dipeptide
制备该丙氨酸二肽系统的下一步是用明确的水分子溶剂化该分子。 在此模拟中,我们将使用添加的TIP3P水分子到系统中。 在这种类型的模拟中,系统具有周期性边界条件,这意味着离开系统一侧的分子将包裹到系统的另一侧。 重要的是,周期盒必须足够大,即丙氨酸二肽周围有足够的水,以使丙氨酸二肽分子不与其周期图像相互作用。
有许多水模型可用于MD模拟。 但是,对于本教程,我们将使用TIP3P水模型进行此模拟。
使用solvatebox命令解决系统问题。
> source leaprc.water.tip3p
> solvatebox foo TIP3PBOX 10.0
TIP3PBOX指定要溶解的水箱的类型。 10.0表示该分子在丙氨酸二肽和周期框壁之间应具有至少10埃的缓冲液。
3.5 保存Amber parm7和rst7输入文件
现在,我们将parm7和rst7文件保存到当前工作目录中。 现在,单元foo包含丙氨酸二肽分子,水分子和模拟所需的周期框信息( periodic box)。 参数将从ff99SB力场分配。
要保存parm7和rst7文件,请使用saveamberparm命令。
> saveamberparm foo parm7 rst7
Checking Unit.
Building topology.
Building atom parameters.
Building bond parameters.
Building angle parameters.
Building proper torsion parameters.
Building improper torsion parameters.
total 4 improper torsions applied
Building H-Bond parameters.
Incorporating Non-Bonded adjustments.
Not Marking per-residue atom chain types.
Marking per-residue atom chain types.
(Residues lacking connect0/connect1 -
these don't have chain types marked:
res total affected
WAT 630
)
(no restraints)
警告: 对于您的parm7和rst7文件未正确构建的任何警告或错误,请密切注意此命令的输出。
3.6 amber参数/拓扑和坐标文件
丙氨酸二肽parm7和输入文件位于此处:
- parm7 *rst7
$$$ 3.7 退出xLEaP
要退出xLEaP,请使用quit。
> quit
四、准备Amber MD sander输入文件
最后需要的组件是定义每个MD运行的程序设置的输入文件。 对于此系统,我们将在系统上执行能量最小化,然后缓慢加热系统,然后在所需的温度和压力下进行生产MD。
- 最小化 minimization
- 从0K到300K以恒定的体积和温度(NVT)加热20ps
- 在300K和1atm的恒定压力和温度(NPT)下生产MD 60ps
我们将保存轨迹并每2ps写入输出文件一次。 Langevin thermostat将用于控制温度。 随机数生成器将使用随机种子初始化。
为了控制所有这些设置,我们将在文本编辑器中编写一个简单的输入文件。 Linux有许多可用的文本编辑器,但是我们将使用Linux计算机上随附的简单文本编辑器。
4.1 在Linux计算机上打开gedit文本编辑器。
gedit的界面如下所示:

最小化输入
4.2 创建文件01_Min.in,其中包括以下设置以最小化:
Minimize
&cntrl
imin=1,
ntx=1,
irest=0,
maxcyc=2000,
ncyc=1000,
ntpr=100,
ntwx=0,
cut=8.0,
/
设置可以总结如下:
- imin = 1 选择minimization运行
- ntx = 1 从ASCII格式的rst7坐标文件中读取坐标但不读取速度
- irest = 0 不要重新启动模拟。 (不适用于最小化)
- maxcyc = 2000 Maximum minimization cycles
- ncyc = 1000 前0-ncyc周期的最速下降算法,然后切换ncyc-maxcyc周期的共轭梯度算法
- ntpr = 100 每个ntpr周期打印到Amber mdout输出文件
- ntwx = 0 未写入amber mdcrd轨迹文件(不适用于最小化)
- cut= 8.0 无键截止距离(以埃为单位)(对于PME,直接空间总和的限制-请勿将其减小到8.0以下。数值越大,精度越高,但计算量却大大增加。)
4.3 加热输入 Heating input
创建文件02_Heat.in,其中包括以下加热设置:
Heat
&cntrl
imin=0,
ntx=1,
irest=0,
nstlim=10000,
dt=0.002,
ntf=2,
ntc=2,
tempi=0.0,
temp0=300.0,
ntpr=100,
ntwx=100,
cut=8.0,
ntb=1,
ntp=0,
ntt=3,
gamma_ln=2.0,
nmropt=1,
ig=-1,
/
&wt type='TEMP0', istep1=0, istep2=9000, value1=0.0, value2=300.0 /
&wt type='TEMP0', istep1=9001, istep2=10000, value1=300.0, value2=300.0 /
&wt type='END' /
设置可以总结如下:
- imin= 0 选择分子动力学(MD)运行[不最小化]
- nstlim = 10000 运行中的MD步骤数(nstlim * dt =运行时间,以ps为单位)
- dt = 0.002 时间步长以皮秒(ps)为单位。 每个MD步骤的时间长度
- ntf = 2 设置为不计算SHAKE约束键的力
- ntc = 2 使SHAKE约束所有涉及氢的键
- tempi = 0.0 初始恒温器温度,单位为K(请参见NMROPT部分)
- temp0 = 300.0 最终恒温器的温度,单位为K(请参见NMROPT部分)
- ntwx = 1000 每ntwx个步骤写入一个amber轨迹文件mdcrd
- ntb = 1 周期性边界用于恒定体积
- ntp = 0 无压力控制
- ntt = 3 用Langevin thermostat进行温度控制
- gamma_ln = 2.0 Langevin thermostat碰撞频率
- nmropt = 1 读取NMR限制和重量变化(请参阅NMROPT部分)
- ig = -1 为伪随机数生成器随机化种子[除非您正在调试模拟问题,否则总是个好主意]
Thermostat temperature via NMROPT
最后三行允许thermostat在整个模拟过程中更改其目标温度。 对于前9000个步骤,温度将从0K升高到300K。 对于步骤9001到10000,温度将保持在300K。
4.3 Production input
警告: 就其本身而言,此输入文件不适用于一般的MD模拟。
NTPR和NTWX设置得很低,因此可以分析这种简短的模拟。 将这些设置用于更长的MD模拟将创建非常大的输出和轨迹文件,并且将比常规MD慢。 对于实际生产的MD,您需要增加NTPR和NTWX。
此模拟的生产时间仅为60ps。 理想情况下,我们将模拟运行更长的时间,但是出于本教程的时间考虑,我们限制了生产模拟的时间。
使用生产MD的设置创建文件03_Prod.in:
Production
&cntrl
imin=0,
ntx=5,
irest=1,
nstlim=30000,
dt=0.002,
ntf=2,
ntc=2,
temp0=300.0,
ntpr=100,
ntwx=100,
cut=8.0,
ntb=2,
ntp=1,
ntt=3,
gamma_ln=2.0,
ig=-1,
/
生产设置可以总结如下:
- ntx = 5 从未格式化的rst7坐标文件中读取坐标和速度
- irest = 1 重新启动先前的MD运行[这意味着rst7文件中应该有速度,并且该速度将用于提供初始原子速度]
- temp0 = 300.0 Thermostat温度。 以300K运行
- ntb = 2 使用恒定压力的周期性边界(periodic boundary)条件
- ntp = 1 使用 Berendsen barostat进行恒压模拟
输入文件
sander输入文件在这里可用:
五、运行Amber MD sander
现在我们有了所有要素:参数和拓扑文件parm7,坐标文件rst7和输入文件01_Min.in,02_Heat.in,03_Prod.in,我们准备运行实际的最小化,加热和生产MD 。
为此,我们将使用程序Sander,Amber的通用MD引擎(还有一个高性能版本,称为pmemd,它是AMBER商业版本的一部分,也是MD引擎的最佳选择,但出于以下目的: 本教程的sander就足够了)。 从命令行运行sander。 在命令行上,我们可以指定其他几个选项,然后选择要用于输入的文件。
首先,在终端上,您需要将包含所有输入文件的目录更改为Tutorial目录。 “〜”是创建主目录的主目录的快捷方式。
$ cd ~/Tutorial
5.1 运行最小化 Run minimization
用sander运行最小化丙氨酸二肽。
$ $AMBERHOME/bin/sander -O -i 01_Min.in -o 01_Min.out -p parm7 -c rst7 -r 01_Min.ncrst -inf 01_Min.mdinfo
Sander在MD模拟的每个步骤中使用一致的语法。以下是sander命令行选项的摘要:
- -O 覆盖输出文件(如果已经存在)
- -i 01_Min.in选择输入文件(默认为mdin)
- -o 01_Min.out 写入输出文件(默认为mdout)
- -p parm7 选择参数和拓扑文件parm7
- -c rst7 选择坐标文件rst7
- -r 01_Min.ncrst 用坐标和速度写入输出重启文件(默认为restrt)
- -inf 01_Min.mdinfo 写入具有模拟状态的MD信息文件(默认为mdinfo)
取决于您的计算机规格,sander会在适当的时间(〜27秒)内完成最小化。
sander完成之后,应该有一个输出文件01_Min.out,一个重新启动文件01_Min.ncrst和一个MD信息文件01_Min.mdinfo。您将使用重启文件01_Min.ncrst来加热系统。
最小化输出文件
最小化输出文件在这里可用:
使用gedit,打开输出文件01_Min.out。
在01_Min.out文件中,您将找到最小化的详细信息。在整个最小化过程中,您应该能够看到系统能量ENERGY降低。
5.2 运行加热MD
现在,使用最初最小化时的重启文件,我们将加热系统。
用sander加热丙氨酸二肽。
$ $AMBERHOME/bin/sander -O -i 02_Heat.in -o 02_Heat.out -p parm7 -c 01_Min.ncrst -r 02_Heat.ncrst -x 02_Heat.nc -inf 02_Heat.mdinfo
以下是sander命令行选项的摘要:
- -c 01_Min.ncrst 现在,对于输入坐标,我们从最小化中选择重新启动文件
- -x 02_Heat.nc MD模拟的输出轨迹文件(默认nc)
取决于您的计算机规格,sander在适当的时间内(约2.5分钟)完成加热。
加热输出文件
加热输出文件在这里。 有些文件已压缩,需要解压缩。
02_Heat.out 02_Heat.ncrst 02_Heat.nc
打开输出文件02_Heat.out以查看系统输出。
在02_Heat.out文件中,您将找到加热MD的输出。 您应该能够看到系统信息,包括时步能量和温度。 例如,在1000时间步长上:
NSTEP = 1000 TIME(PS) = 2.000 TEMP(K) = 29.48 PRESS = 0.0
Etot = -6944.9552 EKtot = 112.3015 EPtot = -7057.2567
BOND = 1.0442 ANGLE = 1.7653 DIHED = 9.4906
1-4 NB = 2.6284 1-4 EEL = 46.3073 VDWAALS = 1448.7074
EELEC = -8567.1999 EHBOND = 0.0000 RESTRAINT = 0.0000
Ewald error estimate: 0.4641E-03
------------------------------------------------------------------------------
NMR restraints: Bond = 0.000 Angle = 0.000 Torsion = 0.000
===============================================================================
一些重要的输出包括:
- NSTEP MD模拟所处的时间步
- TIME 模拟的总时间(包括重新启动)
- TEMP 系统温度
- PRESS 系统压力
- Etot 系统总能量
- EKtot 系统总动能
- EPtot 系统的总势能
请注意,压力为0.0,因为加热中未使用barostat(压力控制)。
5.3 运行生产MD
至此,最小化和加热已完成。 我们继续进行实际生产MD。
用sander运行丙氨酸二肽的生产MD。
$ $AMBERHOME/bin/sander -O -i 03_Prod.in -o 03_Prod.out -p parm7 -c 02_Heat.ncrst -r 03_Prod.ncrst -x 03_Prod.nc -inf 03_Prod.info &
注意:在命令末尾加上“&”,sander现在在后台运行
现在,sander在后台运行。运行生产MD将需要一些时间。
但是我们想监视生产MD的状态。因此,我们将监视sander输出文件以检查运行状态。 Linux程序的尾部将打印输出文件的末尾。
要监视此作业的状态,请使用程序尾部将输出文件打印到终端。
$ tail -f 03_Prod.out
这会在运行sander时打印输出文件。跟踪工作很有用。您还可以监视mdinfo文件(目录03_Prod.info),该文件提供详细的性能数据以及估计的完成时间。
.要退出尾部,请按
完成MD模拟
让MD模拟运行。完成模拟需要花费一些时间[〜10分钟]。
生产产量 Production output
生产MD输出可在此处获得:
03_Prod.out 03_Prod.ncrst 03_Prod.nc
完成后,打开输出文件以检查模拟是否正常完成。
使用gedit打开输出文件03_Prod.out以查看MD模拟的输出。
六、可视化结果
现在,您已经运行了MD模拟。 为了使结果可视化,我们现在将使用称为VMD(可视分子动力学,Visual Molecular Dynamics)的程序。 这是一个分子图形程序,可以在3D模式下渲染分子结构。 VMD不仅加载蛋白质数据库(PDB)结构文件,而且还加载许多程序的MD轨迹。 [有关VMD的更深入的教程可作为可选的动手课程在讲习班计划页面的底部列出]。
要启动VMD,请打开一个终端,将目录更改为Tutorial目录中的Tutorial文件,然后运行vmd。 记住〜/Tutorial是您Tutorial目录的快捷方式。
$ cd ~/Tutorial
$ vmd
VMD应该如下所示:
VMD是使蛋白质,核酸和其他生物分子原子结构可视化的非常有用的工具。最常见的格式之一是PDB生物分子结构格式。要加载PDB,请进入File-> New Molecule….。然后为New Molecule加载文件并选择PDB文件。 VMD应该自动确定文件类型。
但是,我们想可视化丙氨酸二肽的轨迹。现在,我们将加载我们的MD轨迹,以研究丙氨酸二肽的动力学。
- 使用File-> New Molecule …创建一个新分子。
- 加载新分子文件。然后选择Amber参数和拓扑文件parm7。将文件类型设置为AMBER7 Parm。单击加载。
- 加载0的文件:parm7。然后选择amber轨迹文件 03_Prod.nc。将文件类型设置为NetCDF(AMBER,MMTK)。单击加载。
VMD现在会加载要可视化的轨迹。 VMD主窗口可用于控制播放。
您应该能够在显示屏上看到丙氨酸二肽分子以及许多水分子。您可以使用鼠标旋转,缩放和平移显示器周围的分子。

可以在 Graphics -> Representations 窗口中更改许多不同的可视化选项。
您的可视化也可以仅限于丙氨酸二肽。
-
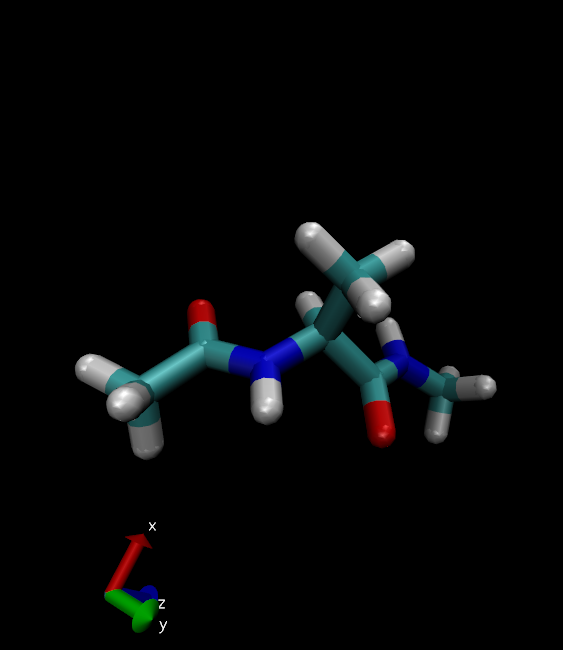
将“Selected Atoms ”更改为全部非水。您可以将分子的drawing方法更改为更有趣的模型。
-
将Drawing Method更改为Licorice。
丙氨酸二肽将如下所示:
更多VMD
VMD具有许多可用于分析和研究MD轨迹的功能。 例如,可以对齐分子,测量均方根偏差(RMSD),保存轨迹的结构以及测量整个轨迹的物理系统参数。 也可以渲染具有轨迹的影片。
但是,这些功能不在本介绍性内容的范围之内。 有关更多详细信息,请参阅AMBER教程主页上的VMD教程。
七、分析MD结果
Amber包括一套用于检查和分析MD轨迹的工具。 在本教程中,我们将使用几个Amber程序进行简单分析并绘制结果。 分析将主要从终端中的命令行完成。
打开一个终端,然后将目录更改为您的教程文件。
$ cd ~/Tutorial
创建一个Analysis目录并切换到该目录。
$ mkdir Analysis
$ cd Analysis
现在,我们将使用分析脚本process_mdout.perl分析MD输出文件。 该脚本将从MD输出文件中提取能量,温度,压力,密度和体积,并将它们保存到单独的数据文件中。
使用process_mdout.perl处理MD输出文件
$ $AMBERHOME/bin/process_mdout.perl ../02_Heat.out ../03_Prod.out
现在,绘制保存在输出文件中的数据非常简单。
我们将使用一个名为xmgrace的绘图程序,在整个模拟过程中自动为以下MD模拟属性(simulation properties)生成图。 为方便起见,我们使用此程序,但您可以使用任何选择的绘图程序。
- MD模拟温度
- MD模拟密度
- MD模拟的总,势能和动能。
但是,对于MD模拟密度,模拟的加热部分不包括密度输出。 您将需要编辑summary.DENSITY 文件以删除空数据点,以使xmgrace正常工作。
使用gedit编辑summary.DENSITY文件以删除空数据点(最高20ps)。
使用以下命令通过仿真绘制这些属性。
$ xmgrace summary.TEMP
$ xmgrace summary.DENSITY
$ xmgrace summary.ETOT summary.EPTOT summary.EKTOT
分析数据文件
分析数据文件位于此处:
生产MD系统属性
警告: 我们应该将模拟运行更长的时间,以使密度达到平衡并且模拟收敛。 但是,为了节省时间,本教程将生产MD模拟时间设置得非常短,以便可以分析结果。
生成的图应类似于以下内容:
丙氨酸二肽MD温度
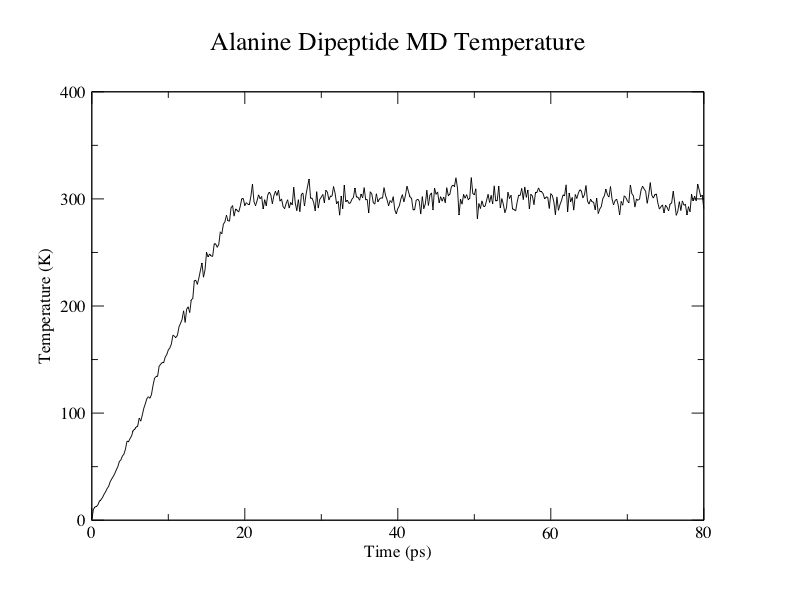
在这里,您可以看到加热期间(0-20ns)温度的线性增加。 接下来是在生产模拟过程中大约300 K的相对稳定的温度波动。
丙氨酸二肽MD密度
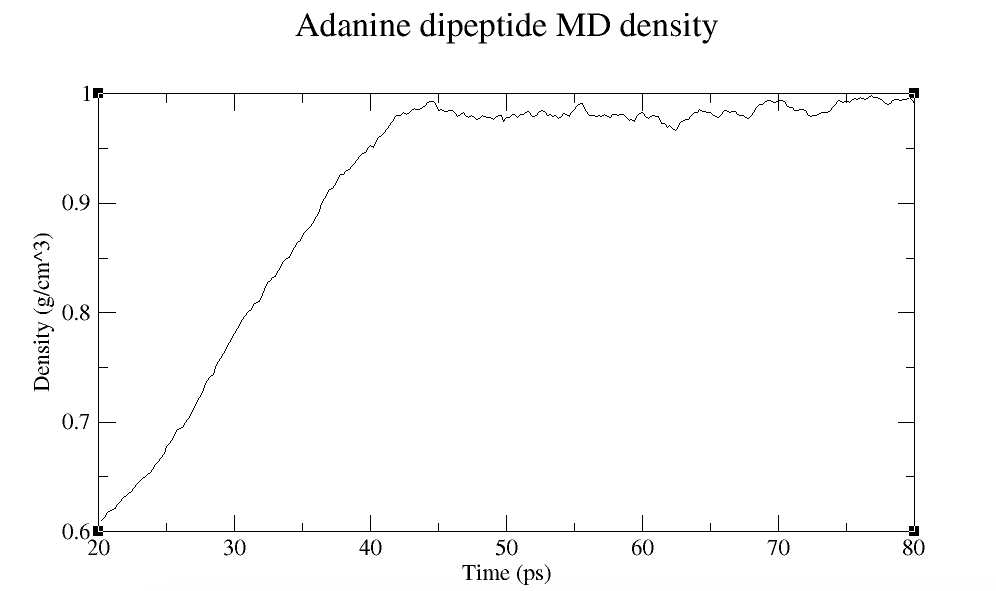
在20-80ps期间,密度达到大约1 g/cm^3。 随着系统密度的收敛,这对应于周期盒(periodic box)尺寸的变化。
丙氨酸二肽MD的总,势和动能
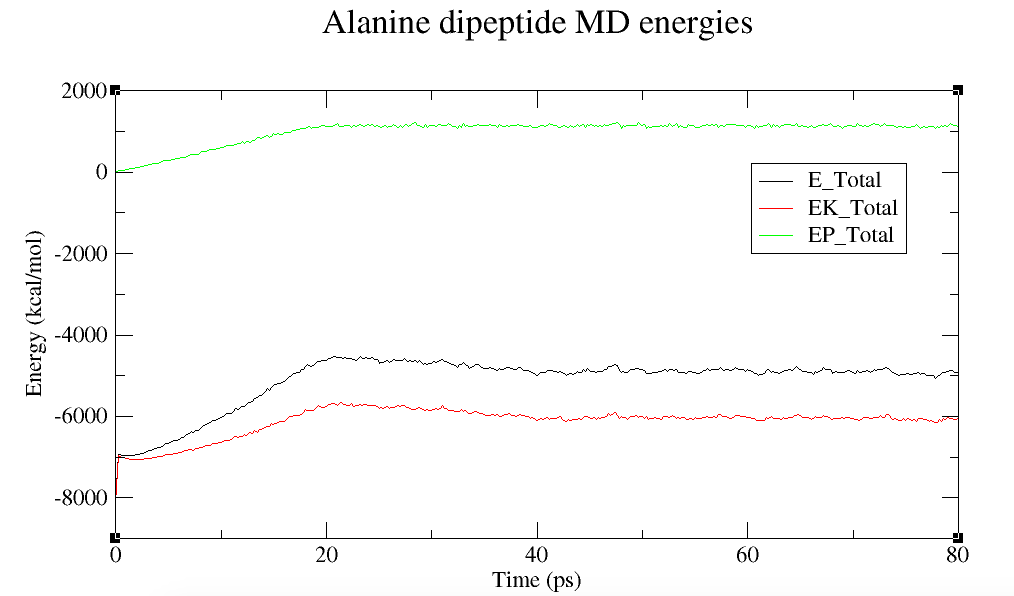
图显示了可以分解为总势能和总动能的总系统能量。
八、使用cpptraj分析RMSD
均方根偏差(RMSD)值是对结构内部原子坐标相对于某些参考分子坐标的相似程度的度量。 对于此示例,我们将测量内部原子坐标相对于最小化结构的变化。 具体来说,我们将分析丙氨酸原子(残基2)。
为了进行此分析,我们将使用cpptraj,这是一个用于处理MD轨迹的相当全面的分析程序。 该程序运行简单的用户编写的脚本,这些脚本选择要加载的轨迹,要运行的分析以及要保存的已处理轨迹或结构。
首先,我们将需要编写一个简单的cpptraj脚本来进行此分析。
使用gedit创建一个名为rmsd.cpptraj的cpptraj脚本。
trajin 02_Heat.nc
trajin 03_Prod.nc
reference 01_Min.ncrst
autoimage
rms reference mass out 02_03.rms time 2.0 :2
这是cpptraj脚本的简短摘要:
- Trajin 02_Heat.nc 加载轨迹02_Heat.nc
- reference 01_Min.ncrst 将结构01_Min.ncrst定义为参考结构
- center :1-3 mass origin 将残渣1-3按质量居中放置到系统原点
- image origin center 使用分子的质心将原子成像到原点
- rms reference mass out 02_03.rms time 2.0 :2 使用参考值计算质量加权RMSD并输出到02_03.rms
Cpptraj输入脚本文件
运行cpptraj
要实际运行cpptraj,我们必须在parm7,nc和参考ncrst文件所在目录的终端中再次运行它。
使用终端,将目录更改为Tutorial文件夹,然后运行cpptraj。 必须指定parm7文件和cpptraj脚本。
$ cd ~/Tutorial
$ $AMBERHOME/bin/cpptraj -p parm7 -i rmsd.cpptraj &> cpptraj.log
现在,我们的RMSD数据存储在文件02_03.rms中。 我们可以简单地使用xmgrace绘制该文件。
用xmgrace绘制RMSD。
$ xmgrace 02_03.rms
Cpptraj输出文件
cpptraj输出文件位于以下位置:
丙氨酸二肽MD RMSD相对于最小化的初始结构
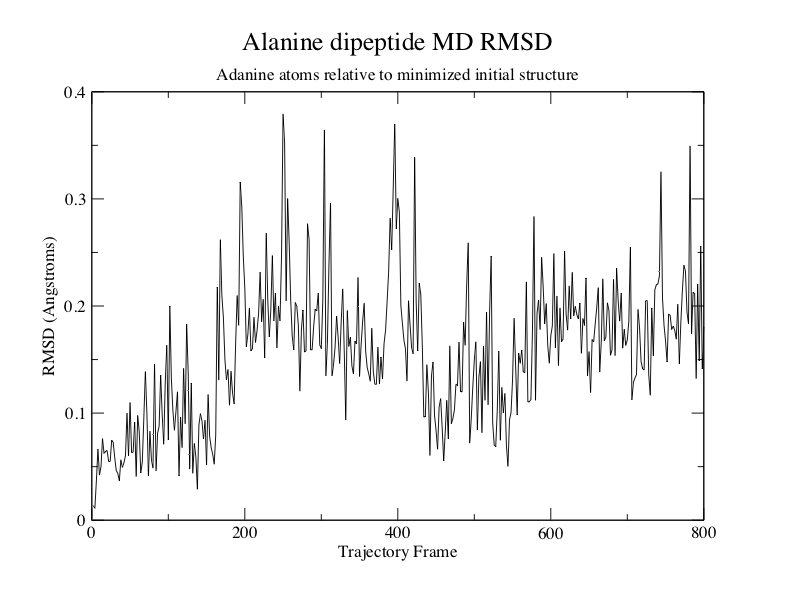
在该实例中,丙氨酸二肽中的n Phi / Psi二面角没有显着的构象变化。 这表明更稳定的肽结构。
九、结论
恭喜你。 现在,您已经运行了第一个完整的MD模拟并成功分析了结果。 这是用于设置,运行和分析自己的MD仿真的工作流的一个非常简单的示例。 如果您想了解更多信息,建议您在AMBER网站上完成其他教程。
十、其他
分析过程中涉及到的各种数据
参考资料
