【2.5.1.4】计算蛋白突变后的结合自由能变化
计算Ras-Raf的结合自由能,并使用丙氨酸扫描与残基突变为丙氨酸的突变型Ras-Raf复合物的结合能进行比较
1.将pdb文件设置为 leap-ready
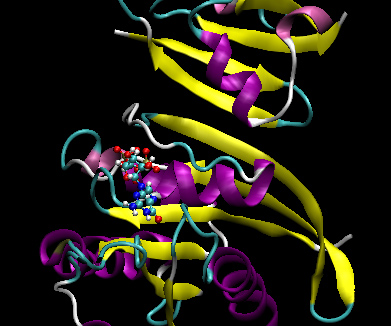
我们将在此模拟中建模的系统是人类H-Ras蛋白与C-Raf1的Ras结合域(Ras-Raf)之间的复合体,它是信号转导级联的核心。 这是RAS-RAF复合系统的部分平衡的,预先准备的pdb文件。
该结构包含ras和raf蛋白,以及生理必需的GTP核苷酸,如下图所示:
现在,我们必须准备要由tleap读取的突变pdb文件。我们强烈建议在运行任何模拟之前准备此pdb和突变体文件的拓扑以及在第1节中创建的初始拓扑文件,以保证一致的prmtop文件。在本教程中,我们选择将残基21(异亮氨酸,I21)突变为丙氨酸,因为这是在受体与配体之间的界面上发现的残基,对结合能具有显着影响。请注意,当前版本的代码仅支持向丙氨酸的突变。
因为仅在受体中发现了I21,所以我们不需要制作突变配体pdb文件。因此,我们只需要更改 ras-raf.pdb 和 ras.pdb。为此,您必须了解所涉及氨基酸的结构。异亮氨酸的侧链是-CH(CH3)CH2CH3。丙氨酸的侧链是-CH3。由于异亮氨酸的侧链比丙氨酸的侧链具有更多的原子,因此我们知道必须从pdb文件中删除原子及其相应的信息(名称,编号,坐标等)。此突变涉及除去除β-碳(CB)之外的所有侧链原子。在I21中,这意味着我们必须删除Ras-Raf和Ras pdb文件中与原子294至305相对应的行。我们不需要添加β-氢(HB)原子,因为tleap会将这些氢添加到正确的位置基于您为系统选择的特定库文件。最后,将残基21的所有剩余原子的残基名称从“ ILE”更改为“ ALA”。此过程将产生两个突变的pdb文件:ras-raf_mutant.pdb和 ras_mutant.pdb 。 RAS-RAF中的I21A突变如下所示。
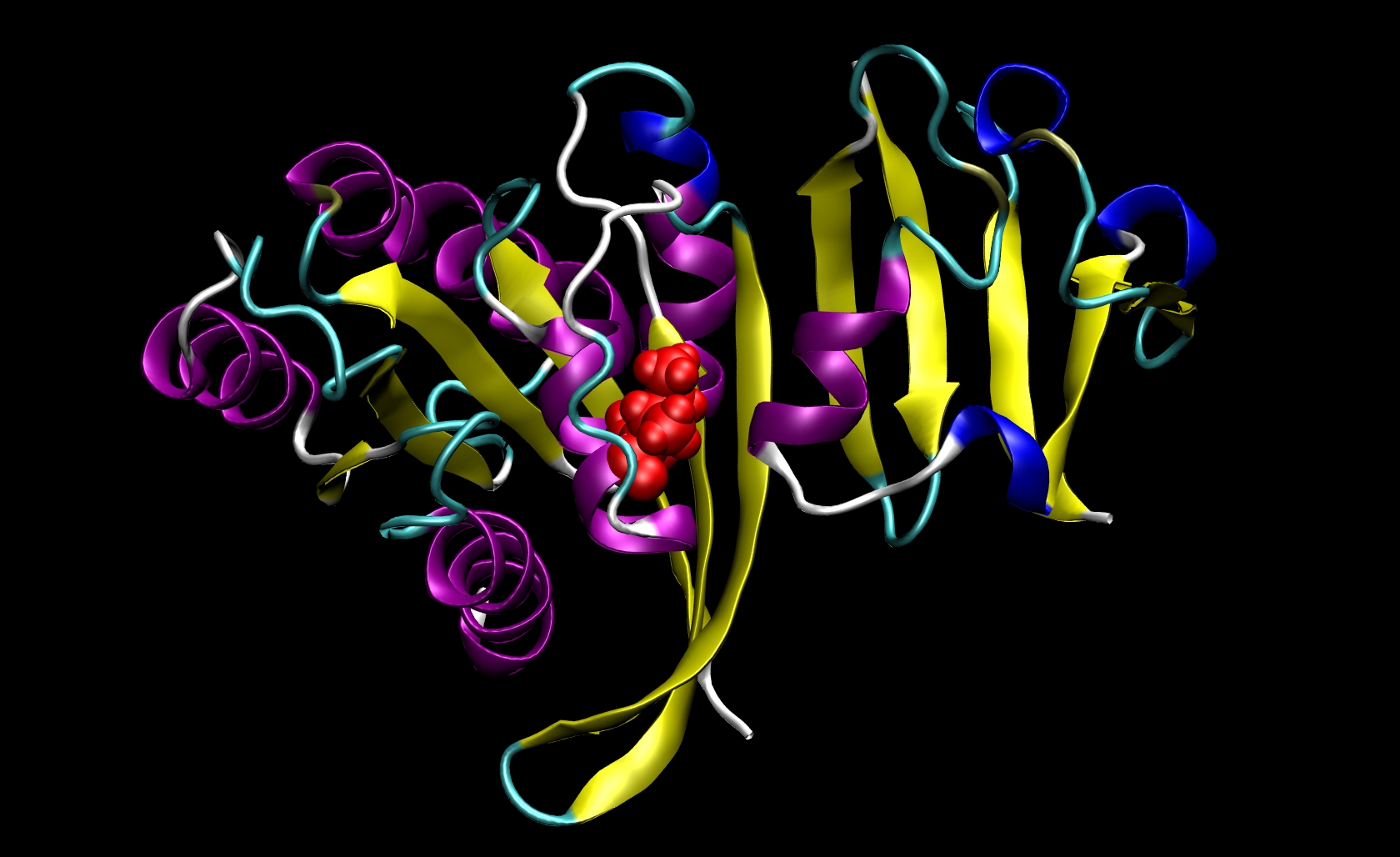
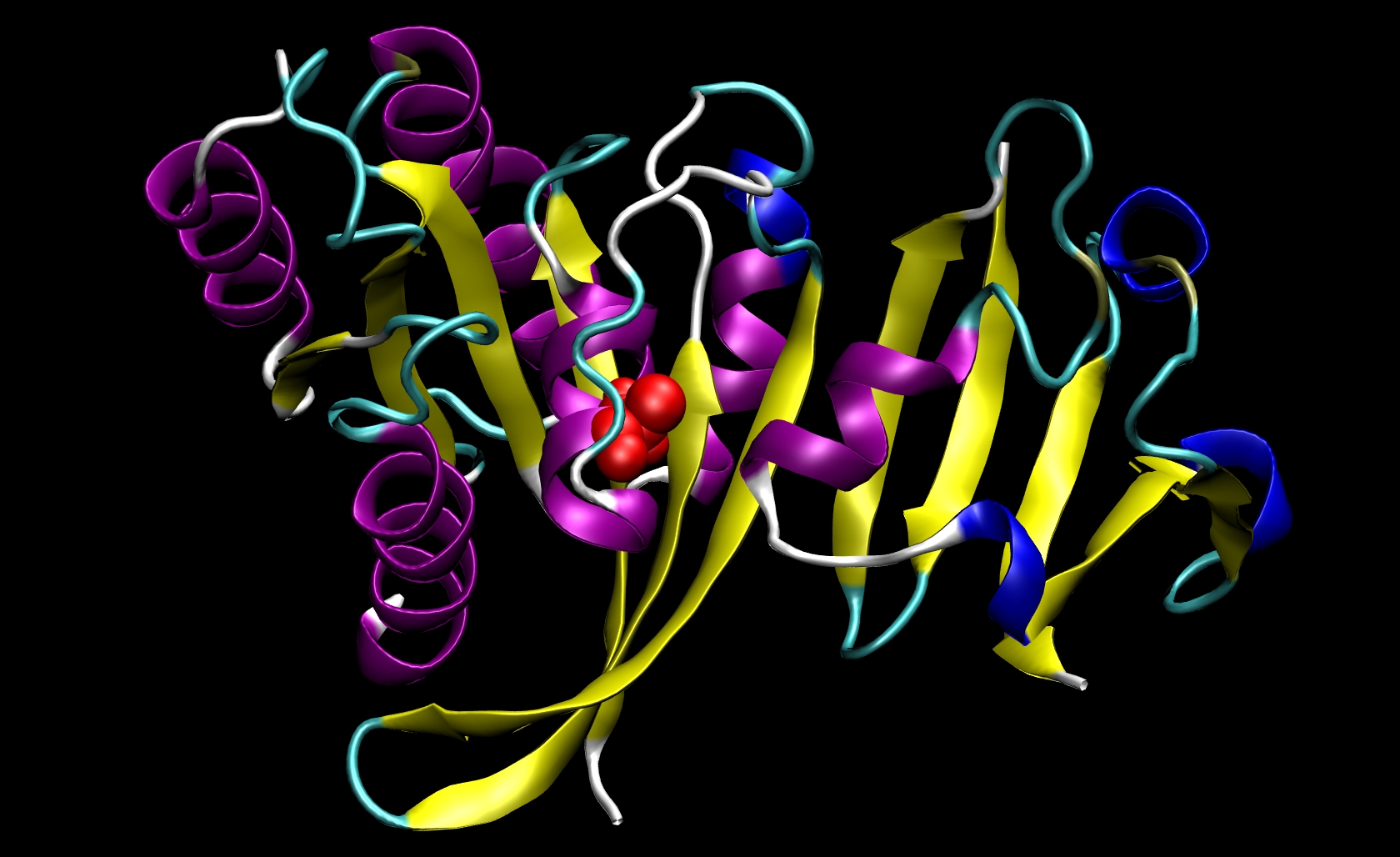
其他突变将能够遵循类似的程序,其中可以将CB之后但羰基碳(C)之前的原子组从pdb文件中删除。 请注意,一次计算只能执行一次突变。
2)建立起始拓扑和坐标文件,并运行模拟以获得平衡的系统。
现在已经制作了pdb文件,我们需要使用tleap为这些结构制作相应的拓扑和coordinate文件。 首先,我们将使文件对应于非突变复合体:
> $AMBERHOME/exe/tleap -s -f $AMBERHOME/dat/leap/cmd/leaprc.ff99SB
com = loadpdb ras-raf.pdb
ras = loadpdb ras.pdb
raf = loadpdb raf.pdb
saveamberparm com ras-raf.prmtop ras-raf.inpcrd
saveamberparm ras ras.prmtop ras.inpcrd
saveamberparm raf raf.prmtop raf.inpcrd
您还应该创建溶剂化复合物以运行MD模拟:
charge com
> Total unperturbed charge: -0.000000
> Total perturbed charge: -0.000000 (Hence there is no need to add counter ions)
solvatebox com TIP3PBOX 12.0
saveamberparm com ras-raf_solvated.prmtop ras-raf_solvated.inpcrd
现在,在退出tleap之前,您应该创建拓扑和坐标文件,根据刚创建的突变pdb文件:
com_mut = loadpdb ras-raf_mutant.pdb
ras_mut = loadpdb ras_mutant.pdb
saveamberparm com_mut rasraf_mutant.prmtop rasraf_mutant.inpcrd
saveamberparm ras_mut ras_mutant.prmtop ras_mutant.inpcrd
quit
我们仅创建了12个文件(六个.prmtop文件和六个.inpcrd文件)。非突变的.prmtop和.inpcrd文件已用于运行分子动力学(MD)模拟,以使用第1节和第2节中概述的过程来获得平衡的系统。
使用MMPBSA.py计算绑定自由能的重要文件是拓扑文件(非突变和突变),mdcrd文件是使用非突变拓扑和坐标文件运行的(ras-raf_alascan.tgz)
3)对Ras-Raf的结合自由能进行丙氨酸扫描计算。
现在,我们将计算复合物,受体和配体的相互作用能和溶剂化自由能,并对结果求平均值,以获得结合自由能的估计值。然后,在对mdcrd文件中的坐标进行了突变以与“野生型”结构进行比较之后,将对变异结构执行相同的计算。请注意,在本教程的这一部分中,我们将不会计算熵对绑定的贡献,因此严格来说,我们的结果将不是真正的自由能,但可用于与类似系统进行比较。请参见第3.5节中的示例,该示例使用正态模式分析(Nmode)计算系统的熵贡献,或取消注释常规部分中的最后一行以使用AMBER中的ptraj模块执行准谐波熵计算。
为了进行比较,我们将同时使用MM-GBSA方法和MM-PBSA方法进行结合能计算。这是通过以下MMPBSA.py输入文件完成的:
mmpbsa.in
sample input file for running alanine scanning
&general
startframe=1, endframe=50, interval=1,
verbose=1,
/
&gb
saltcon=0.1
/
&pb
istrng=0.100
/
&alanine_scanning
/
MMPBSA.py的输入文件被设计为类似于AMBER的sander模块中使用的mdin文件的设置。每个名称列表的开头均以&符号表示,后跟名称列表的名称。此外,反斜杠(/)或'&end’可用于结束名称列表。有关所有变量的完整列表,请参见amber手册。该输入文件分为四个名称列表:general,pb,gb和alanine_scanning。通用名称列表旨在指定并非特定于计算的特定部分而是特定于所有部分的变量。在此设置中,我们已将RAS定义为受体,将RAF定义为配体。 “ verbose”变量允许用户指定在计算结束时要删除的文件。有关mpi命令的更多信息,请参见手册或第3.4节。 “&gb”和“&pb”名称列表标记使脚本知道使用这些名称列表中定义的给定值执行MM-GBSA和MM-PBSA计算。 “ alanine_scanning”名称列表标记在脚本中初始化丙氨酸扫描。 &alanine_scanning名称列表中唯一可识别的输入变量是“ mutant_only”,该变量在手册中有更详细的描述。
$AMBERHOME/bin/MMPBSA.py -O -i mmpbsa.in -sp ras-raf_solvated.prmtop -cp rasraf.prmtop -rp ras.prmtop -lp raf.prmtop -y *.mdcrd -mc rasraf_mutant.prmtop -mr ras_mutant.prmtop
这将以交互方式运行脚本,并将计算进度打印到STDOUT,并将任何错误或警告打印到STDERR。 最后,一旦计算完成,将打印计时,显示计算的每个步骤所花费的时间。
命令行上的'-y * .mdcrd’告诉脚本读取工作目录中所有以'.mdcrd’结尾的文件,并将它们用作要分析的轨迹。
这是所有输出文件:ALASCAN_output.tgz。
该脚本使用ptraj创建三个未溶剂化的mdcrd文件(复合物,受体和配体),这是在GB和PB计算期间分析的坐标。 * .mdout文件包含所有指定帧的能量。 如果执行熵计算,则将平均pdb文件创建为用于最小化的结构。 除最终输出文件FINAL_RESULTS_MMPBSA.dat外,由MMPBSA.py创建的所有文件均应以前缀__MMPBSA_开头。
FINAL_RESULTS_MMPBSA.dat
| Run on Thu Feb 11 13:11:48 EST 2010
|Input file:
|--------------------------------------------------------------
|sample input file for running alanine scanning
| &general
| startframe=1, endframe=50, interval=1,
| verbose=1,
|/
|&gb
| saltcon=0.1
|/
|&pb
| istrng=0.100
|/
|&alanine_scanning
|/
|--------------------------------------------------------------
|Solvated complex topology file: ras-raf_solvated.prmtop
|Complex topology file: rasraf.prmtop
|Receptor topology file: ras.prmtop
|Ligand topology file: raf.prmtop
|Initial mdcrd(s): bigprod.mdcrd
|Mutant complex topology file: rasraf_mutant.prmtop
|Mutant receptor topology file: ras_mutant.prmtop
|Mutant ligand topology file: raf.prmtop
|
|Best guess for receptor mask: ":1-166"
|Best guess for ligand mask: ":167-242"
|Calculations performed using 50 frames.
|Poisson Boltzmann calculations performed using internal PBSA solver in sander.
|
|All units are reported in kcal/mole.
-------------------------------------------------------------------------------
-------------------------------------------------------------------------------
GENERALIZED BORN:
Complex:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -1863.7944 17.1704 2.4283
EEL -17200.7297 75.9366 10.7391
EGB -3142.2247 63.1977 8.9375
ESURF 91.3565 1.3938 0.1971
G gas -19064.5240 77.8536 11.0102
G solv -3050.8682 63.2131 8.9397
TOTAL -22115.3922 51.5332 7.2879
Receptor:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -1268.1888 14.2342 2.0130
EEL -11557.0773 71.7127 10.1417
EGB -2444.8629 54.9156 7.7662
ESURF 64.2843 1.1143 0.1576
G gas -12825.2661 73.1118 10.3396
G solv -2380.5786 54.9269 7.7678
TOTAL -15205.8447 36.8422 5.2103
Ligand:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -529.3090 9.4198 1.3322
EEL -4684.4720 36.1449 5.1117
EGB -1661.8286 26.5442 3.7539
ESURF 37.0493 0.6185 0.0875
G gas -5213.7811 37.3522 5.2824
G solv -1624.7794 26.5514 3.7549
TOTAL -6838.5604 25.6515 3.6277
Differences (Complex - Receptor - Ligand):
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -66.2966 4.2751 0.6046
EEL -959.1803 34.9190 4.9383
EGB 964.4668 32.9201 4.6556
ESURF -9.9770 0.3759 0.0532
DELTA G gas -1025.4769 35.1797 4.9752
DELTA G solv 954.4898 32.9223 4.6559
DELTA G binding = -70.9871 +/- 6.6875 0.9457
-------------------------------------------------------------------------------
-------------------------------------------------------------------------------
I21A MUTANT:
GENERALIZED BORN:
Complex:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -1855.4226 17.0765 2.4150
EEL -17210.2882 75.8866 10.7320
EGB -3145.1010 63.2477 8.9446
ESURF 91.8639 1.3913 0.1968
G gas -19065.7108 77.7842 11.0003
G solv -3053.2370 63.2630 8.9467
TOTAL -22118.9478 50.9582 7.2066
Receptor:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -1261.9126 14.1817 2.0056
EEL -11566.4419 71.5475 10.1183
EGB -2447.4831 55.0008 7.7783
ESURF 64.5090 1.1105 0.1570
G gas -12828.3545 72.9394 10.3152
G solv -2382.9741 55.0120 7.7799
TOTAL -15211.3287 36.2055 5.1202
Ligand:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -529.3090 9.4198 1.3322
EEL -4684.4720 36.1449 5.1117
EGB -1661.8286 26.5442 3.7539
ESURF 37.0493 0.6185 0.0875
G gas -5213.7811 37.3522 5.2824
G solv -1624.7794 26.5514 3.7549
TOTAL -6838.5604 25.6515 3.6277
Differences (Complex - Receptor - Ligand):
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -64.2010 4.0841 0.5776
EEL -959.3742 34.9114 4.9372
EGB 964.2108 32.9092 4.6541
ESURF -9.6943 0.3800 0.0537
DELTA G gas -1023.5752 35.1495 4.9709
DELTA G solv 954.5164 32.9114 4.6544
DELTA G binding = -69.0588 +/- 6.5302 0.9235
-------------------------------------------------------------------------------
-------------------------------------------------------------------------------
RESULT OF ALANINE SCANNING: (I21A MUTANT:) DELTA DELTA G binding = 1.9283 +/- 9.3470
-------------------------------------------------------------------------------
-------------------------------------------------------------------------------
POISSON BOLTZMANN:
Complex:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -1863.7944 17.1704 2.4283
EEL -17200.7297 75.9366 10.7391
EPB -3227.2145 64.4523 9.1149
ECAVITY 68.4754 0.7567 0.1070
G gas -19064.5240 6061.1875 857.1813
G solv -3158.7391 64.4568 9.1156
TOTAL -7522.2032 51.2973 7.2545
Receptor:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -1268.1888 14.2342 2.0130
EEL -11557.0773 71.7127 10.1417
EPB -2485.3559 54.5638 7.7165
ECAVITY 47.5088 0.4610 0.0652
G gas -12825.2661 5345.3320 755.9441
G solv -2437.8471 54.5658 7.7168
TOTAL -5118.8075 38.9610 5.5099
Ligand:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -529.3090 9.4198 1.3322
EEL -4684.4720 36.1449 5.1117
EPB -1684.5802 28.2572 3.9962
ECAVITY 28.1687 0.3939 0.0557
G gas -5213.7811 1395.1865 197.3092
G solv -1656.4114 28.2599 3.9966
TOTAL -2313.4381 24.9082 3.5225
Differences (Complex - Receptor - Ligand):
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -66.2966 4.2751 0.6046
EEL -959.1803 34.9190 4.9383
EPB 942.7215 33.8861 4.7922
ECAVITY -7.2022 0.3069 0.0434
DELTA G gas -1025.4769 1237.6138 175.0250
DELTA G solv 935.5194 33.8875 4.7924
DELTA G binding = -89.9575 +/- 8.2480 1.1664
-------------------------------------------------------------------------------
-------------------------------------------------------------------------------
I21A MUTANT:
POISSON BOLTZMANN:
Complex:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -1855.4226 17.0765 2.4150
EEL -17210.2882 75.8866 10.7320
EPB -3229.1405 64.8100 9.1655
ECAVITY 68.5521 0.7596 0.1074
G gas -19065.7108 6050.3755 855.6523
G solv -3160.5884 64.8144 9.1661
TOTAL -7520.1586 50.6710 7.1660
Receptor:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -1261.9126 14.1817 2.0056
EEL -11566.4419 71.5475 10.1183
EPB -2487.5603 54.6289 7.7257
ECAVITY 47.6466 0.4632 0.0655
G gas -12828.3545 5320.1609 752.3844
G solv -2439.9137 54.6309 7.7260
TOTAL -5118.8820 38.4370 5.4358
Ligand:
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -529.3090 9.4198 1.3322
EEL -4684.4720 36.1449 5.1117
EPB -1684.5802 28.2572 3.9962
ECAVITY 28.1687 0.3939 0.0557
G gas -5213.7811 1395.1865 197.3092
G solv -1656.4114 28.2599 3.9966
TOTAL -2313.4381 24.9082 3.5225
Differences (Complex - Receptor - Ligand):
Energy Component Average Std. Dev. Std. Err. of Mean
-------------------------------------------------------------------------------
VDWAALS -64.2010 4.0841 0.5776
EEL -959.3742 34.9114 4.9372
EPB 942.9999 34.0350 4.8133
ECAVITY -7.2632 0.3107 0.0439
DELTA G gas -1023.5752 1235.4872 174.7243
DELTA G solv 935.7367 34.0364 4.8135
DELTA G binding = -87.8385 +/- 8.0665 1.1408
-------------------------------------------------------------------------------
-------------------------------------------------------------------------------
RESULT OF ALANINE SCANNING: (I21A MUTANT:) DELTA DELTA G binding = 2.1190 +/- 11.5368
-------------------------------------------------------------------------------
-------------------------------------------------------------------------------
统计文件的开头包括日期/时间,基于给定值和文件的任何警告,mmpbsa.in文本,脚本使用的文件,分析的帧数以及哪个PB solver(如果有)用过的。统计信息文件的其余部分包括GB的所有平均能量,标准偏差和均值的标准误差,其后是PB。在每个部分之后,结合的ΔG随误差值一起给出。在每种方法之后,报告了结合的ΔΔG,这表明突变对复合物的结合的ΔG具有相对影响。特定的突变也打印在文件的末尾。在这种情况下,我们将残基21从异亮氨酸突变为丙氨酸(即I21A)。该文件中不同能量术语的含义如下:
VDWAALS = MM的van der Waals贡献。 EEL =由MM力场计算出的静电能。 EPB / EGB =分别由PB或GB计算得出的对溶剂化自由能的静电贡献。 ECAVITY =通过经验模型计算得出的对溶剂化自由能的非极性贡献。 DELTA G binding=根据上述条件计算得出的最终估计结合自由能。 (千卡/摩尔)
请注意,尚未报告总气相能,因为使用单轨方法,受体和配体的键合势项的值应恰好抵消了配合物的键合势项的值。如果能量未在允许的公差范围内消除,则会出现错误消息。
人们通常期望找到一种非常有利的静电能和一种不利的溶剂化自由能。这象征着人们必须使用的能量来使结合颗粒去溶剂化并对齐其结合界面。
参考资料
