【6.3.1】利用minion_qc绘制质控图
前面我们提到过利用guppy进行碱基识别后生成一个sequencing_summary.txt文件,这个文件是列表格式的统计结果,R语言最喜欢列表格式了,直接使用R可以对这个列表进行统计绘图。其实如果使用官方的MinKNOW软件自带这些绘图功能,如果是自己进行的碱基识别,可以使用minion_qc来进行统计绘图,非常的容易,其实后面我们介绍的NanoPlot也可以使用这个文件绘图。
一、软件安装
软件官网:https://github.com/roblanf/minion_qc
这个工具其实就是一个R脚本,在R语言环境下直接运行就行。安装和使用都非常容易。
#下载软件
wget https://raw.githubusercontent.com/roblanf/minion_qc/master/MinIONQC.R -O MinIONQC.R
下载完成之后应该还不能直接运行,如果你的系统环境里缺少这些R包,还需要将一下这些包安装一下。
install.packages(c("data.table",
"futile.logger",
"ggplot2",
"optparse",
"plyr",
"readr",
"reshape2",
"scales",
"viridis",
"yaml"))
二、软件使用
软件的使用非常容易,输入文件就为guppy生成的sequencing_summary.txt。
Rscript MinIONQC.R -i sequencing_summary.txt -o output -p 2
软件运行的还比较快,因为没有太大的计算量,最终结果会在output中,包括如下内容:
channel_summary.png
flowcell_overview.png
gb_per_channel_overview.png
length_by_hour.png
length_histogram.png
length_vs_q.png
q_by_hour.png
q_histogram.png
reads_per_hour.png
summary.yaml
yield_by_length.png
yield_over_time.png
2.2 结果解析
结果并不难理解,主要是一些图,根据图的名字就能大概能知道每个图的意思,我们通过这些图来看一下测序情况。这里面比较重要的就是看一下长度分布,质量值分布等。下面拿几个具体图展示一下。

图1 read长度分布
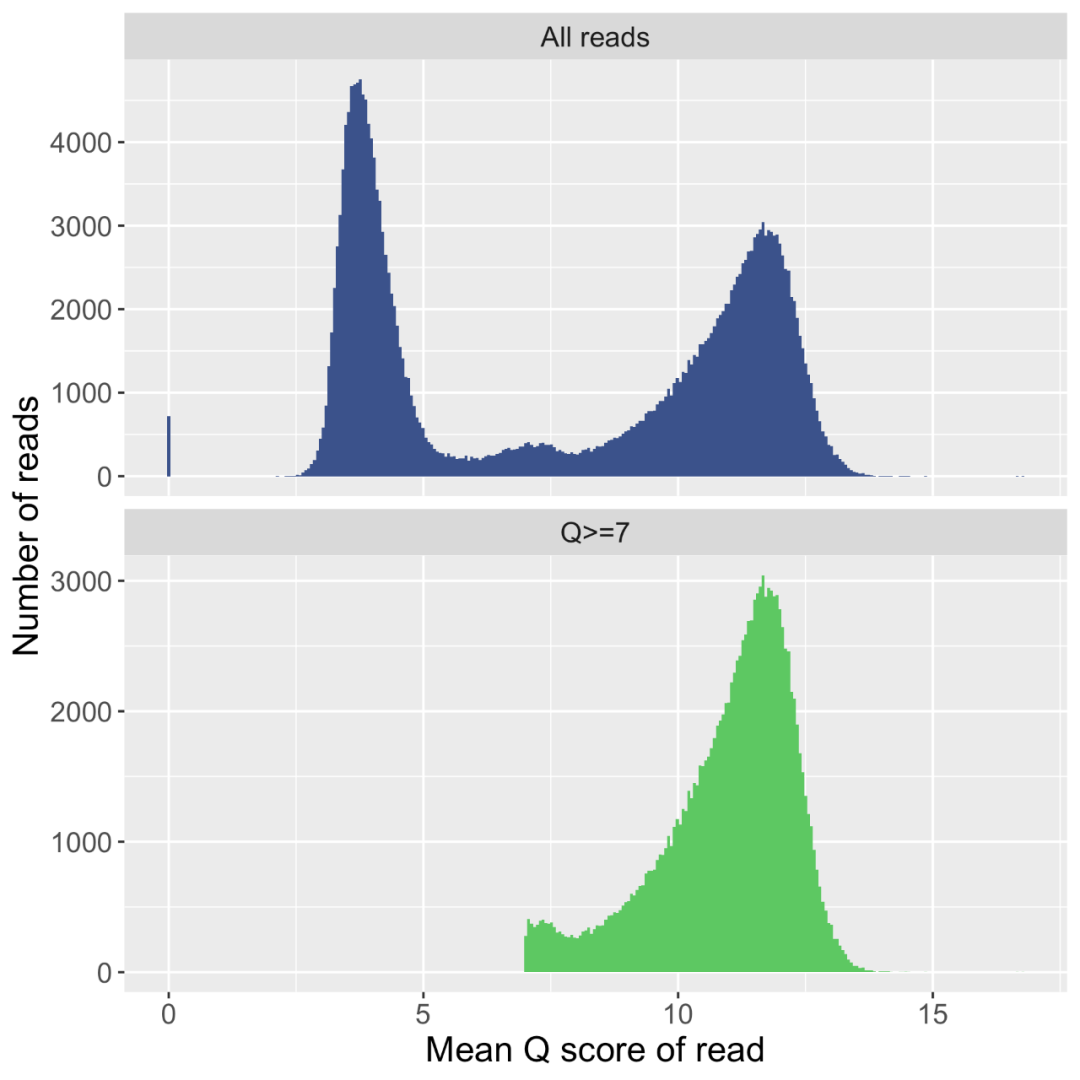
图2 reads平均质量值分布,目前Q7 作为也临界标准,大约Q7的越多越好

图3 reads长度与质量二图散点图
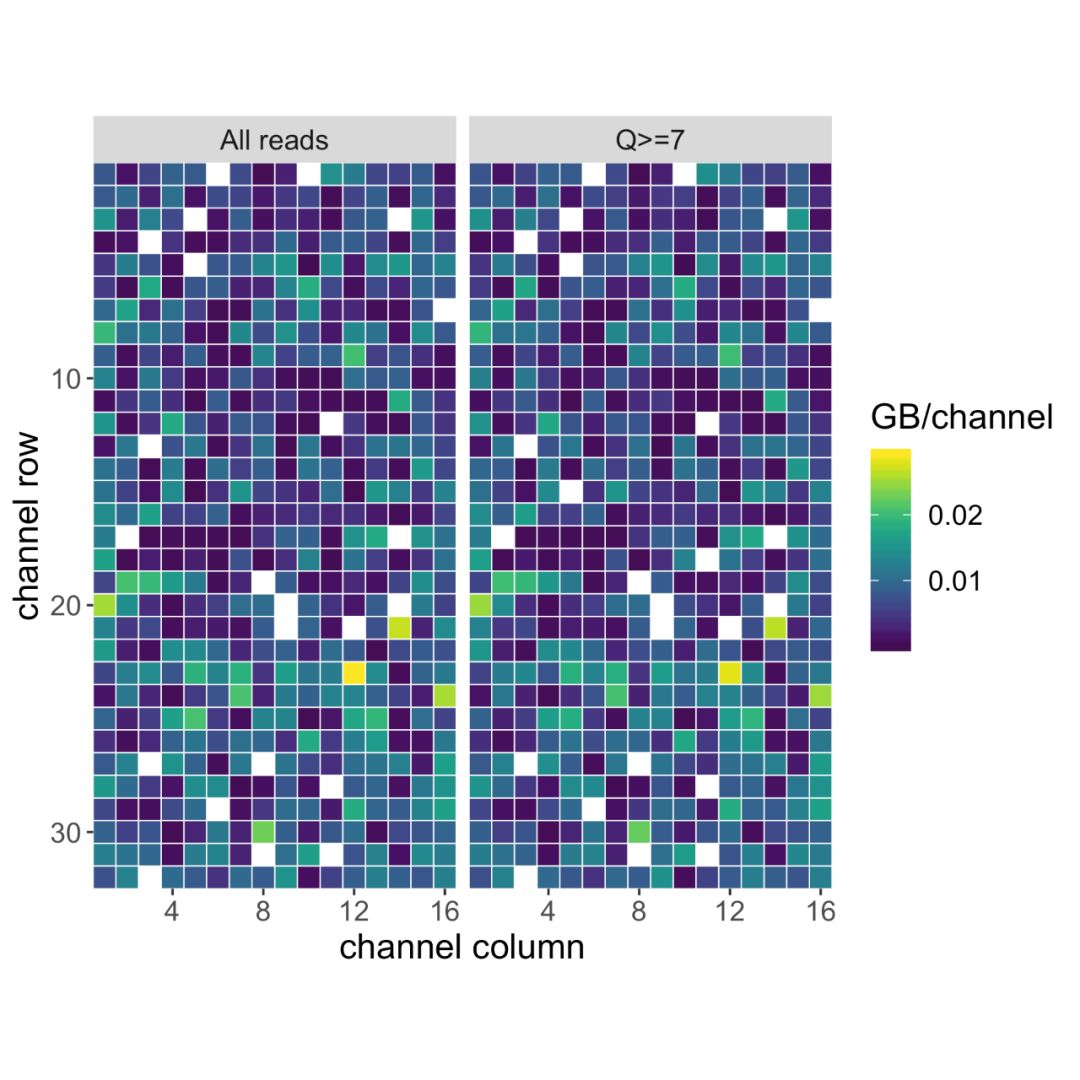
图片图4 每个纳米孔产数据情况统计,颜色越亮,产数据越多
参考资料
