【3.2】NGS中重要的几个指标(Metrics)
通过target capture的方法获得序列然后测序的方法,最后我们需要衡量一下真个富集的Panel到底咋样。
这里介绍一下IDT人为的几个重要的指标
- Unique vs. Duplicate
- On Target
- Coverage Depth
- Uniformity of Coverage
一、Unique vs. Duplicate
Duplicate reads来自同一DNA片段的PCR拷贝测序出来的多个结果。当随机测序DNA片段的时候,一些duplication是不可避免的。大多数测序分析流程是去掉PCR dulicates的,但是因此也容易去掉一些有用的信息,例如,罕见的缺失突变以及Repeat区域的测序。PCR反应是不用做去除duplicates的哈
二、 On TARGET
有多少序列能够map到指定区域以及有多少序列碱基能map到目标区域
Method #1: % On Target =_On Target Reads/_Total Aligned Reads
Method #2: % On Target = _On Target Bases/_Total Aligned Bases
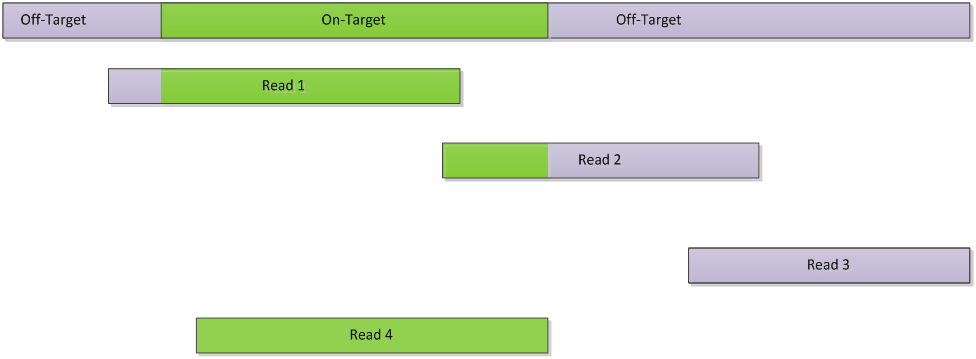
在目标区域的碱基的判断标准就是这个点比对到目标区域上。 一条read中只要有一个碱基能比对到目标区域,就认为它在目标区域上(如上图,read1,read2,read4为比对到目标片段,read3没有比对上)
三. Coverage Depth
Coverage depth代表一个区域被测序到的次数,被测序的次数越高,其coverage depth越高。 不同的项目中,对测序深度的要求不一样。
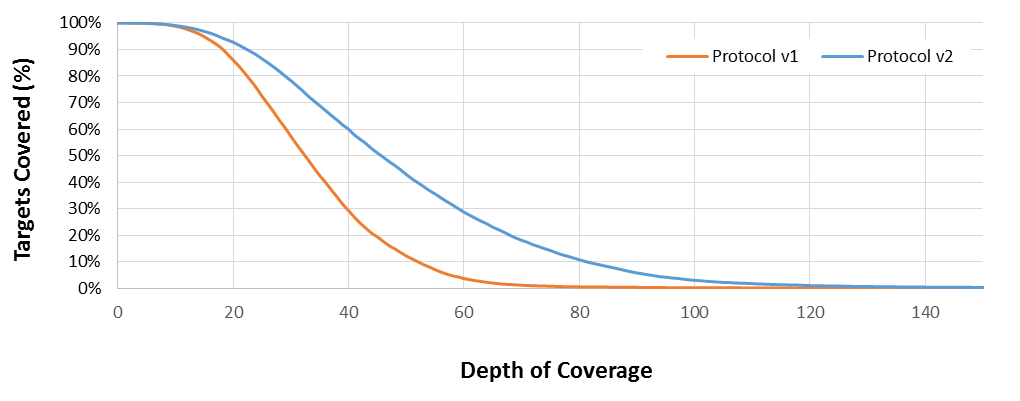
for detecting mutations with 5% (0.05) allele frequency, you would need 400X coverage depth.
根据你的目的来决定Depth。一般的要求是30-50X,???
四. Uniformity of coverage
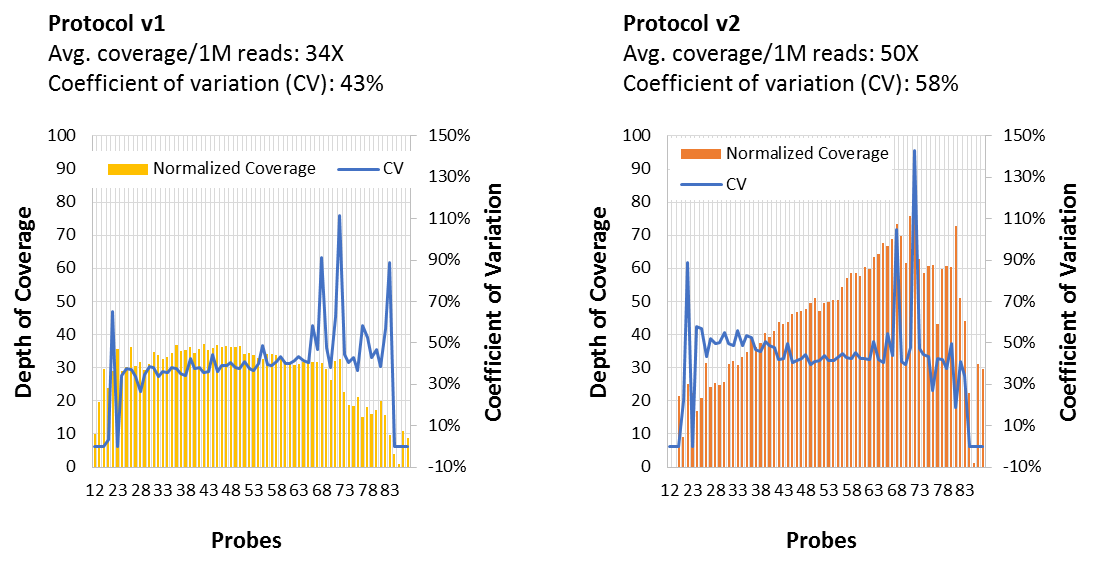
Unifomity是检验测序好坏的一个很重要的指标,体现在每个amplicons的表现是否一致或者是否在我规定的范围内。
IDT主要有两种方式来衡量uniformity,一种是计算大于平均深度的0.2, 0.5, 1倍的比例。这种方法有利于找到低丰度的那些序列,但是不能很好的体现高丰度的序列。
另一种方式就是每个amplicon的CV(coefficient of variation , which is the standard deviation divided by the mean),值越小,uniformity就越好罗。当然可以结合着GC来一起看。
参考资料:
http://www.illumina.com/science/education/sequencing-coverage.html
