【4.3.5】基于结构预测非连续BCE--DiscoTope-1.0
官网:http://tools.iedb.org/discotope/
发现不连续的B细胞表位是疫苗设计中的主要挑战。先前的表位预测方法主要是基于蛋白质序列,并不是很有效。在这里,我们介绍 DiscoTope,一种使用蛋白质三维结构数据进行不连续表位预测的新方法。该方法基于氨基酸的统计信息,空间信息和通过抗体/抗原蛋白复合物的X射线晶体学确定的不连续表位的汇编数据集中的表面可及性。 DiscoTope是第一种明确关注不连续表位的方法。我们表明,基于新结构的方法比仅基于序列信息的方法具有更好的预测不连续表位残基的性能,并且它可以成功预测已由不同技术鉴定的表位残基。 DiscoTope检测到不连续表位中的15.5%残基,特异性为95%。在这种特异性水平上,用于预测线性B细胞表位的常规Parker亲水性量表只能识别出不连续表位中的残基的11.0%。 DiscoTope方法的预测可以指导合理的疫苗设计和诊断工具开发中的实验表位作图,并可能导致更有效的表位鉴定。
一、前言
疫苗设计的主要任务是选择和设计包含能够诱导有效免疫反应的抗体结合表位(B细胞表位)的蛋白质。可以通过相关蛋白质或蛋白质区域中的表位预测来辅助选择。此外,对B细胞表位的预测可能有助于鉴定已使用基于抗体亲和力结合的实验技术进行分析的蛋白质中的表位,例如Western blotting,免疫组织化学,放射免疫测定(RIA)和酶联免疫吸附测定(ELISA) 。
现有的大多数预测B细胞表位的方法仅使用蛋白质序列作为输入,最适合预测由连续氨基酸组成的表位(线性表位)(Hopp和Woods 1981; Parker等人1986; Jameson和Wolf(1988); Debelle等(1992); Maksyutov和Zagrebelnaya(1993); Alix(1999); Odorico和Pellequer(2003)。通常,这些方法是基于使用许多氨基酸倾向量表对亲水性,柔韧性,β-转角和表面可及性的预测。线性表位上存在大量数据(Leitner等,2003; Saha等,2005; Toseland等,2005),因为注释可以通过测量抗原肽片段与抗体的结合来完成。但是,这种注释方法可能导致注释错误,因为即使肽的某些残基不与抗体相互作用,肽也可以特异性结合抗体。预测线性表位仍然是一项艰巨的任务,并且可获得的预测准确性相当差(Van Regenmortel和Pellequer 1994; Van Regenmortel 1996; Blythe和Flower 2005)。然而,隐式马尔可夫模型和由Parker等人构建的亲水性尺度的结合。 (1986)最近导致线性B细胞表位的预测有所改善(Larsen等,2006)。
据估计,> 90%的B细胞表位是不连续的,即由在病原体蛋白质序列中远距离分离并通过蛋白质折叠而接近的区段组成(Barlow等,1986; Van Regenmortel 1996) )。不连续表位的鉴定是困难的,因为必须在天然抗原结构的背景下进行完整的分析。鉴定不连续表位的最有用和最准确的方法是通过X射线晶体学确定抗原-抗体复合物的结构(Fleury等,2000; Mirza等,2000)。由目前可用的X射线结构衍生的不连续表位的使用存在两个主要问题:
- 第一,与线性表位相比,不同抗原中不连续表位的可用数据大大减少;
- 其次,已经研究了很少的抗原来完全鉴定同一抗原中的各种不连续表位。
数据集中没有发现的未检测到的表位的存在可能使开发更好的预测算法变得更加困难,因为它们会影响测得的性能。但是,有关抗体-抗原复合物的详细结构知识正在增长,可以对各种抗原中的不连续表位进行更广泛的分析,并开发出更好的预测方法。
表面暴露与B细胞表位之间的相关性已为人所知(Novotny等,1986; Thornton等,1986)。最近,已经发表了两种使用蛋白质结构和表面暴露预测B细胞表位的新方法(Kulkarni-Kale等,2005; Batori等,2006)。但是,这些使用蛋白质结构作为输入的新方法都没有将主要精力放在不连续的表位上。
在这里,我们提出了一种预测方法,用于预测不连续的B细胞表位中的残基。 DiscoTope结合使用了氨基酸统计,空间信息和表面暴露。在来自抗体/抗原蛋白复合物的76个X射线结构的不连续表位的汇编数据集上进行训练。我们将DiscoTope的性能与Parker亲水性标度(Parker et al。1986)进行比较,以与经典的基于序列的方法进行比较,该方法最近被证明可以很好地预测线性表位(Larsen et al。2006)。 另外,我们将性能与基于使用NACCESS程序在抗原结构上测得的表面可及性的预测进行比较(Hubbard和Thornton,1993年)。我们证明DiscoTope通常是此处描述的所有方法中性能最好的。最后,我们提出了在疟疾蛋白顶膜抗原1(AMA1)中表位的描述,其中DiscoTope成功地预测了已使用各种实验或序列分析技术鉴定出的表位残基。
二、结果
2.1 不连续B细胞表位的性质
为了为预测方法的开发和评估奠定良好的基础,我们从抗体和蛋白质抗原之间的复合物的76个X射线结构中汇编了一个不连续的表位数据集。我们分析了数据集,以找到每个表位的残基数,表位中每个连续序列的残基数以及每个表位的最长连续序列的分布。这些分布如图1所示。每个表位的残基总数为9至22,> 60%的表位由14至19个残基组成(图1A)。具有单个表位残基的片段代表数据集中528个片段的> 45%(图1B)。每个表位中已鉴定残基的最长序列延伸范围为3至12个残基,> 75%的表位包含最大长度为4至7个残基的序列延伸(图1C)。这些发现证实,数据集中的大多数表位确实是不连续的,并且由形成抗体结合区的抗原序列的小部分组成。

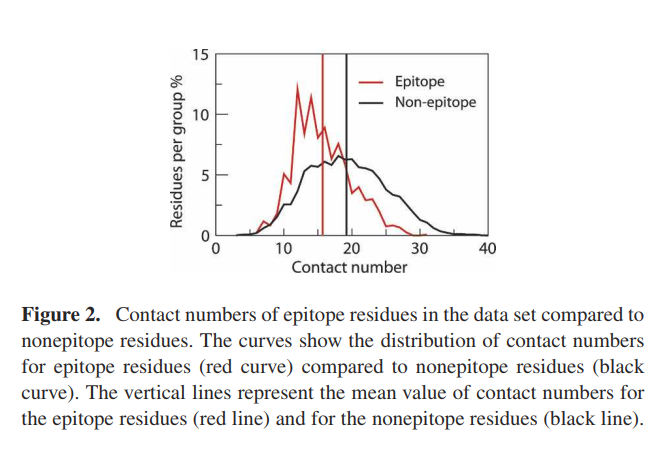
通过确定每个残基的分子内Cα原子接触数,对数据集进行了表面暴露分析(图2)。低接触数与抗原结构的表面附近或突出区域中的定位相关。 t检验表明,与非表位残基相比,在数据集中被识别为表位一部分的残基的接触数量要低得多(P <10-5)。表位残基的平均接触数和平均值的标准误为15.7±0.12,非表位残基的平均接触数和平均值的标准误为19.2±0.05(见图2,垂直线)。表位位于暴露或突出区域的发现与先前对B细胞表位的分析相一致(Novotny等人1986; Thornton等人1986)。如图2所示,两个分布是重叠的。这很可能是由于数据集的注释不完整,或者由于联系号以外的其他因素在定义表位中也很重要。
为了开发和评估预测方法,将数据集中的76个抗原分为25个非同源组(更多详细信息,请参见材料和方法)。 从这25个组中,构建了五组(每组五个),并用于五重交叉验证的训练和评估,以避免对相似抗原进行优化和评估。
2.2 根据表位数据集计算的对数比 Log‐odds ratios calculated from the epitope data set
我们通过计算数据集肽段的对数比来分析数据集表位和非表位中氨基酸的统计数据。选择了基于肽的相似性降低方法,以避免对数比值偏向数据集中的高度冗余表位。数据集中具有高相似性的肽的权重低于具有低相似性的肽,因此,肽的长度在对数比的推导中起着重要作用。我们使用原始的对数比作为表位倾向来预测训练集中的表位,并发现9个残基的肽长度是最佳的。
表1显示了从76种蛋白质的总数据集中减少了同源性的肽段计算出的表位对数比。在这20个氨基酸中,天冬酰胺(N),精氨酸(R),脯氨酸(P)和赖氨酸(K)的对数比最高,这意味着与数据集的非表位相比,它们在表位中的表达过多。半胱氨酸(C),丙氨酸(A),亮氨酸(L),缬氨酸(V)和苯丙氨酸(F)的对数比非常低,并且相应地在表位中代表性不足。有趣的是,我们发现了Parker亲水性标度和对数奇数比之间的一些差异(表1)。例如,疏水性最大的残基色氨酸(W)的对数奇数比没有特别低。最亲水的残基,天冬氨酸(D)和谷氨酸(E),具有相对中等的对数比。精氨酸(R)和脯氨酸(P)的对数比最高,但排在Parker亲水性量表的中间。半胱氨酸(C)和丙氨酸(A)的排名接近Parker量表的中部,但对数比最低。

2.3 B细胞表位预测的非组合方法的评估
为了测试不连续表位上的接触数的预测强度和对数比的表位倾向量表,我们使用了不同评估集上接收者操作员曲线(AUC)平均值下的面积(请参见材料和方法中的详细信息)。我们还用对数奇数比的连续平均值作为预测得分进行了测试,类似于Parker等人为亲水性标度推荐的方法。 (1986)。根据对训练集的预测性能,发现对数奇数比进行顺序平均的最佳窗口大小为9个残差(数据未显示)。
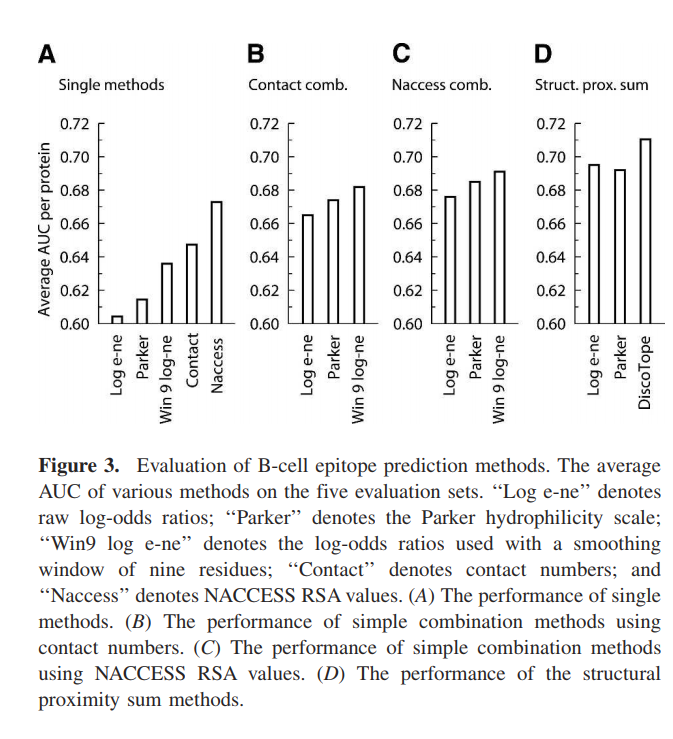
我们发现,在不连续的表位上,顺序平均使用的表位对数比比顺序平均的Parker亲水性量表表现更好(图3A)。原始表位的对数倾向量表在评估集上的平均表现为0.604。使用九个残基的顺序平均值平滑对数比,将性能提高到0.636。使用Parker标尺,具有七个残渣的平滑窗口,性能为0.614。与基于倾向量表的方法相比,基于接触数和NACCESS相对表面积(RSA)值的方法分别具有0.647和0.673的更高性能(图3A)。
2.4 表位预测的组合方法
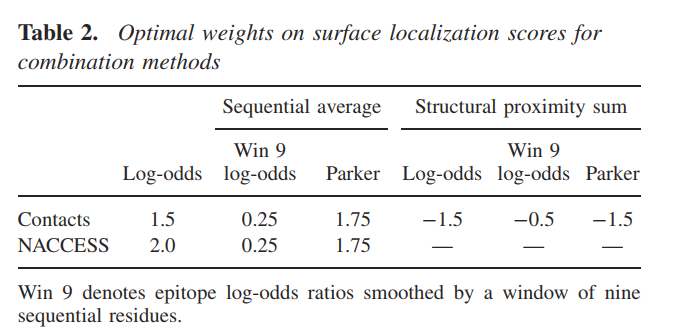
我们还使用了基于接触数或NACCESS RSA的表面定位值,结合表位对数比或Parker亲水性标度,通过表面定位值对表位残基的预测进行了测试。 一种组合方法是使用基于顺序信息(对数比或亲水性得分)的表面定位度量和方法的加权预测得分的总和。 第二种方法是通过对空间接近度的对数比值,顺序平均对数比值或残基的Parker规模得分进行求和并加上接触数得出预测得分来进行测试的。 对于每种组合,我们通过优化在平均AUC中测得的训练集的预测性能来估计表面定位分数的相对权重。 表2列出了优化的权重。
通过根据评估数据集的预测计算平均AUC来测试组合方法的预测性能(图3B–D)。 Parker量表,原始对数比和平滑对数比的简单线性组合与基于结构的方法(联系号和NACCESS RSA值)总体上改善了性能(图3B,C)。使用原始对数比的组合方法,对于具有接触数的组合,其性能为0.665;对于具有NACCESS RSA值的组合,其性能为0.676。帕克方法的线性组合对于接触数组合具有0.674的性能,对于NACCESS RSA组合具有0.685的性能。结合使用平滑对数比和接触数可以得到0.682的性能。简单线性组合的最佳方法是将平滑对数比与NACCESS RSA相结合。该方法在评估集上的性能为0.691。
基于倾向量表的结构接近总和与接触数相结合的方法在评估集上表现出最佳性能(图3D)。基于Parker预测值并结合接触数的结构邻近总和方法的性能为0.692。相应的使用原始对数比的结构邻近总和方法的性能为0.695。评估数据集上表现最佳的方法是顺序平滑的表位对数比与接触数相结合的结构接近度总和。结果表明,该方法的性能为0.711,明显优于使用原始对数比(P = 0.040)进行结构平均的方法。该方法也显着优于Parker方法(P = 0.007),略高于NACCESS RSA方法(P = 0.105)。我们称此方法为DiscoTope。
2.5 DiscoTope方法用于不连续B细胞表位预测的分析
我们决定进一步分析Parker亲水性,NACCESS RSA和DiscoTope预测,以更详细地比较这些方法的性能。 根据多种选定的特异性比较了三种方法的敏感性(表3)。 在表3中,我们另外列出了预测阈值,以促进对B细胞表位预测使用所有三种方法的通用。 对于所有五个特异性水平,DiscoTope具有三种方法中最高的灵敏度。 在95%的特异性水平下(这意味着只有5%的假阳性预测),DiscoTope检测到了15%的表位。 对于95%和90%的特异性水平,Parker方法的灵敏度高于NACCESS RSA方法。 这与五个评估集上的平均AUC值形成对比,NACCESS方法的平均AUC值高于Parker方法的平均AUC值(图3A)。

为了分析这三种方法在不同抗原组上的性能,我们比较了25个非同源抗原组中每个组的预测AUC值(图4)。对于数据集中的大多数抗原组,DiscoTope方法的性能优于Parker方法(图4A)。但是,在八组抗原中,使用Parker方法可以更准确地预测表位残基。对于NACCESS RSA方法,观察到了相同的趋势,其中Parker方法对12组的效果最佳(图4B)。 DiscoTope和NACCESS RSA方法的比较表明,即使25个组的平均AUC值在DiscoTope方法中最高,但NACCESS RSA方法在10个抗原组中表现最佳(图4C)。我们发现DiscoTope和NACCESS RSA方法有六个共同点,其中Parker方法表现最佳。这些组由PDB抗原条目1JPS,2JEL,1TQB,1AR1、1OAZ,1EO8表示。两种基于表面可及性的方法均具有比Parker规模法更低的性能这一事实表明,所测得的单个抗原链的表面可及性不足以预测所有类型抗原中的表位。六个组中的三个(由抗原1JPS,1AR1和1EO8表示)包含具有细长结构的抗原。此外,1JPS,1AR1和1EO8的抗原都是与膜结合的较大生物复合物的亚基(Ostermeier等,1997; Fleury等,2000; Faelber等,2001)。也许不足为奇,DiscoTope方法采用的单抗原链方法显然无法正确测量此类蛋白质中所有残基的表面可及性。例如,图1中显示了1AR1抗原的结构。在图中,抗原中具有最低接触数的大多数残基不在表位附近(图5A,B)。实际上,抗原中具有最低接触数的30%残基中只有一个表位残基。 1AR1抗原是跨细胞色素C氧化膜的膜的一个亚基(图5C),具有低接触数的残基的最大连续区域对应于被称为跨膜的蛋白质区域(Ostermeier等1997) )。


2.6 顶端膜(apical membrane)抗原1中的B细胞表位残基的预测
为了评估我们使用X射线晶体学以外的其他方法定位的B细胞表位的方法,我们测试了AMA1胞外域结构上DiscoTope的预测(Bai等人2005; Pizarro等人2005)。从PDB衍生的不连续表位的数据集中没有AMA1表位。然而,单克隆抗体Mab1F9和Mab4G2识别的两个独立的表位已通过实验在AMA1胞外域定位:Mab1F9抗原表位是通过肽的噬菌体展示和E197的点突变进行定位的(Coley等,2006)。不连续的Mab4G2表位通过9个残基的点突变进行了详细定位(Pizarro et al。2005)。另外,Bai等(2005年)已将五个残基(包括E197和结构相同区域中的其他残基)归类为恶性疟原虫AMA1序列中的高度多态性。已经提出,多态性是由对抗原的选择压力以避免宿主免疫系统引起的。我们使用的DiscoTope预测阈值为-4.7,对应于90%和24%敏感性的特异性(表3)。在AMA1中,311个残基中的43个被预测为表位残基。大多数预测的表位残基都聚集在AMA1结构的三个单独区域中(图6)。 DiscoTope成功地鉴定了1F9表位的8个残基中的两个,这些残基是通过噬菌体展示法绘制的(D196和E197)。在不连续的4G2表位中,除D348外的所有9个残基均被预测为表位的一部分。 Bai等人描述的所有五个高度多态性残基(2005)被预测位于表位。因此,DiscoTope成功地预测了AMA1的表位残基,这些残基已通过使用多种方法进行了定位。

三、讨论区
在本文中,我们介绍了DiscoTope,这是一种预测位于不连续B细胞表位中的残基的新方法。 DiscoTope将蛋白质结构的表面定位和空间特性与新颖的表位倾向标度结合在一起。根据接触数的简单加权总和与空间上接近的残基的顺序平均表位对数比之和来定义组合。由于多种原因,我们建议使用DiscoTope预测不连续的表位残基。
- 首先,我们在不连续表位的数据集上显示,DiscoTope的平均预测性能显着高于Parker倾向量表,略高于NACCESS RSA分数定义的表面定位分数。
- 其次,我们已经证明DiscoTope可以正确预测已使用不同技术(例如噬菌体展示,点突变和序列分析)鉴定的表位中的残基。
- 第三,DiscoTope预测方法可在 www.cbs.dtu.dk/services/DiscoTope 上公开获得,并且该方法的输出易于解释。
Parker亲水性标度通常用于通过平滑七个残基窗口中的值来预测线性B细胞表位(Parker等人1986)。与在此处开发的九个残基窗口上平滑的表位对数比相比,Parker量表对于预测数据集中不连续的B细胞表位的准确性不高。两种量表之间的等级差异表明,我们的对数比代表了表位的更多特征,而不仅仅是亲水性。可能的是,这种差异有助于在数据集上实现更好的预测性能,因为包括亲水性,柔韧性,可及性和β转向预测在内的各种倾向性尺度的组合都比表位预测的单个倾向性尺度更好(Pellequer等人1991)。我们的发现表明表面可及性值改善了B细胞表位中残基的预测,这与Batori等人最近报道的结果一致。 (2006)。此外,倾向性比例尺方法与结构信息的结合大大提高了性能。这表明可及性和化学特性在不连续B细胞表位的描述子中都很重要。使用多种倾向量表的组合方法已用于B细胞表位超过15年了(Pellequer等,1991)。然而,DiscoTope是第一个报道的将倾向性标度与三维结构信息(例如空间接近度)结合在一起的方法。
Van Regenmortel(1996)解决了使用蛋白质序列预测B细胞表位的问题,实际上是多维的。他得出结论,准确的预测需要更多的输入数据,例如抗原三维结构。 B细胞表位预测的结构输入要求是该方法普遍使用的限制因素。但是,结构基因组学项目有助于增加通常由蛋白质确定的X射线晶体学结构的数量,并覆盖更大的结构空间区域。因此,蛋白质结构作为预测方法的输入的需求将成为一个减少的问题,因为将确定更多的结构并且可以获得更好的同源性模型。
通常,基于结构信息的方法显示出预测不连续B细胞表位中的残基,与仅使用顺序信息的倾向性标度方法相比,在平均AUC中测得的性能更高。在所有评估方法中,DiscoTope方法均显示出最高的性能。但是,我们发现在95%和90%的特异性水平上,Parker亲水性标度比NACCESS RSA方法具有更高的灵敏度。这些结果说明了使用AUC以外的其他绩效衡量指标进行评估的重要性。
我们发现,对于包含较大生物复合物一部分抗原的抗原组,NACCESS RSA方法和DiscoTope方法的性能均相对较低。较低的性能是由于对蛋白质相互作用的区域或嵌入在膜中的区域的表面可及性的测量不正确。因此,我们认为B细胞表位的预测方法的结果应与有关特性的其他信息相结合,例如生物复合物的形成,膜相互作用和糖基化。
所描述的B细胞表位预测方法的准确性仍然相对中等。这可能部分是由于数据集抗原中表位的不完全鉴定所致。如果这些方法正确预测了在相应的复杂PDB文件中未被抗体结合的表位,则将其视为假阳性。但是,由于在这里描述的所有方法的评估中都使用了相同的数据集,因此我们假设不完全识别对所有方法的预测性能具有相同的影响,因此对它们的相对排名的影响可以忽略。 Batori等人开发的方法的预测性能(2006)评估了一种单一抗原的六个表位。该评估方法使用一种抗原,在该抗原中所有表位都被更完整地识别,可能会产生假阳性率较低而测得的性能较高的效果。在我们的评估方法中,我们选择包括尽可能多的变异,从而避免使该方法偏向某种类型的抗原或表位。但是,将来对使用具有更完全鉴定的表位的抗原数据集对我们的DiscoTope方法进行评估会很有意义。
最近,Schlessinger等(2006)开发了一种识别抗体/抗原复合物结构中表位的复杂方法。该方法基于抗体的互补决定区(CDR)的分析和鉴定,以及随后通过将抗原中的残基定位于CDR附近来鉴定表位。本文所述的鉴定通常仅基于抗原残基附近的抗原残基,因此有可能是Schlessinger等人开发的鉴定方法的未来应用,可以改善DiscoTope方法。
由于它们的非线性,在疫苗设计中,不连续的表位会带来除线性表位以外的其他问题。新疫苗不仅必须包含结合和引发特异性抗体所必需的氨基酸或原子,而且还需要保留正确的空间构象。 DiscoTope可以预测可能是不连续表位一部分的残基。随后,抗体结合研究和定点诱变可能有助于将预测的表位残基分组为表位并验证结合。分析抗原结构中抗原决定簇残基的局部构象也可能有助于疫苗的设计,因为基于不连续抗原决定簇的疫苗必须保留这些构象。可以使用天然蛋白质,蛋白质的亚域,携带表位的重新设计的蛋白质或疫苗中的模拟表位肽来获得保存。因此,我们认为不连续的表位可用于合理的疫苗设计。
四、材料和方法
4.1 准备数据集
从抗体晶体结构信息的SACS数据库中获得了实验确定的蛋白质抗原-抗体结构的列表(Allcorn和Martin 2002)。列表被过滤,仅包含分辨率> 3的结构,其中蛋白抗原> 25个氨基酸。从蛋白质数据库(PDB, http://www.rcsb.org/pdb )下载了与过滤列表相对应的坐标文件。最终数据集包含76种抗体-抗原对复合物。数据集中的表位残基定义为原子氨基酸距离抗体原子4埃以内的抗原氨基酸。基于五个已鉴定表位的子集进行比较,这些表位的残基据报道与抗体发生相互作用(Padlan等,1989; Muller等,1998; Fleury等,2000; Mirza等,2000; Romijn等,2003),阈值4Å给出的注释与人类专家所做的注释相当好(正确识别了92%的表位残基,只有1%的非表位残基被识别为表位残基)。 每个PDB文件中仅代表一个表位。在我们的分析中,给定抗原中可能存在的所有其他表位被视为非表位。某些抗原在数据集中多次出现(29种抗原是溶菌酶的变体)。因此,我们根据抗原同源性将数据集分组。使用BLAST搜索(Altschul等,1997),针对BLOSUM80矩阵针对数据集中的所有其他抗原,并结合Lund等人(1997)所述的同源性阈值,确定了76种蛋白质的数据中的同源性。然后将抗原分为25组,各组之间的同源性较低(各组之间的BLAST E值> 0.30)。数据集注释和抗原组可在 http://www.cbs.dtu.dk/suppl/immunology/DiscoTope 上公开获得。最后,将25个非同源抗原组分为五个数据集,用于交叉验证的训练和评估。
4.2 Parker亲水标尺的使用
按照Parker等人的建议,将每个残基表位的预测值使用七个残基窗口上的平均Parker标度值。 (1986)。
4.3 表面残留物的定义
通过使用残基接触数获得氨基酸表面定位和结构突出的组合量度。 残基接触数是指在残基Cα原子10Å以内的抗原中Cα原子的数目(Nishikawa and Ooi 1980)。 为了更直接地测量残留溶剂的可及性,使用NACCESS程序(Hubbard and Thornton 1993)计算了从每个PDB文件中提取的抗原链的每个残基的相对溶剂可及表面积。 使用NACCESS默认选项时,探头半径为1.4。
4.4 绩效指标 Performance measures
接收机操作员特性曲线(receiver operator characteristics curve,AUC)(Swets 1988)下的面积用作性能指标。通过改变预测阈值并在X轴上绘制假阳性比例或1-特异性相对于Y轴上真实阳性比例或灵敏度来绘制接收机操作员特征曲线(Swets 1988; Lund等(2005年)。我们根据每种蛋白质计算AUC。这确保了预测,其中蛋白质中的所有残基仅被预测为表位或仅非表位的预测的AUC为0.5,对应于随机预测。每种方法的性能均以25个抗原组的平均AUC,平均特异性和平均灵敏度进行衡量。
4.5 统计分析
使用双面t检验分析表位残基和非表位残基的接触数平均值(标准差= 0.121,表位残基的n = 1202,标准差= 0.050,非表位残基的n = 13,242。)该方法用于平均AUC值的成对比较,以确定性能的重要性(Efron和Tibshirani 1993)。对于每种方法,每个抗原组的25个平均AUC值被重新采样100,000次,以获得对P值的可靠估计。
4.6 表位对数比的推导
Derivation of epitope log‐odds ratios
五个数据集(训练集)中的四个用于推导表位对数比。通过在训练集中的抗原序列上滑动一个奇数大小的窗口,可以产生一系列肽。然后根据中间位置作为表位残基或非表位残基的鉴定,将肽分为表位组和非表位组。使用Nielsen等人的方法从每组中的肽计算重量矩阵。 (2004年),包括序列聚类,序列加权和权重为200的伪计数。最后,计算了抗原决定基组中20个氨基酸相对于非抗原决定基组在中央矩阵位置的对数比以半位为单位,用作表位倾向量表。
4.7 使用对数比进行表位预测 Using log‐odds ratios for epitope prediction
为了预测表位残基,可单独使用原始对数比,或与计算表位倾向性标度值的序列平均值的平滑窗口结合使用。相对于用于计算对数比的训练集的预测性能,确定了用于推导对数比的最佳肽段长度和平滑窗口的最佳大小。报告的性能是数据集的五倍交叉验证的性能。这减少了高估性能的风险,因为对数比的计算和其他参数(例如肽长度和平滑窗口大小)的优化是在训练集上估算的,因此不会因评估而产生偏差设置数据。
4.8 倾向量表与基于结构的方法的简单组合
接触数,NACCESS RSA和Parker亲水性值通过减去平均值并除以标准偏差进行标准化。归一化的接触数乘以-1,以使高值与表面定位相关。随后,使用线性组合将不同的倾向等级与接触数或NACCESS RSA进行组合,表面度量的权重范围为0.001至100。使用训练集确定最佳权重。最后,在评估集上评估了性能。
4.9 表位对数比的结构邻近总和
另外,通过对所有残基中每个具有10个Å距离内的Cα原子的残基求和,使用表位对数比或Parker亲水性标度。例如,我们根据到中心残基的距离,残基的接触数以及两者的组合,对邻近度和进行了多种加权方案的测试。但是,所有残基均具有相同重量的简单方法在训练集上具有最高的性能(数据未显示)。
4.10 AMA1中的表位预测
来自恶性疟原虫的AMA1胞外域的链A(PDB代码1Z40)用于DiscoTope表位预测。 我们选择使用1Z40代替全长AMA1胞外域结构(1W8K),因为在后者中未观察到4G2表位的主要残基部分。 348、351、352、354-356、385和388-389残基被算作4G2表位中的残基(Pizarro等人,2005年); 191-199位残基被计为1F9表位的一部分(Coley等,2006); 残基187、197、200、230和243被视为高度多态性残基(Bai等人,2005年)。
参考资料
- P. H. Andersen, M. Nielsen and O. Lund. 2006. Prediction of residues in discontinuous B cell epitopes using protein 3D structures. Protein Science. 15:2558-256. 网址:https://onlinelibrary.wiley.com/doi/full/10.1110/ps.062405906
