【8.1】在NGS库中寻找治疗性抗体
最近,使用新一代测序(NGS)可以查询天然抗体库的多样性。 这些方法能够在单个实验中产生数百万个序列。 在这里,我们比较临床阶段治疗抗体与Observed Antibody Space Database中60个独立测序研究的~1b序列。 在242个I期后抗体中,我们发现16个重链和轻链的序列同一性匹配度为95%或更高。 在NGS输出中还有54个与治疗性CDR-H3区域的完美匹配,表明自然观察到的序列与人工开发的序列之间存在大量的收敛。 这对于抗体治疗的发现和商业抗体的法律保护都具有潜在的意义。
网址: http://naturalantibody.com/
一、前言
抗体是下颚脊椎动物中的蛋白质,其识别用于消除有害分子(抗原)。 有机体表达数百万种不同的抗体,以增加它们中的一些能够结合外来抗原的机会,从而启动适应性免疫应答。 现在可以使用B细胞受体库的下一代测序(NGS)查询这种巨大的多样性,从而能够快速收集来自任何给定个体的数百万抗体序列。
蛋白质的序列,例如抗体,是在专利权利(patent claim)要求中表征该分子的主要载体之一。 然而,自然序列在美国不能获得专利。 Mayo和Prometheus案件中的开创性裁决开创了拒绝对自然发生的DNA序列的专利权利要求的先例,并随后扩展到包括所有自然产物。 现在在公共领域中可获得的大量抗体序列提高了发现与商业序列相同的天然序列的可能性。
通过大规模有组织的努力,使得自然来源的抗体NGS数据和分析更容易获得。 具体来说,我们最近创建了Observed Antibody Space(OAS)数据库,该数据库从公共档案中提取NGS抗体数据并使其易于处理。 OAS目前拥有来自60项独立研究的~1b(~960m重链和~60m轻链)序列。 该数据集涵盖多种生物(人类,小鼠,兔,骆驼等),个体,免疫状态(非免疫,免疫等),并包括对小鼠(246m序列)和人类((246m序列)和人类的自适应全集的整体深度测序( 318米序列)。 在这里,我们量化了序列与我们在OAS中可以找到的当前临床阶段治疗(Clinical Stage Therapeutic,CST)抗体序列的匹配程度。
二、结果
我们使用了一组242个CST抗体序列。 这些都是通过临床试验第一阶段的序列。 我们分别将CST可变区(VH或VL),来自VH或VL和CDR-H3的三个互补决定区(CDR)的组合与OAS中的所有序列(参见方法)对齐。 我们对所有生物,个体和免疫状态进行了全面的搜索,以反映无数的抗体类型(人源化,完全人类,嵌合或小鼠)。 关于OAS最佳匹配的CST的个体身份在图1和表1中给出,它们的分布在图2中绘制。对齐的序列可在补充材料和我们的网站 http://naturalantibody.com/therapeutics 中获得。
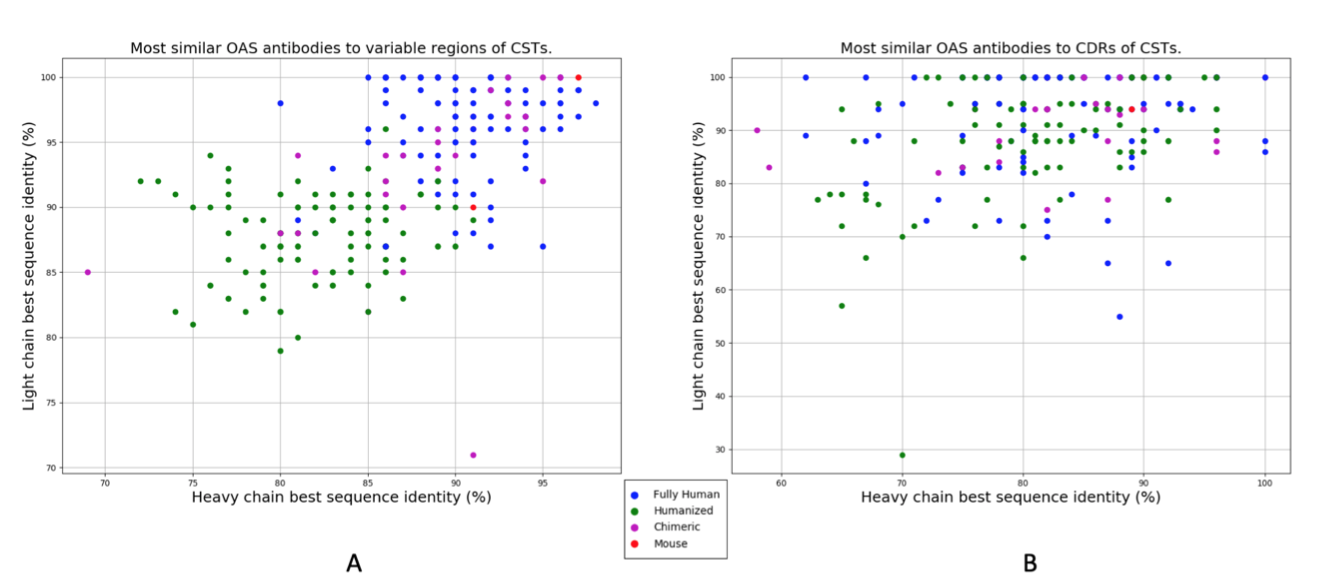
图1. 最佳序列同一性与自然来源的NGS数据集中的临床阶段治疗(CST)相匹配。 A)来自Raybould等人的242个CST序列的重链和轻链可变区与OAS中的可变区序列对齐。 B)242个CSТ的重链和轻链IMGT CDR区与OAS中的IMGT CDR区对齐。 完全人类序列用蓝点表示,绿色表示人源化,品红色表示嵌合。 两个小鼠序列以红色显示。 http://naturalantibody.com/therapeutics 上提供了这些图表的交互式版本。
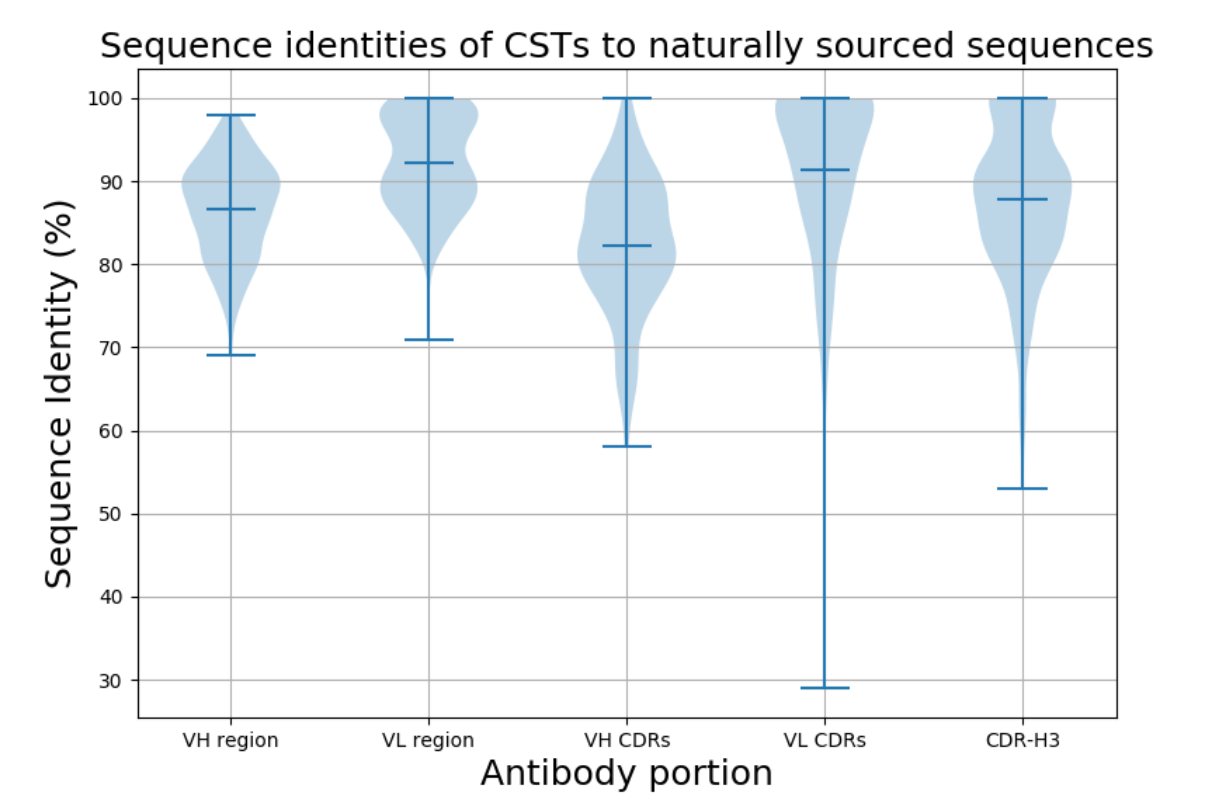
图2 临床阶段治疗(CST)与天然来源NGS的序列同一性匹配的分布。 小提琴图显示CST的可变重链(VH)和轻链(VL),重链和轻链CDR区和CDR-H3的序列同一性的分布,以在OAS中最佳匹配。
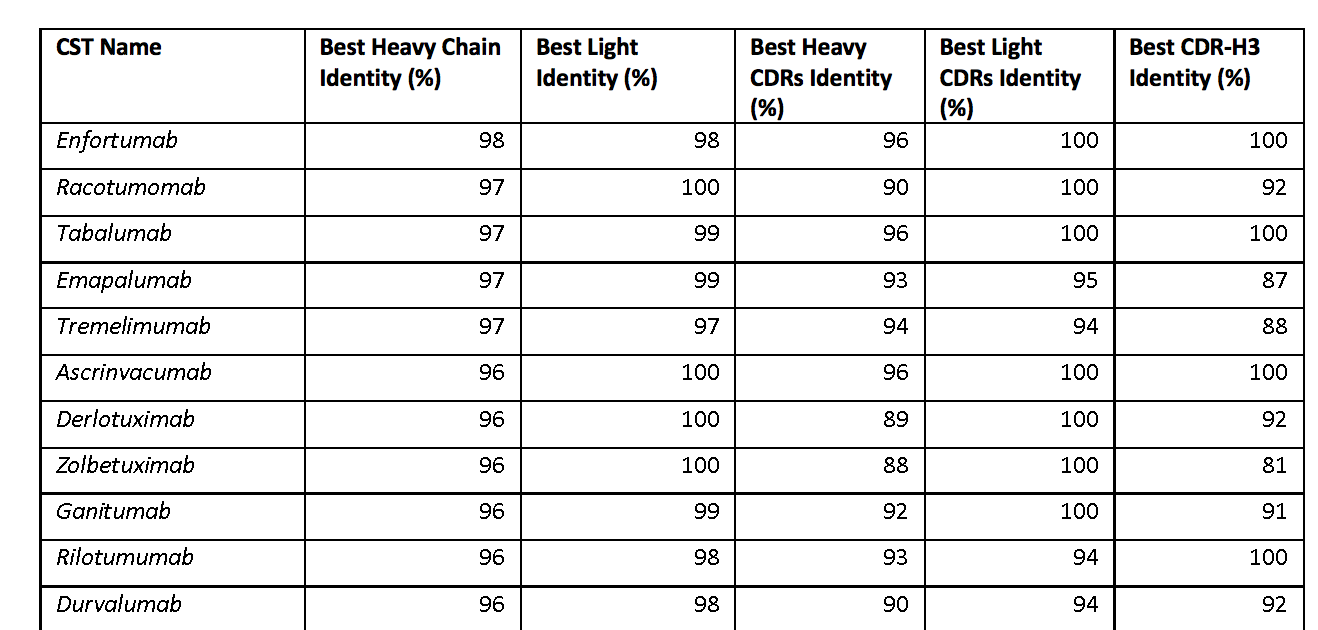
表1. 临床阶段治疗(CST)抗体与公共NGS储存库中发现的序列的最佳序列同一性。 给出序列同一性以使序列从公共储存库到CST重链或轻链可变区,重链或轻链CDR区或单独的CDR-H3(IMGT定义的)的最佳比对。 CST由最左侧列中的名称标识。 条目按最高重链标识从上到下排序。 该表的交互式版本以及对齐的序列可在 http://naturalantibody.com/therapeutics 上获得。
2.1 临床阶段的分析治疗序列与自然来源的NGS数据集匹配
图1A中给出了CST可变区与OAS中天然来源的NGS数据集的最佳序列同一性匹配。 90个CST重链具有≥90%序列同一性(seqID)的OAS匹配,其中18个(7.4%)≥95%seqID。 我们发现158个(65.2%)治疗性轻链,其序列号≥90%,对于OAS序列,96个(39.7%)≥95%seqID,28个(11.5%)具有100%seqID。 对于16(6.6%)的CST,我们发现重链和轻链匹配≥95%seqID。 在极端情况下,Enfortumab,我们能够找到98%seqID的重链和轻链匹配(差异是H38:NS,H88:SY,L37:GS,L52:FL,其中第一个氨基酸来自Enfortumab 和来自OAS序列的第二个)。
CST与任何给定的天然表达抗体之间的最大差异通常集中在确定抗原互补性的CDR区中。 然而,尚不清楚工程化治疗剂的高度可变CDR环与天然表达的CDR环的程度不同。 我们在OAS搜索了与CDR区域最佳的CST匹配。 使用来自重链或轻链的IMGT定义的CDR三联体进行搜索,忽略框架区(表1,图1B和图2)。 我们发现46个(19.0%)的CST重链CDR区与具有≥90%seqID的OAS CDR区匹配,15个(6.1%)具有≥95%seqID和4个(1.6%)具有100%seqID。 存在156个(64.4%)CST轻CDR区,其对于OAS CDR区具有≥90%seqID,其中110(45.4%)≥95%seqID,90(37.1%)具有100%seqID。 我们在两个CST,Obiltoxaximab和Zanolimumab中找到了轻链和重链CDR的完美匹配。
在六个互补决定区中,CDR-H3是序列最多且结构多样的。由于其在结合中的关键作用,它受到广泛的抗体工程。我们检查了在天然来源的序列中找到CST衍生的CDR-H3的可能性。为了评估这一点,我们在OAS中搜索了最佳的CST CDR-H3匹配,无论框架区域和剩余的CDR如何(表1,图2)。在我们的242个CST CDR-H3中,我们在OAS中找到了54个完美匹配。完美匹配倾向于较短的CDR-H3,但也发现了一些具有完美匹配的较长环(参见补充第1节)。在最近Briney等人的深度测序数据集中发现了29个完美匹配。 (2019),表明单一的综合NGS研究可以涵盖大量的CDR-H3多样性。除了Briney等人(2019)之外,在OAS数据集中发现了47个完美匹配,显示在天然来源的NGS中可以独立地观察到某些人工CDR-H3序列。在Briney等人的数据和其他OAS数据集发现了二十二个CDR-H3匹配。这22个共享序列来自9个人源化和13个完全人类CST。 54个完美的CDR-H3匹配分布在所有抗体类型中,具有23个人源化,22个完全人,8个嵌合和1个小鼠(分别为21.9%,22.0%,22.8%和50.0%)。这些结果表明,尽管CDR-H3区域可获得大的理论序列空间,但在来自60个NGS研究的仅约960m重链序列中发现了治疗上可利用的CDR-H3环(参见补充部分2)。这种趋同与CDR-H3环通常介导抗体特异性和结合亲和力的事实相结合,可能表明本质上驱动抗原识别中的偏差,与人工发现方法无关。
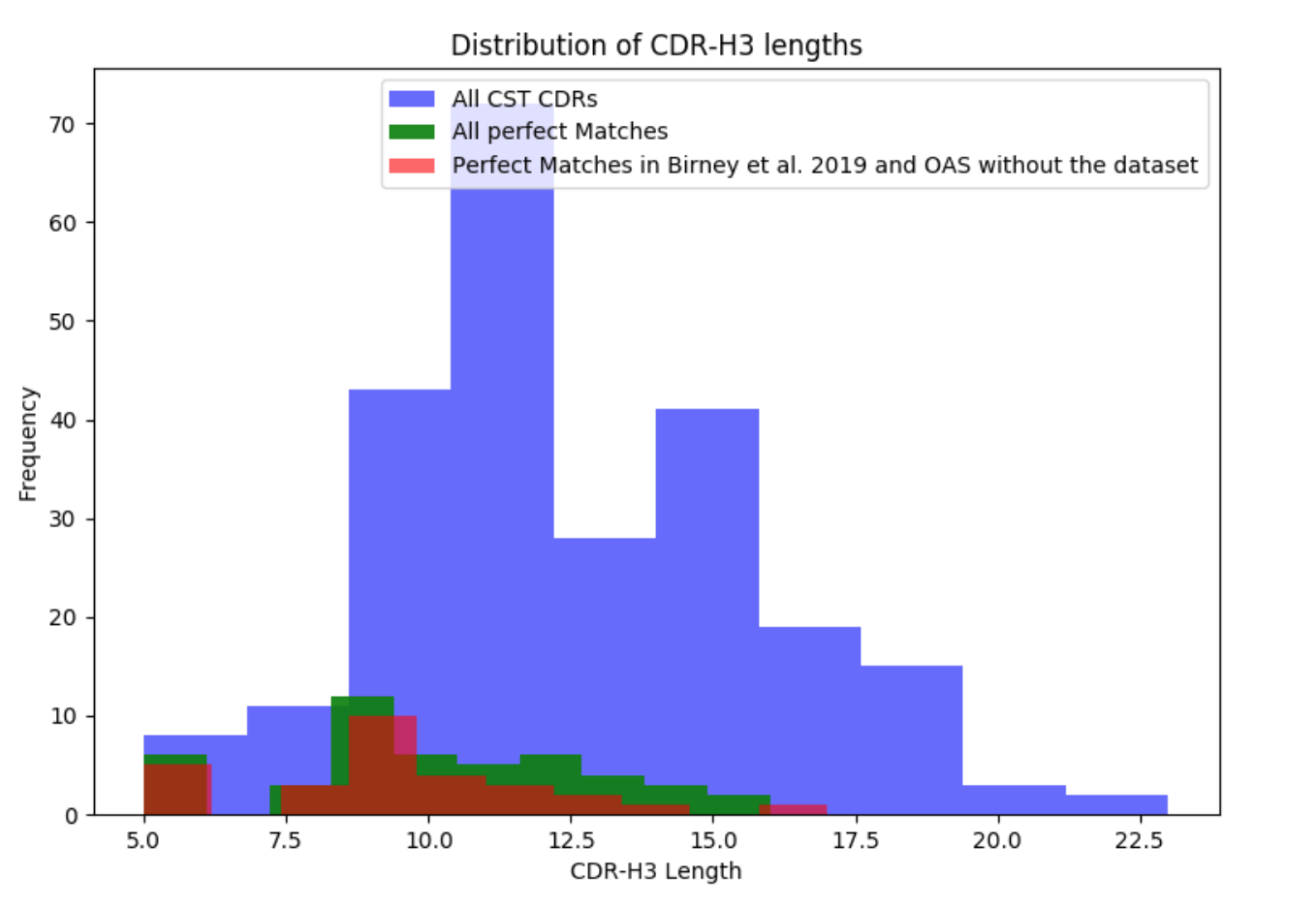
补充图1. CST CDR-H3长度(蓝色)的分布覆盖在完美匹配的长度(绿色)上。 红色直方图显示了CST CDR-H3的长度分布,其可以在Birney等2019的深度测序数据集中,以及在去除Birney等2019数据时在OAS中的其他数据集中找到。
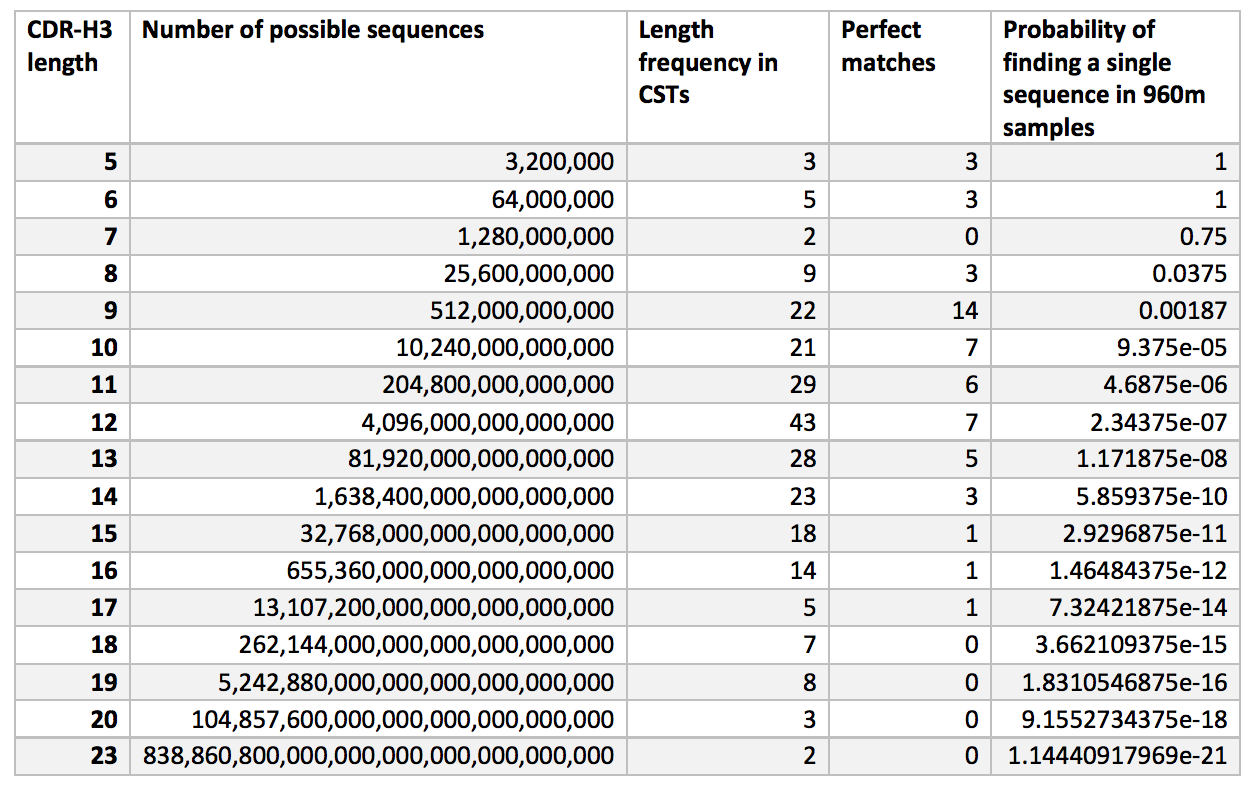
补充表1. 给定940m独立样品的每个IMGT长度的CDR-H3序列的估计理论数和找到单个序列的概率。
2.2 通过抗体类型对OAS中的最佳CST匹配进行分层
对于任何给定的CST序列,我们可以找到的可变区匹配的质量似乎高度依赖于发现平台/抗体类型。 图3表明通过更人工方案(例如人源化)产生的抗体与OAS中的序列具有比完全人分子更低的可变区序列同一性。 对于大多数完全人序列,我们发现90%seqID或更好的匹配,而与大多数人源化分子的匹配低于90%seqID(图3)。 嵌合抗体似乎具有介于两个类之间的seqID值(图3)。
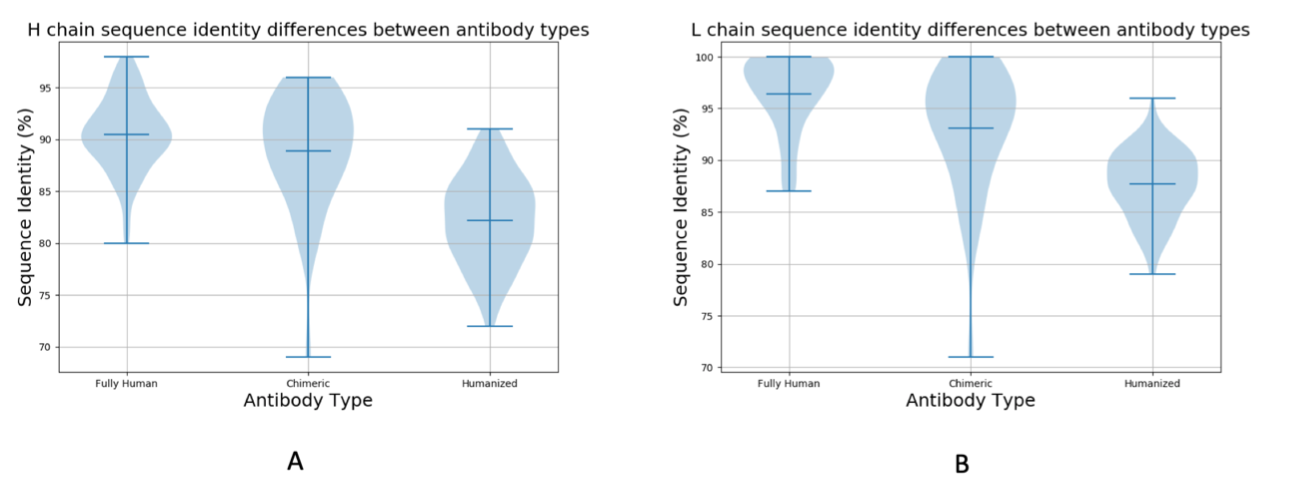
图3.临床阶段治疗(CST)可变区与通过CST抗体类型分层的天然来源的NGS数据集的序列同一性匹配。 CST A)重链和B)轻链与OAS中NGS序列的同一性,其由完全人,嵌合和人源化抗体类型分层。 由于样本太小,两个小鼠分子被省略。
CST抗体类型也反映了产生最佳NGS seqID匹配的生物体。 在100个完全人CST中,90个(90.0%)最相似的重链,100个(100.0%)最相似的轻链和55个(55.0%)最相似的CDR-H3环来自人源NGS。 在105种人源化抗体中,82种(78.0%)重链和79种(75.2%)轻链在人源NGS中发现最接近的匹配,而在小鼠资源NGS中鉴定出71种(67.6%)最佳CDR-H3匹配。 这进一步反映了CDR-H3在结合中的主导地位,因为治疗公司倾向于从结合小鼠抗体移植该环以转移特异性和结合亲和力。 它还表明,对数据集(如OAS)进行数据挖掘可以比我们当前的指标更准确地衡量抗体的“humanness”
三、讨论
我们的结果表明,尽管理论上允许抗体大量多样性,但在人工开发的CST和天然来源的NGS序列之间存在非平凡的趋同。 与CST最接近的NGS匹配来自OAS中可用的60个(80.0%)独立数据集中的48个,表明在大多数NGS数据集中可能找到与至少一个CST的紧密匹配。
先前曾提出,这种重叠可能会导致声称治疗性抗体专利的问题。在有关该主题的法律意见可用之前,尚不清楚NGS数据是否会对单克隆抗体的可专利性造成潜在问题。首先,序列是在专利权利要求中表征分子的主要方式之一,但是它本身并不提供关于其同源抗原和治疗作用的信息。然而,这种信息在专利申请中是至关重要的,以证明本发明的新颖性。 NGS研究产生大量序列,但它们并不单独与任何靶分子相关。其次,抗体可变区是两条多肽链(重链和轻链)的产物,其功能与该组合密切相关。目前,大多数可用的NGS数据集分别报告重链和轻链。第三,已知NGS输出具有高错误率,并且最后,不清楚序列同一性与公开可用序列或其部分(例如CDR-H3)的匹配程度是否会在确定序列的创造性方面引起问题。例如,仅存在四对具有重链序列同一性超过94%的CST(参见补充部分3)。在三个这样的高序列同一性对中,两个序列来自同一公司,第四个是原始的专利过期抗体及其衍生物。将其与18种治疗性重链进行比较,其与OAS的匹配优于95%。
鉴于正在进一步整合抗体NGS数据并使其更易获取,随之而来的是在已发表的NGS数据集中找到治疗候选序列将变得更容易
四、方法
我们使用Observed Antibody Space数据库作为NGS序列的来源。 自第一次发布以来,该数据库已经被另外四个数据集扩展,最值得注意的是Briney等人最近对人类抗体库的深度测序。 我们使用来自Briney等人的加工共有序列,去除具有终止密码子的任何序列,因为这些序列被认为是非生产性的。
我们使用来自Raybould等人的242抗体作为Clinical Stage Therapeutic(CST)抗体的来源。 我们使用ANARCI根据IMGT方案对CST序列进行编号。 根据其国际非专利名称,将CST序列分为四组。 名称中包含'-xizumab',' - ximab’或'-monab’的序列标记为’chimeric'。 不匹配此标准但在其名称中包含“-zumab”的序列被归类为“人源化”。 在其名称中仅包含'-umab’的序列被标记为’完全人类'。 将两种小鼠抗体(Abagovomab和Racotumomab)标记为“小鼠”。
我们将重链,轻链,三条重链或轻链IMGT定义的CDR和IMGT定义的CST的CDR-H3的组合,分别与OAS中的每个序列比对。如果在’query’CST中的IMGT位置也在来自OAS的’template’序列中找到,我们注意到匹配,并且它们具有相同的氨基酸残基。对于完整序列比对,匹配数除以查询的长度和模板的长度,产生两个序列同一性。最终的序列同一性是这两者之间的平均值。以这种方式计算序列同一性可防止当一个序列是另一个序列的子串时的情况,从而创建具有大长度差异的人为高序列同一性。当IMGT定义的环长度匹配时,进行CDR比对。对齐的序列可在补充部分4中找到,也可通过 http://naturalantibody.com/therapeutics 上的图1和表1的交互式版本获得。
参考资料
- bioRxiv preprint first posted online Mar. 9, 2019; doi: http://dx.doi.org/10.1101/572958. Looking for Therapeutic Antibodies in Next Generation Sequencing Repositories
