【4.5.6】抗体CDR结构分类的数据库--PyIgClassify
官网: http://dunbrack2.fccc.edu/PyIgClassify/default.aspx
抗体互补决定区(CDR)结构的分类对于抗体结构预测和计算设计至关重要。我们之前已经对抗体CDR构象进行了聚类,并定义了一个系统的术语,由CDR,长度和从数据集中最大到最小的簇开始的整数组成(例如L1-11-1)。我们介绍了PyIgClassify(用于基于Python的免疫球蛋白分类;可从 http://dunbrack2.fccc.edu/pyigclassify/ 获得),该数据库和Web服务器可访问PDB中所有CDR结构对我们分类系统的分配。该数据库包括对多个物种的重链和轻链的IMGT种系V区的分配。对于人源化抗体,框架的分配是人类种系,而CDR是小鼠或其他物种来源的种系。可以通过PDB条目,簇标识符和IMGT种系组(例如人IGHV1)来搜索数据库。整个数据库都是可下载的,因此用户可以根据需要对数据进行过滤,以进行抗体结构分析,预测和设计。
一、前言
脊椎动物的免疫系统产生各种抗体序列和结构,以识别微生物和细菌表面的外来抗原以及异常的自身抗原。抗体蛋白的序列是由免疫球蛋白基因产生的,这些免疫球蛋白基因已通过称为V(D)J重新组合的过程在不同的基因位点进行了重排,该基因位点包含最终重组基因每个片段的多个拷贝,每个组分由一个选择可变区(V),多样性区段(D,仅在重链基因中发现)和连接区(J),其后是恒定区(C)(1)。大多数哺乳动物,鱼和禽类抗体均由重链和轻链组成,它们各自分别是V(D)J或VJ重组的产物。在每个物种中,轻链可由一个或多个基因座产生,从而产生额外的多样性。例如,在大多数哺乳动物中,κ和λ基因座用于产生轻链蛋白。
由于第一个抗体的序列和结构是在1960年代和1970年代确定的(2-4),因此已尝试通过序列和结构对互补决定区或CDR进行分类。最早的关于结构的综合尝试是Chothia等人的尝试。 (5,6),他为抗体CDR创造了“规范结构”一词,表明每个CDR(L1,L2,L3,H1,H2,H3)可能仅基于长度和序列采用一些通用结构。随着更多结构的确定,Chothia等人在1990年代中期扩展了早期的分类。 这些分类在接下来的十年中会定期更新(9),其他分类也出现在当前PDB的子集中(例如H3 CDR或链)(10-12)。 Nikoloudis等最近,基于截至2011年12月的PDB(13),提出了抗体CDR结构的分层集群(13),但未作为服务器或数据库使用。
2011年,我们基于二面角度量和亲和力传播聚类算法发布了抗体CDR结构的全面定量分类(14)。到2011年,独特抗体结构的数量超过300种,并且可以在高质量数据集上执行自动聚类(即删除具有低分辨率和/或高B因子的结构)。与Chothia系统相反,我们为抗体CDR簇开发了系统的命名法,使得每个簇都以CDR和长度命名,然后是一个以最大簇开头的整数,例如L1-11-1是最大的CDR L1长度11簇。提供了每个簇与基因座(重链,κ和lambda)和物种的暂定关联。抗体CDR构象的最新数据库已使用我们的分类系统(13,15)作为参考,并且在更广泛的抗体文献(16,17)和工业界(18-20)中得到了认可。
抗体结构的分类及其与基因座,种类和序列的相关性导致改进的抗体结构预测(21–23)和抗体设计的机会(24,25)。因此,我们在CDR结构分类系统中实现了PDB中CDR结构的自动分配(14),在本文中,我们提供了这些分配的综合数据库和服务器PyIgClassify(用于基于Python的免疫球蛋白分类) ,它将定期更新。 PyIgClassify也将根据需要使用新的群集进行更新。即使在2011年,很可能已经观察到人和小鼠抗体中的所有主要构象簇,并且由于来自种系或来自CDR的CDR长度的体细胞或工程学改变,唯一的新构象要么是先前未观察到的长度,要么是PDB以前没有代表的新物种的结构。
除了了解PDB的最新信息外,我们还研究了CDR簇与框架和CDR区的种系V区之间的关系。 PDB中的许多抗体已从种系序列中进行了充分的成熟(substantial maturation),并且在许多情况下已进行了精心设计。在治疗药物的某些情况下,CDR来自一种抗体和物种,例如小鼠,而框架主要是人类起源的。因此,分配正确的种系V区是一个具有挑战性的问题。我们已经根据IMGT术语(26)仔细确定了PDB中每种抗体的种类和种系V区,并鉴定了将CDR移植物从小鼠或其他物种移植到人骨架上的抗体。在许多情况下,CDR1和CDR2片段的长度与种系V区中最类似于构架序列的相同CDR的长度不匹配。这些结构提供了有关将不同长度的CDR移植到与曲妥珠单抗和其他抗体密切相关的常用的高度稳定的框架(例如人IGHV3-66 / IGKV1-39框架)上的可能性的有用信息。我们发现PDB中有9.5%的非冗余抗体是小鼠/人移植物,而16.6%的CDR长度与框架种系的CDR长度不匹配,从而提供了充足的数据集来检查抗体的计算设计。
二、材料和方法
补充方法中介绍了确定PDB中哪些蛋白质序列包含抗体VH和VL结构域以及将IMGT V区种系分配给这些序列的方法。
2.1 确定抗体CDR簇
对于具有鉴定的抗体VH或VL结构域的每个PDB结构,我们确定CDR序列及其长度,这代表我们分类系统的第一级(例如L1-11,L2-8等)。对于具有完整骨架坐标的CDR,我们使用内部脚本计算每个CDR中残基的ω,ϕ和ψ二面角。
下一级别的分类是通过环中残基的顺式-反式模式进行的。一些CDR长度组合通常具有顺式脯氨酸残基(例如7位的L3-9),而出乎意料的CDR具有顺式-非脯氨酸残基,这可能是由于低分辨率和不良的结构精制(请参见结果部分) )。如果长度是新的(13个案例)或顺式-反式模式是新的(53个新案例),我们用通用簇标识符(例如,长度为5的CDR L3的L3-5- *标记该环,该长度在整理的2011年数据集;或L1-11-cis4- *为CDR L1长度11,在位置4具有顺式残基)。这些簇中没有一个具有超过七个的非冗余序列(H2-11- *),在66个中的47个(71%)中只有一个序列。在2011年的分析中,我们排除了具有顺式-非脯氨酸残基的CDR。当前数据库涵盖了所有抗体结构,无需进行先验过滤。
对于在我们原始分析中具有簇的每个CDR长度和顺式-反式,我们使用相同的二面体,计算了该环状结构与该CDR具有相同长度和顺-反式的簇的每个质心的距离角度度量标准(如2011年的工作):
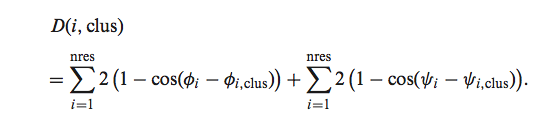
这是方向统计中使用的两个角度之间的适当距离(28)。 数据库和Web服务器提供了距质心的距离,我们发现40度的平均二面角距离和1.5Å的主干RMSD距离对于识别聚类成员是合理的。 来自任何现有群集质心的RMSD中大于40°或1.5Å的CDR被分配给形式为L1-11- *的通用群集。
2.2 数据库和网站
PyIgClassify的内部数据库和“可下载”数据库是SQlite( http://www.sqlite.org )关系数据库,因为它支持并直接集成到各种计算语言和分子建模套件中,包括R,Python,C ++,BioPython和Rosetta( https://www.rosettacommons.org )。每个数据库的制表符分隔文本版本也可用。每个数据库至少包含四个表:cdr_data,SpeciesNames,GermlineAssignments和CdrClusterSum。 cdr_data表保存有关每个CDR的簇,序列,结构和种系的信息,以及每个已识别抗体结构的框架。 SpeciesNames表列出了数据库和网站中使用的物种及其简称,而GermlineAssignments表通过将每个抗体序列与IMGT进行比较,为CDR和框架提供了种系分配(http://www.imgt.org/ )种系序列。
在每个数据库中,每个CDR群集都有一个汇总表(CDRClusterSum)。该表包括一个簇中唯一序列的数量以及其他有用的摘要信息,例如中值PDB,基因和可以找到该簇的已识别PDB种类。从群集质心(或中位数)的二面角的平均偏差是根据公式计算得出的
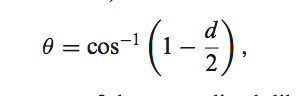
其中d是群集中每个成员与群集中位数的标准化二面角距离的平均值。 在此文件中,PercentLoop是特定簇中的结构数除以该CDR(例如所有L1)的结构数。 PercentUniqSeq是群集中唯一序列的数量除以数据库中该CDR的唯一序列的数量。 环构象是就Ramachandran构象而言的中位环构象,而ConsSeq是簇中序列的共有序列(簇中独特序列中每个位置的最常见残基)。 数据库每月更新一次,以反映PDB的当前状态。
此外,在Honegger-Plückthun编号方案(29)中对所有鉴定出的抗体进行了重新编号,并可从网站下载。
三、结果
在显示在PyIgClassify服务器上执行的搜索示例之前,我们对PDB中抗体条目的当前结构和种系覆盖率进行一些分析(总结在补充表S1-S6中)。
3.1 识别抗体V区
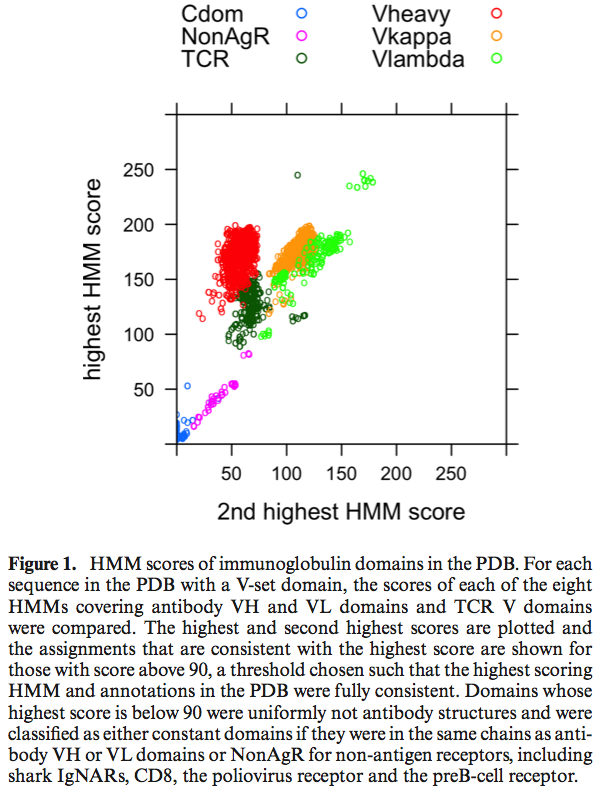
我们使用一组八个涵盖抗体VH,Vκ和Vλ区的隐马尔可夫模型(HMM)来识别抗体VH和VL区(对于包含相对于其他Vλ序列的框架插入的Vλ6序列中的一个),以及T细胞受体的α,β,γ和δ链。尽管其他免疫球蛋白序列之间的距离较远,但是在将它们的CDR构象聚类时,区分抗体和T细胞受体域很重要。在图1中,我们显示了PDB中每个正得分域的得分最高的HMM(y轴)相对得分次高的HMM(x轴)的散点图。根据经验,四个抗体HMM的最高分值90的临界值与PDB中每个序列的注释一致。这些点由它们对重链,Vκ,Vlambda,TCR,nonAgR(对于非抗原受体)和恒定域(Cdom)的分配标记。非抗原受体包括鲨鱼Ig-NAR,CD8,v-preB受体和人脊髓灰质炎病毒受体。总共,我们发现1897个PDB条目具有一个或多个抗体的VH或VL结构域,该抗体包含5711个链和17260个CDR。 T细胞受体共有240个条目。
3.2 Germline assignments
如补充方法中所述,我们根据种的PDB注释以及PDB的完整序列,构架序列和CDR序列的比较,将种系V区(而非D或J片段)分配给PDB中的抗体序列抗体与IMGT种系库中的抗体有几个种。从IMGT,我们能够获得人,小鼠,大鼠,雷氏达尼奥,猕猴,美洲驼,骆驼和兔子的VH区的种系序列。人,小鼠,兔,绵羊,大鼠和猪的Vκ区;人,小鼠,兔,大鼠和猪的Vλ区。补充表S1中针对所有条目和非冗余条目(针对CDR序列的每个唯一的级联序列一个)给出了对当前PDB的这些分配的摘要。对于人类和小鼠的59种IMGT种系,目前PDB中不存在11种:Hu_IGKV5,Hu_IGLV4,Hu_IGLV8,Mo_IGHV11,Mo_IGHV15,Mo_IGHV16,Mo_IGKV7,Mo_IGKV11,Mo_IGKV18,Mo_IGKV20和Mo_IGLVV2(分别是-IMGT中定义的区域)。
对于人类种系群,该表还包括由嫁接在这些人类(或人源化)框架上的小鼠CDR组成的结构数(请参阅补充方法)。该表显示了大量基于人源化4D5框架的抗体(27),与大多数此类抗体中的框架片段具有约95%的同一性,与人种系序列IGHV3-66和IGKV1-39框架最接近。共有78个具有不同CDR序列的抗体结构使用这些框架中的至少一个,而36个不同的抗体结构则使用两者。我们注意到,PDB中的绝大多数人源化抗体(那些具有小鼠CDR但具有人样框架的抗体)都包含人κ轻链框架,而所有这些都包含κ小鼠CDR。据推测这是因为小鼠没有产生大量的λ抗体(30),并且移植物(grafts)几乎总是κ到κ,而不是κ到λ,反之亦然。
该表还显示了每个种系组中结构中的CDR与亲本种系序列中的长度不同的次数。长度变化的分布如图2所示。这可能是由于工程序列或体细胞突变,它们可以通过复制密码子或消除重复的密码子来改变CDR的长度(31)。大多数改变的CDR长度与种系CDR长度仅相差+1或-1个氨基酸。 V区通过CDR3的第一个残基或两个残基覆盖了VH和VL域,因此补充表S1中仅显示了CDR1和CDR2的数据。由于CDR长度变异性的差异,这些数字高度不一致。例如,几乎所有的哺乳动物L2种系序列的长度均为8,因此CDR2的IGKV和IGLV序列没有显示长度不匹配,这显然是因为CDR L2既不经历也不容忍CDR长度的体细胞变化。 CDR H1在大多数小鼠和人类胚芽长度中的长度为13,而CDR H2在种系中显示多种长度。它还显示了与PDB中人抗体中框架种系的更多差异。

尽管PDB并不是一组代表性的抗体,但它确实包含有关VH和VL构架相互关联的频率的信息。 补充表S2中给出了常见人类VH和VL结构域的最常见关联的矩阵。 在括号中,给出了每对观测值与预期值之比。 IGKV1 / IGHV3组合代表了许多基于人源化4D5抗体的结构(32)。 但是其他关联值得注意,包括IGHV4区域与λ域关联的趋势。
3.3 CDR构象簇 CDR conformational clusters
轻链和重链的CDR构象的最大簇分别在补充表S3和S4中给出。给出了每个簇中唯一序列的总数,并且每个簇中都存在物种和基因座(对于L1,L2和L3为κ或λ)。如果簇中仅存在一个或两个种系V区(例如,表中的Mo_IGKV3缩写为Mo_KV3),则会列出这些区域。这些表提供了有关每个CDR的序列长度分布和簇分布的统计信息,这两者都是高度不平衡的。例如,在簇L2-8-1中有98.8%的L2 CDR的长度为8,而89.0%为长度。 H1也非常狭窄地分布,长度13的91.8%和簇H1-13-1中的81.1%。就变量分布而言,L3是下一个CDR,其长度9的83.0%和簇L3-8-cis7-1的70.9%。剩余的CDR,L1和H2在长度和簇方面,尤其是L1分布更为广泛。
现在,PDB中出现了许多新的CDR长度组合,2011年PDB中不存在这种组合,某些长度的一些顺反构型也是如此。这些都没有超过七个独特的序列。十三种新的CDR长度组合为:H1-9(1个序列),H1-11(1),H1-18(1),H1-20(1),H1-24(1),H2-11(7 ),H2-14(1),L1-7(1),L1-8(4),L1-9(3),L3-5(6),L3-6(3)和L3-13(1) 。尽管当时我们排除了具有顺式-非脯氨酸残基的CDR,但2011年分析中未发现PDB中存在53种顺式-反式模式。在这53个新的顺式-反式模式中,总共有45个是具有顺式-非脯氨酸残基的模式,很可能其中大多数是错误地精制的结构。例如,PDB条目1OCW具有10个不是脯氨酸的顺式残基(VH中为9个,VL中为1个)(33)。至少可以说,在大多数情况下,结构的分辨率不支持在PDB(顺式-非脯氨酸残基)中非常罕见的结构特征(34)。
我们对簇与种系之间的相关性感兴趣(在每个方向上),因此分析了每个簇(例如H1-13-1)的每个种系V区组(例如Hu_IGHV1)的患病率,反之亦然。关联表S5和S6中显示了最强相关性的结果。一些最大的簇,例如H1-13-1,包含来自小鼠,人类和其他物种的VH区的代表,实际上74%的独特H1序列属于簇H1-13-1。其他较不常见的CDR长度仅属于每个物种的某些V区,此外,某些轻链簇是位点特异性的(κ与λ),甚至是特定种系群特异性的种。例如,L1-11-3,L1-14-1和L1-14-2仅包含λ序列。此外,有趣的是注意到,人框架上的大多数小鼠CDR移植物都属于与小鼠CDR一致的簇,部分原因是大多数此类移植物已添加到具有相似CDR长度和CDR簇的人框架中。
补充表S6列出了每个主要种系组的主要簇。对于某些种系组,该组的唯一CDR长度也是仅包含一个簇的一个。例如,Hu_IGHV2和Mo_IGHV8种系序列仅包含长度为15的CDR1,并且全部完全在簇H1-15-1中。值得注意的是,一些重链种系分为H2-10-1或H2-10-2,这可能对结构预测有用。 Hu_IGHV1,Hu_IGHV5,Mo_IGHV1,Mo_IGHV9和Mo_IGHV15主要是群集H2-10-1,而Hu_IGHV3,Mo_IGHV4和Mo_IGHV5主要在群集H2-10-2中。 Hu_IGLV1和Hu_IGLV6分别整齐地分为簇L1-13-1和L1-13-2。
3.4 搜索PyIgClassify网站
在PyIgClassify网站上可以进行四种搜索:(i)PDB ID或指定了链的PDB ID; (ii)从列表框中选择的CDR簇(例如L1-11-1); (iii)从列表框中选择的CDR或CDR长度组合(例如L1或L1-11); (iv)IMGT种系群(例如Hu_IGHV1)。 图3和图4分别显示了PyIgClassify的聚类搜索和种系搜索的结果。

PDB ID查询(例如1N8Z(32))将在输入结构中返回CDR和CDR簇的列表。 1N8Z是一种人源化小鼠抗体(hum4D5或曲妥珠单抗),其框架与人IMGT种系序列(Hu_IGKV1_39 * 01和Hu_IGHV3_66 * 02)的94%相同。轻链CDR最接近的种系V区是Mo_IGKV6_17 * 01,重链CDR最接近的种系V区是Mo_IGHV14_3 * 02。该表还包含序列长度,聚类ID,距聚类中位数(°)的距离,序列和Ramachandran构象。
通过PDB搜索的结果(单击群集标识符)或直接搜索CDR群集,用户可以获得该特定群集存在的所有结构,如图3所示的群集L1-11-。 1。可以单击右上角的序列徽标图标以显示较大的图像。点击“仅显示非冗余链”按钮将仅显示代表性序列(每个序列的最高分辨率结构)。 “导出到csv文件”可以将任何PyIgClassify查询结果页面导出到“逗号分隔值”格式的文本文件,该文件可以轻松地解析或导入到包括Microsoft Excel在内的各种程序中。大多数H3循环发生在标有星号的群集中,因为它们的群集效果不佳,例如H3-24- *。这些页面可用于查看每个长度的序列以及它们出现的框架种系。
通过种系搜索有三种选择。 列表框包含在IMGT当前抗体序列中鉴定的所有种系。 用户可以使用该种系组中的框架,该种系组中的CDR或两者来搜索结构。 人类IGKV3序列(Hu_IGKV3)的示例如图4所示。 仅显示该表的CDR1部分。 显示了构架和与那些种系的CDR的种系和序列同一性,以及链中每个CDR的簇。
用户还可以向我们的网站提交序列或PDB格式的结构。 服务器识别输入序列的CDR,或提交结构的CDR和簇,并允许用户下载生成的Honegger-Plückthun重编号的PDB坐标文件(29)。
可以通过单击PyIgClassify主页上的“下载”按钮 http://dunbrack2.fccc.edu/pyigclassify 来下载整个数据库。
四、讨论
近年来,随着PDB中可用抗体结构的数量急剧增加以及对个人抗体库快速测序的能力,许多抗体服务器和数据库已经发布。其中许多努力,例如NEP(35)和Paratome(36)分别集中于抗原表位和paratopes的鉴定。诸如IgBLAST(37)和DigIt(38)之类的服务器已经引入了用于抗体可变域及其相关CDR区序列分析的工具。
类似于PyIgClassify的SAbDab服务器(15)在PDB中提供抗体的CDR构象的聚类。 SAbDab基于具有RMSD度量标准的分层群集,并允许用户以任何输入的RMSD截止值创建群集。如果在我们的2011年论文或Chothia的论文中存在SAbDab集群中的至少一个PDB,则会为每个输出集群提供我们的集群名称以及Chothia的集群名称。因此,SAbDab的输出不同于PyIgClassify,后者基于固定的命名法和聚类方案直接重新编译CDR结构的簇。动态群集具有其优势,但对于PDB中最常见的构象,一组稳定的群集也是如此。 PyIgClassify提供了到聚类质心的二面角距离,可以轻松地识别出聚类的潜在离群值或成员,这些偏离聚类或质心的位置偏离质心太远,无法视为真实成员。
SAbDab提供IMGT子组(例如IGHV1),但不提供完整的IMGT名称(例如IGHV1-69 * 01),也没有单独分析框架和CDR序列,也不提供与种系的序列同一性。它仅提供PDB提供的物种信息,不幸的是,在许多情况下这是不准确的。当VH和VL结构域分别完全是人或小鼠时,PDB中至少有150条抗体链被标记为小鼠或人。在某些情况下,物种名称可能属于恒定域,而不是V区。 SAbDab没有指定IMGT种系亚组的种类。这是有问题的,因为人类和小鼠(及其他物种)种系亚组的编号方式不同。例如,人IGKV1最接近鼠标IGKV16和IGKV10,与鼠标IGKV1的距离很远。因此,PyIgClassify提供了有关CDR簇与IMGT种系信息的关联的完整而准确的信息。
最后,我们开发PyIgClassify数据库的目的是提供适合于预测抗体结构以及更重要的是抗体计算设计的信息。我们认为,大型簇中的序列变异提供了足够的信息,可用于指导设计程序,如Rosetta(39)来采样与PDB中代表性良好的结构簇兼容的氨基酸类型,这一原理已被使用。用于其他蛋白质家族(40)。此外,准确的种系分配可以检查给定种系框架及其CDR上的序列和结构变异,可用于对具有相同种系或种系的特定起始抗体进行序列改变。为了启用这些类型的项目,所有数据都可以从PyIgClassify网站下载。
我们感谢Greg Adams和Matthew Robinson进行的有益的讨论。
参考资料
- https://academic.oup.com/nar/article/43/D1/D432/2437503 .PyIgClassify: a database of antibody CDR structural classifications
