【4.4.8】去除TCE的网页工具
官网:http://tools.iedb.org/deimmunization/
针对蛋白质治疗剂的有害免疫反应会降低功效或导致不良反应。 T细胞反应是这种反应发展的关键,并且针对蛋白质序列中的免疫优势区域,通常与结合HLA II类分子的几个等位基因变体(混杂的结合物,promiscuous binders)有关。在此,我们基于先前对促红细胞生成素蛋白免疫原性的研究,报告了一种新型的预测“去免疫”肽的计算策略。该算法(或方法)首先预测目标蛋白序列内的混杂(promiscuous)结合区域,然后识别预测减少HLA结合的残基取代。此外,该方法预期了任何给定取代对侧翼肽的影响,从而规避了新生HLA结合区的产生。作为原理的证明,该算法被应用于Vatreptacogα,这是一种工程蛋白VII分子,具有意想不到的免疫原性。该算法正确预测了包含工程残基的两种免疫原性肽。作为进一步的验证,我们选择并评估了预计会同时降低两种肽的HLA结合的七个取代的免疫原性,五种对照替代,且没有预期的HLA结合能力降低,以及其他侧翼区域对照。在预测有HLA结合减少的肽培养物中有21·4%和在侧翼区域中有11·4%的培养物中检测到体外免疫原性,而预测有免疫原性的肽培养物中则有46%。该方法已实现为交互式应用程序,可从 http://tools.iedb.org/deimmunization/ 免费在线获得。
一、前言
天然和工程蛋白被广泛用作治疗各种疾病的药物。使用中经常遇到的问题之一是蛋白质,无论是自身来源还是非自身来源,都经常与某种程度的潜在免疫原性有关。反过来,这种免疫原性可能与功效降低和/或潜在的安全性问题有关。因此,开发预测潜在免疫原性的能力,提供降低蛋白质药物潜在免疫原性的策略已成为广泛研究的课题。包括De Groot和他的同事在内的几个小组就此课题进行了研究并产生了宝贵的贡献。
几个不同的变量会影响蛋白质的免疫原性,包括它们形成聚集体的倾向,与自身的差异程度,抗原呈递细胞的摄取以及众所周知,HLA II类结合是T的必要条件(尽管不足)。细胞免疫原性,以HLA结合表位的形式表示。 CD4 T细胞反应是由HLA II类分子介导的,并参与对外源给药蛋白质的反应。因此,在蛋白质免疫原性方面,HLA II类结合是一个特别相关的变量。
如其他地方所述,有几种方法可用于测量和预测HLA结合。在各种方法中,突出的是使用定量分析中的纯化HLA分子直接测量肽结合能力,以及使用矩阵,神经网络和结合蛋白预测结合。 对于某些HLA II类等位基因,相对于I类或更与当前研究相关的与免疫原性相关的测量或预测结合阈值的研究相对较少。
在临床环境中,基于HLA结合的预测和减少免疫原性方法的应用要求开发和验证解决HLA异质性的策略。 HLA II类分子由四个主要基因座编码,即DRB1,DRB3/4/5,DQ和DP,每个基因座都是多态的。存在成千上万种不同的HLA变体,这种高度的多样性反映在一般的大多数人群中,尤其是在患者人群中。 在一系列不同的研究中,我们表明,由每个基因座编码的HLA分子,在其肽结合库方面经常在基因座间和基因座内(both inter‐locus and intra‐locus)具有显着程度的重叠。 确实,无偏聚类分析表明,HLA II类可分为几种主要的超型结合特异性。结合多个HLA等位基因(混杂表位)的肽表位在一般人群中占大多数响应。
以前我们已经表明,关注46个HLA II类分子的一个子集,在世界范围内一般人群中最多代表,这使我们能够表达约90%的HLA变体。我们进一步表明,27个等位基因的一个子集还可以覆盖绝大部分的HLA分子。此外,基于计算一组七个原型等位基因(seven prototype alleles)的中位HLA结合的方法(“ 7-等位基因方法”)被证明可有效预测人群水平的免疫原性。在这里,我们 评估可预测降低的免疫原性的不同方法,描述在不同修饰中进行选择的方法的实现,并提供免费的在线工具来实现此目标。
二、材料和方法
2.1 训练数据集
训练数据集是从先前发表的促红细胞生成素(EPO)脱免疫研究中提取的。在该研究中,从EPO中产生了具有10个残基重叠的15mer肽,并在5个供体中进行了测试。根据白介素-2(IL-2)ELISPOT分析和与15个选定等位基因的结合亲和力,确定了两个区域具有高度免疫原性。将不同的氨基酸取代应用于这些区域,并选择免疫原性最低的肽。
因此,EPO数据集由两组肽组成。第一组与蛋白质中免疫原性区域的鉴定有关,包括跨越整个EPO序列的31个肽(15聚体)。在这31种肽中,有4种在≥50%的供体中具有免疫原性。这四个肽段跨越两个区域,包括残基91–120(区域1)和136–150(区域2)。
第二组肽属于非免疫原性变体的选择,Tangri等人。探测了区域1的12个类似物(靶向位置101-115)和区域2的9个类似物(靶向位置16-150)赋予降低的免疫原性的能力。每种类似物的序列和免疫原性见表1。
2.2 预测方法选择
我们应用了免疫表位数据库(IEDB)推荐的方法(共识,参见参考文献52)来预测HLA II类对三组不同等位基因的结合能力:
- EPO文件中描述的相同的15个等位基因,
- Paul等人描述的七个等位基因子集
- 26个最常见和/或最具代表性的等位基因,在本文中被称为26等位基因方法。
七等位基因方法基于中位数百分位数预测的七个等位基因的结合来预测免疫原性,这七个等位基因代表了普通人群中最常见的结合基序。
2.3 绩效评估参数
通过计算马修相关系数(MCC)和接收器工作特性曲线(AUC)下的面积以及以下参数,评估了不同预测方法的性能:
TP,非免疫原性肽被预测为非免疫原性
FP,预测为非免疫原性的免疫原性肽
TN,预测为免疫原性的免疫原性肽
FN,非免疫原性肽被预测为具有免疫原性
灵敏度(Sensitivity),非免疫原性肽中正确预测的比率
特异性(Specificity),免疫原性肽中正确预测的比率
准确性(Accuracy)正确预测免疫原性和非免疫原性肽的比例。
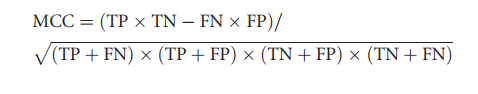
2.4 开发在线工具
用于自动管道的Python脚本分为两个部分,其中第一部分选择蛋白质中预测的混杂结合区域,第二部分应用并评估各种氨基酸取代对所选区域/肽的预测作用。 这些脚本是使用Django平台( https://www.djangoproject.com/ )编写和实现的。
2.5 实验环节
。。。
三、结果
3.1 在EPO的两个主要免疫原区域上的各种预测策略的等效性能
先前的一项研究评估了EPO在体外对人T细胞的免疫原性,确定了两个主要的免疫原性区域。然后,这些区域的修饰显示出降低了整个蛋白质的免疫原性。在该研究中,进一步证明了这两个主要区域 可以根据对15种常见HLA分子的混杂结合亲和力来确定免疫原性区域。 图1(a)显示了先前报道和发表的EPO免疫原性。 EPO数据来自Tangri等。 就我们所知,选择它们是因为它们是唯一的数据集,其中还设计了用于较低HLA结合的给定蛋白质类似物的免疫原性。
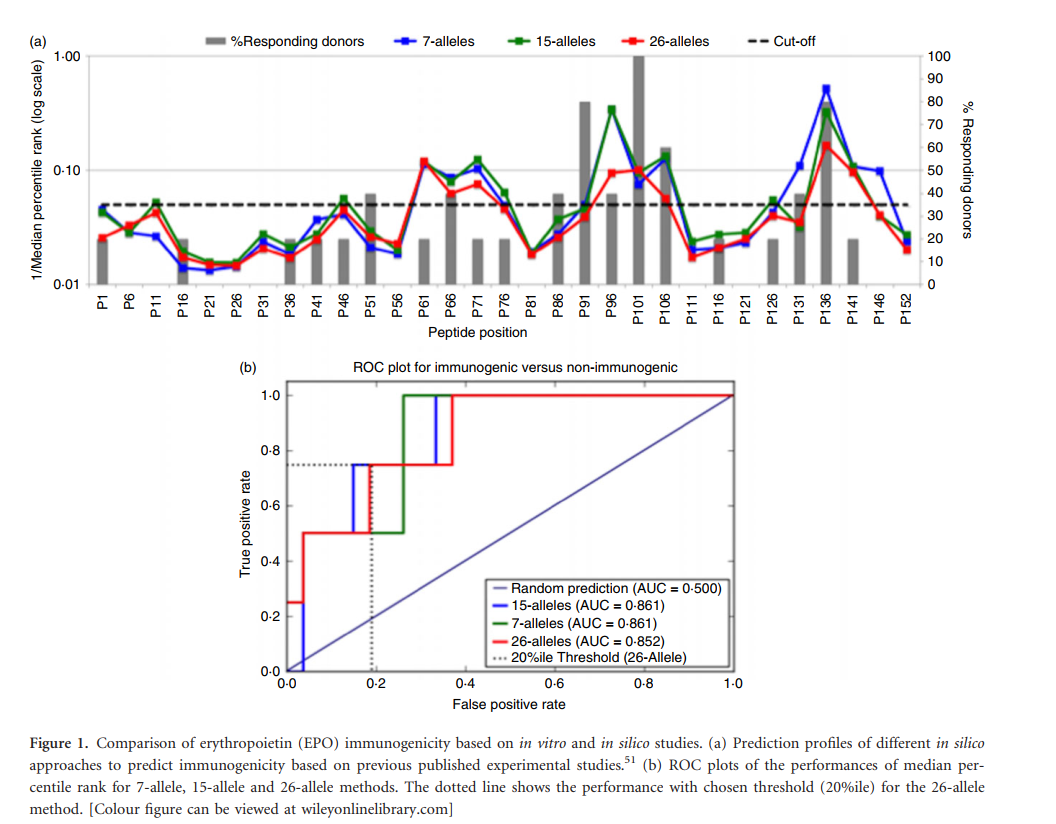
在这里,我们要评估几种常用的预测方法,以鉴定它们识别这两个主要免疫原性区域的能力。首先,我们使用“ IEDB推荐”方法来预测上述EPO研究中使用的相同15个等位基因的各种肽的HLA结合亲和力。此外,我们评估了在普通人群中最常见的26个等位基因组的预测和最近描述的7个等位基因子集(“ 7个等位基因方法” )的预测效力,以鉴定免疫原性最高的蛋白区域如材料和方法中所述。
为了可视化各种方法的结果,图1(a)显示了最初使用的15个等位基因,7等位基因方法和26等位基因组的1 /中位数预测值,每个均覆盖了已发表的免疫原性值。图中的虚线突出显示了1 /中值 0·05,它对应于第20个百分位数的中值,先前被定义为一般人群的广泛免疫原性阈值。
接收器工作特性(ROC)图提供了阈值无关的方法来比较不同模型的功效。根据ROC曲线,可以计算AUC值来表示总体性能。在这种情况下,AUC值为0·5表示随机性能,而值为1则是理想的预测。图1(b)提供了7、15和26等位基因方法的中位数百分位数的ROC图和相关的AUC值。从这些结果可以得出结论,各种方法的性能相似,AUC值在0·852-0·861范围内。
3.2 取消免疫项目中考虑的替代性质 The nature of substitutions considered in de‐immunization projects
先前的研究报告了用于降低免疫原性的产生氨基酸取代的不同策略(有关最新研究,请参见参考文献2、5、9、10、55-57)。在一些研究中,已经使用了丙氨酸替代品(参见参考文献12、25、56、58、59)。丙氨酸替代的基本原理是,它是大多数氨基酸的相对保守或半保守替代,因此被认为不太可能干扰所考虑蛋白质的生物学活性。使用丙氨酸的潜在缺点是变化的温和性质可能不会以足够决定性的方式影响HLA结合。 Tangri等人,提出的另一种方法是引入非保守取代作为干扰HLA结合的靶向策略。在这里,我们查看发布的数据以获得对这些问题的更多见解。
Stern等人[60]的先前研究解决了流感病毒307-319表位的X射线晶体结构以及与T细胞受体(TCR)复合的HLA DR1。已确定9mer核心的1、4、6和9位上的肽残基(对应于肽的309、312、316和319残基)是主要的HLA接触残基,而-1、3和8是主要的TCR接触残基。
独立研究确定了HA 307-319类似物对DR1的HLA结合能力。 如图2所示,丙氨酸取代仅干扰位置1的结合,而不会干扰其他三个锚定位置(即位置4、6)和9)。可以通过以下事实来解释:丙氨酸是第4(Q)和9(L)位天然残基的半保守取代,并且是第6位T的保守取代(丙氨酸取代实际上会增加结合在这个位置)。相反,通过破坏结合超过10倍,非保守取代(K或E)可以正确识别所有四个HLA接触残基。

在T细胞识别的水平上,亚历山大等人研究了丙氨酸取代对大多数位置的影响。同样总结在图2中的结果表明,丙氨酸取代消除了所有三个主要TCR接触处的T细胞识别残基。这些发现不仅限于这种特定的HLA /表位组合,通过在DRB1 * 0401和DRB1 * 0701和HLA DRB1 *15:01背景下的HA肽的X射线结构分析,结合和T细胞活化分析得出相同的结论
总之,这些结果表明,丙氨酸取代是广泛干扰TCR识别的可行策略。但是,由于在大多数情况下都保留了HLA结合,因此,类似药物可能会引发一组具有不同TCR特异性的T细胞。数据表明,丙氨酸取代不是广泛干扰HLA结合的可行策略,非保守取代可能更适合此目的。
3.3 EPO类似物与免疫原性降低相关
Tangri等研究描述了几种单取代替代品,评估了它们对免疫原性的影响。 在这里,我们将这些类似物制成表格(表1),记录其序列和对免疫原性的影响。 为了进行分析,我们定义了免疫原性降低的情况,这种情况是在50%或更多的供体中反应降低了50%以上。
3.4 评估26等位基因方法以预测与免疫原性降低相关的类似物
接下来,我们评估了上述各种方法在表1数据集中预测免疫原性降低的能力。对于每种方法,我们记录了真阳性(TP),假阳性(FP),假阴性(FN)和真阴性(TN)的数量。还计算了MCC,以便在测试的不同方法之间进行比较。当用于验证的数据集与二进制结果相关联时(就实验上降低的免疫原性而言是/否,在预测方面而言是/否),此措施最合适。 MCC的范围是-1至1,其中-1表示完美的负相关,0表示随机分布,而1表示完美的正相关。通常,MCC值为+ 0·70或更高表示非常强的正向关系,+ 0·40至+ 0·69为强的正向关系,+ 0·30至+ 0·39为中等正向关系,而+ 0·20至+ 0·29表示弱正相关。67
初步分析表明,26等位基因方法比其他方法具有更好的性能。因此,作为预测降低免疫原性的标准,相对于野生型抗原决定簇,我们考虑了给定类似物的26等位基因中位数百分位数与野生型序列中位数的差异。分别或合并评估了两个EPO区域。使用不同的中值阈值检查性能时,发现最佳差值为8·5。该临界值是根据经验定义的。表2中的数据用于评估我们的预测模型在EPO的两个免疫原区域中的性能。根据敏感性,特异性,准确性和MCC评估了性能,区域1,区域2和两个区域的MCC值分别为0·5、0·5和0·41。根据这些结果,我们继续实施一种算法,对于任何给定的类似物序列,将自动计算野生型潜在抗原决定簇和所述类似物之间的26等位基因方法值的中位数之差,如更详细所述在以下各节中。
3.5 权衡在侧翼区域引入新表位的潜在影响的方案
A scheme to weigh the potential effect of introducing new epitopes in flanking regions
任何取消免疫的策略不仅必须考虑降低目标表位的HLA结合能力,而且还应避免在相邻序列中意外创建新表位。在这种情况下,15mer肽中的氨基酸取代也可能影响蛋白质序列中最多两个相邻的肽(因为我们正在考虑连续的15mer肽与10个残基重叠的集合)。在某些情况下,即潜在的表位在蛋白质的C或N端时,仅发现一个相邻的15mer,而在分离的肽中,则没有发现相邻的15mer。
为了评估在相邻肽段中创建其他免疫原性区域的潜在影响,我们采用了一种方案,该方案也对相邻的15mers进行了评分(表3)。在这种任意方案中,较低的分数与更优选的类似物相关,尤其是预测对相邻肽无作用或赋予免疫原性降低的类似物。任意分数的范围是1到10,其中最低分数(即1)是不存在任何邻近肽(即15聚体肽进行了免疫接种)的类似物的评分,根据定义,没有邻近肽可以受替代影响。下一个最低分数(2)被授予“最高”情景,即仅存在一个相邻肽,并且此类相邻肽的类似物(C端或N端肽)也被预测具有较低的免疫原性,基于与野生型肽相比更低的中位评分。下一个得分是3,对应于两个相邻肽都存在并且被预测与降低的免疫原性相关的情况。下一个最低的4分是类似物,其中两个相邻的肽中,一个不存在或被预测为具有降低的免疫原性,而另一个在免疫原性上没有变化,依此类推。得分最高的是类似物,在邻近区域中创建了其他免疫原性位点。该方案(表3)用于在以下各节所述的预测工具中对脱免疫预测的结果进行排名和评估。

3.6 开发用于26等位基因方法免疫原性计算的接口
总体而言,去免疫工具包括两步过程(图3):(i)鉴定免疫原性区域和(ii)评估所有类似物以鉴定结合亲和力降低且不影响相邻肽的类似物。 为了在通常可用的蛋白质去免疫工具的背景下促进实施此过程,我们首先开发了一个Web应用程序(图4),该应用程序接受蛋白质或肽的序列并自动生成预计将被识别的特定区域作为输出。 总体水平上的优势表位。 还报告了预测的26等位基因百分位数的中位数。


因此,在输入模块中输入了蛋白质序列,用户可以选择一个阈值/截止值来确定该蛋白质中的预测免疫原性区域(图4a)。 默认阈值为20%,因为先前已将其定义为一般人群的较宽的免疫原性阈值(在体外得出)45,并且在数据情况下还与最佳性能(就MCC而言)相关 来自上述EPO研究。 但是,用户可以应用适合其特定需求或上下文的任何阈值。
我们的预测策略基于固定的中值。 我们先前证明,中值在其他免疫原性预测中表现最佳[50,52],而使用其他参数(例如均值或最小值)可能会使选择偏向于单个“离群值”。 除中位数和固定值外,其他方法也可以在该工具的未来版本中实现。
提交用户输入后,中间页面(图4b)显示通过26等位基因方法预测的潜在免疫原性区域,自动将序列解析为15个10个残基重叠的肽。 该交互式页面允许选择各种预测的肽(15mers)中的一个或多个进行进一步分析,该分析会自动重定向到后续结果页面。
对于EPO序列示例,此页面在默认设置下返回八种肽,包括在Tangri研究中实验分析过类似物取代的两种肽(136–150和101–115)。 这八个中的大多数是连续重叠的15聚体,分别对应于三个大区域:61-85、96-120和136-155。 这些区域中的两个对应于通过实验分析检测到的主要免疫原性区域。
3.8 消除免疫的工具的输出屏幕 Output screen for the de‐immunization tool
该算法会自动计算野生型潜在表位与可能的285个类似物(15个位置×19个可能的取代)中的26个等位基因“ IEDB推荐”方法值的中位数百分等级差异。结果以表格形式汇总,第一行代表野生型肽,第二行代表各种类似物的评分(图4c)。显示了每个类似肽序列的中位数百分等级,以及野生型和类似物之间的百分等级差异。输出还显示了取代对相邻肽(CN1,CN2,NN1,NN2列)的影响。这些列分别代表在C和N末端的(一个/两个)相邻肽(图4c)。在最后一列中,还提供了一个分数,该分数反映了取代对相邻肽的累积作用,其中较低的值更适合于解除免疫。默认情况下,摘要表仅包括相差大于8·5的类似物,但用户可以选择和选择不同的阈值(或多或少严格)(图4c)。根据表3中所示的方案对结果进行排序,然后对26等位基因方法按中位数差异进行排序。
如上所述,该工具以两步方式运行。第一步是鉴定相对较快的免疫原性肽(15聚体少于5秒)。第二步是评估每个15mer的所有可能变体及其侧翼肽(15mer肽的285个变体)。在当前实施中,此步骤大约需要9分钟(准确地说是550·01秒)。考虑到计算的计算强度,我们还提供将结果通过电子邮件发送给用户。
3.9 应用去免疫工具选择免疫原性降低的因子VII类似物
为了说明该程序的实用性,我们使用了Vatreptacog α的序列,这是一种工程化的VII因子分子,由于在人类临床测试过程中由于免疫原性问题而终止了开发过程.在人类中,VII因子序列是自体的,因此在很大程度上不是-免疫原性的。但是,在野生型序列中引入了一些突变以提高药物功效,无意中使药物具有免疫原性。
当Vatreptacog α序列通过去免疫工具运行时,发现26等位基因方法预测了10个肽段,对应于7个区域(因为在196–215、271–290和291–230中预测了两个连续的肽段) 。 Vatreptacog α由野生型序列中的三个突变(V158D,E296V,M298Q)产生。在这三个突变中,预测有两个突变会提高291-310区域的免疫原性,跨越了两个预测的15聚体(291-305位和296-310位置)(表4)。其余六个预测为免疫原性与野生型VII因子序列相同。

因此,我们选择了291–310地区进行实验验证。该区域,特别是嵌套的291–305肽,在10种预测的肽中排名第一,中位值为6·965。但是必须注意的是,并非所有预测的区域都是免疫原性的,可能是因为其他区域是100%自身的,因此由于T细胞耐受性而未被识别。
为了进行进一步的分析,我们选择了覆盖预测的免疫原性区域的两种肽(291–305和296–310),并生成了与降低的免疫原性相关的类似物清单。对于肽291–305,我们鉴定了32种不同的类似物,这些类似物预计与降低的免疫原性相关(表5A)。其中13个代表L297的取代,L296的一个,Q298的两个,L300的七个,L305的八个和R304的取代。对于296-310肽,预测有45个取代具有降低的免疫原性(参见补充材料,表S1)。这些替换对应于七个不同的位置(V297,V299,L300,N301,V302,R304,L305)。
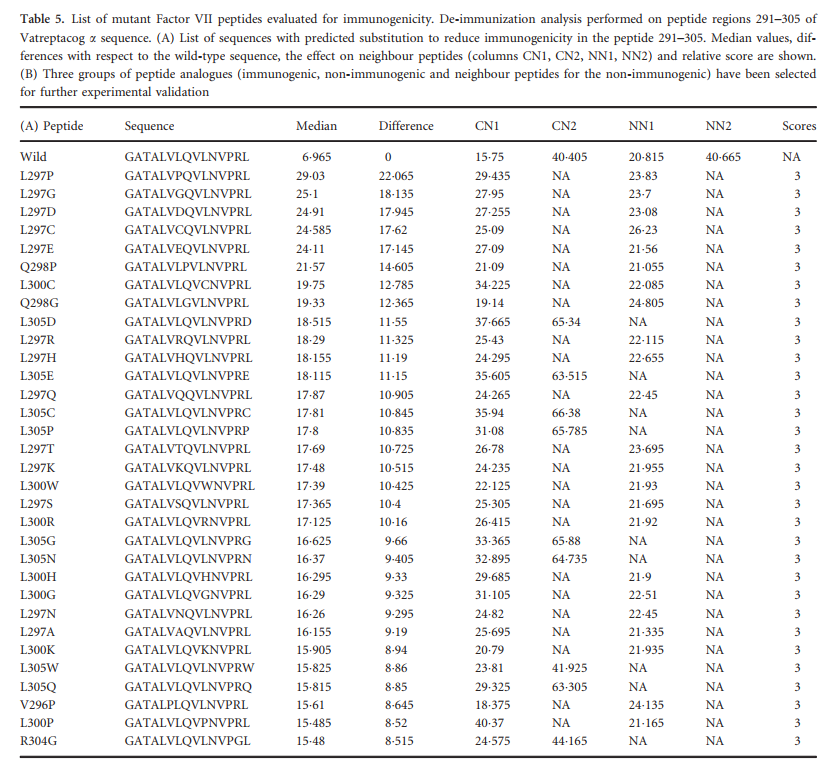
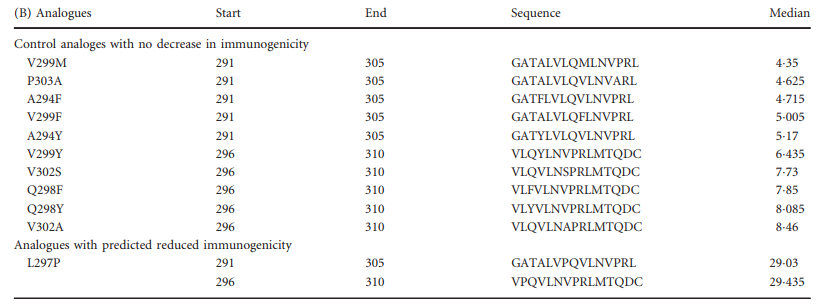
通过仔细研究这两个集合,我们观察到25个不同的取代(见补充材料,表S2)被预测对两个预测的表位都具有有害作用,并且在相邻肽方面也具有有利的特征(标为“ 3”;见表)。 3)。 因此,这25个替换将被预测为最有希望进行进一步测试。 四个位置(L297,L300,R304和L305)占全部25个替换。
3.10 消除免疫的工具的预测的实验验证
为了通过实验验证去免疫工具的输出,我们接下来在体外T细胞免疫分析中合成和测试了一组基于291-305和296-310野生型表位的肽类似物,包括两个相邻的表位肽(一个C末端,一个N末端)(表5B)。该小组包括预测与两种表位的免疫原性降低相关的类似物。这组类似物包含在四个不同位置(297、300、304和305)进行的每个表位的七个取代。另外,对于这七个取代中的每个,我们合成了七个邻近的肽,因为只有一个邻近的肽会受到两个选定的291-305和296-310肽中每个共同取代的影响。最后,作为对照,包括10个代表五个取代的肽,分别在两个不同的帧(291-305和296-310)中(基于增加的中位亲和力)被预测与免疫原性没有降低有关。
使用2周的体外再刺激方案,对来自五名正常供体的人PBMC的所有肽进行了免疫原性测试53,然后进行ELISPOT分析,以测量20小时后分泌干扰素γ或IL-5的细胞数量每个特定的肽刺激。给定测试的供体和肽的数量较少,汇总数据如图5(a,b)所示。补充材料中提供了各个肽段数据的结果(表S3)。在对照肽的情况下,在那些预计具有免疫原性的对照中有46%的培养物中检测到阳性反应,但在免疫原性降低的类似物中只有21·4%和侧翼区域对照中有11·4%(图。 5a)。当考虑响应幅度时,相应的(几何平均值)响应分别为704、445和360点形成单元(图5b)。对照的单尾Mann-Whitney U检验与免疫原性类似物降低相比,应答频率和应答幅度均存在显着差异(图5a,b)。

四、讨论
就我们所知,本报告代表了第一个可自由访问的方法,该方法可自动识别通常针对蛋白质序列(尤其是蛋白质药物)的免疫接种的潜在价值的替代。该工具可通过IEDB的分析资源( http://tools.iedb.org/deimmunization/ )向公众公开,旨在使用户具有灵活性,因为如果用户希望偏离标准,可以选择不同的截止日期。
蛋白质免疫原性在蛋白质免疫治疗中非常重要。T细胞是直接通过超敏反应以及间接提供免疫球蛋白转换和产生高亲和力抗体反应所需的T细胞帮助,来确定蛋白质免疫原性的关键参与者,通过亲和力成熟。 T细胞免疫原性是一个多因素过程,这一事实得到了明确的赞赏。主要因素包括蛋白质的可用性和药效学,免疫细胞的蛋白质摄取和加工,与HLA分子的结合以及对抗原特异性T细胞的合适库的识别。
尽管为了降低免疫原性,可以针对每个因素进行调整,但我们的方法专门针对HLA结合能力。因此,我们强调我们的方法与旨在调节其他变量的方法具有潜在的互补性和协同作用。我们的方法基于以下观念:HLA结合亲和力是T细胞免疫原性的关键条件。几项研究表明,HLA结合对于T细胞免疫原性是必要的(尽管还不够),在这方面,如果HLA结合活性降低或被取消,则免疫原性也相应降低。在先前的研究中,使用EPO作为模型系统,本文的报告也证明了确实如此。
该工具的目的是提供在人群水平上预测免疫原性的能力,不仅包括DRB1基因座,而且还包括先前在7等位基因方法中所述的DP和DQ基因座。该工具未设计为了预测特定等位基因的免疫原性,建议采用完全不同的策略,包括测试HLA匹配的供体和使用HLA特定的预测。为了确保更广泛的HLA等位基因覆盖范围,我们选择了26等位基因方法而不是7等位基因方法,因为它们表现出相同的性能。重要的是,我们发现,显着降低HLA结合能力的最佳替代品是一般的非保守替代品。这与大量的免疫化学知识是一致的,这表明即使在关键的锚定位置引入保守取代也不太可能显着影响结合。就这些取代的潜在用途而言,这具有相关性,并且需要额外的实验测试和/或结构模拟来选择这些取代中的哪些与保留蛋白功能和药物效力兼容。
文献中也报道了其他基于基于将单个氨基酸取代引入已知表位的去免疫研究(例如,参见参考文献2、5、9、10、12、25、55-59,如上所述)。在这些研究中,轻度和保守的替代策略(如“丙氨酸扫描”)被证明是有效的。这些类型的保守取代可能由于干扰TCR识别而有效(并且已常规用于绘制TCR接触残基)。但是,同时,如图2所示,由于保守取代不能可靠且显着地干扰MHC结合,因此它们随后易受新T细胞集合的诱导。相反,我们的方法考虑了能够破坏HLA结合和TCR识别的非保守取代。
同时,尽管保守的替代策略可能引发新一轮的T细胞识别同一表位区域上一组不同的TCR可及残基,但它们可能具有重要的优势,即不大可能干扰蛋白质功能。此外,也有可能使用“低调”取代(例如丙氨酸)来缓解此问题,因为已证明HLA II类限制性T细胞通常集中在大型且带电荷的氨基酸侧链上。
需要强调的是,改变HLA结合能力不能解决T细胞耐受性的中心问题。 T细胞通常对自身蛋白序列具有耐受性或非反应性,因此,对自身蛋白序列的任何修饰都可导致免疫原性增加,因为该分子被认为是非自身分子,并被免疫检查迅速识别。有鉴于此,应谨慎使用完全自我分子的蛋白质修饰,并应同时进行自我/非自我状态的确定。
这是我们方法的关键特征,因为它提供了可以在一般人群中实施的可行策略。存在用于预测在不同基因座表达的数百个不同等位基因变体的结合能力的算法,但是寻找同时影响如此大量等位基因的替代是不切实际的。此外,必须在全球人口水平上考虑不同替代方案的影响。先前在单等位基因或遗传近交动物模型中进行的几项研究提供了证据来支持减少结合可以降低免疫原性的概念。但是,通常需要具备“跳入”与高度异质性相关的人类患者群体的能力,尤其是在HLA II级的水平上。先前的研究一方面定义了代表HLA多样性的等位基因,另一方面定义了在HLA II类分子的肽结合特异性水平上存在的广泛库汇合。我们的方法提供了一种识别候选基因的策略氨基酸取代可降低免疫原性,普遍适用于异种群体。
参考资料
- Sandeep Kumar Dhanda, Alba Grifoni, John Pham, Kerrie Vaughan, John Sidney, Bjoern Peters, Alessandro Sette (2017). Development of a strategy and computational application to select candidate protein analogs with reduced HLA binding and immunogenicity. Immunology。 https://onlinelibrary.wiley.com/doi/full/10.1111/imm.12816
