【4.5.8】基于序列的抗体经典环结构注释SCALOP
scalop: Sequence-based antibody CAnonical LOoP structure annotation.
- SCALOP是抗体上六个互补决定区域(H1,H2,L1,L2和L3)中五个区域中的五个的基于序列的 canonical form预测器。
- 序列将由ANARCI编号,并由SCALOP分配为规范形式(canonical form)。
- SCALOP不能检测到冲突,也不能预测嫁接环中的侧链构象。 请使用ABodyBuilder生成完整的模型。
官网:
http://opig.stats.ox.ac.uk/webapps/newsabdab/sabpred/scalop
一、安装和使用
依赖的环境:
- Python 2.7+
- HMMER3 (version 3.1+ recommended)
安装
cd /data/software/scalop
tar -zxvf scalop-1.0.tar.gz
cd scalop-1.0
python setup.py install
(安装到了 /data/software/anaconda2/lib/python2.7/site-packages/scalop;数据库在/data/software/anaconda2/lib/python2.7/site-packages/scalop/database 下面)
如果没有root权限,可以这么来
python setup.py install --user
python setup.py install --home="~"
例子:
SCALOP --numbering_scheme imgt --cdr_definition north -i EVQLQQSGAEVVRSGASVKLSCTASGFNIKDYYIHWVKQRPEKGLEWIGWIDPEIGDTEYVPKFQGKATMTADTSSNTAYLQLSS......LTSEDTAVYYCNAGHDYDRGRFPYWGQGTLVTVSA -o result
参数例子;
--numbering_scheme {imgt,chothia}
--cdr_definition {north,imgt,chothia}
二、文献解读
摘要:
- 动机:抗体互补决定区(CDR)的规范形式于1987年首次描述,此后多次被重新定义。 由于可以从序列中预测,规范形式( canonical forms)通常用于近似抗体结合位点的形状。 快速预测器将有助于在现在可从下一代测序实验中获得的大量库数据中注释CDR结构。
- 结果:SCALOP注释抗体序列的CDR规范形式,并由自动更新数据库支持以捕获最新的簇信息。 它的准确性可以与标准结构预测器相媲美,但速度要快800倍。 SCALOP的自动更新特性可确保始终获得最佳覆盖范围。
- 可用性:SCALOP可以作为Web应用程序使用,并可以在 opig.stats.ox.ac.uk/webapps/scalop 的GPLv3许可下下载。
2.1 前言
抗体是免疫系统中与外来分子结合的蛋白质。结合位点主要由六个互补决定区(CDR)组成:每个重链和轻链上三个。构象簇(Conformational clusters)被称为“规范形式”(canonical forms),已在六个CDR中的五个中观察到(例如Chothia和Lesk,1987; North等,2011; Nowak等,2016)。规范形式已在文献中多次重新定义,但是每次更新都是可用数据的静态快照。这些不断的更新说明了结构数据的增长是如何不断修改我们对CDR环结构的理解的,1987年有10种规范形式(Chothia和Lesk,1987年),到2016年有26种规范形式(Nowak等,2016年)。
已经开发了几种基于序列的规范形式预测方法(例如Chothia和Lesk,1987; North等,2011; Nowak等,2016; Long等,2018)。 Chothia和Lesk(1987)提出了结构上确定标准形式分配的残基。使用类似的方法,Swindells等(2017)发布了一个可免费使用的Web服务器,该服务器可处理批量canonical forms分配,但某些cluster缺乏代表性的结构。还为cluster分配建立了隐马尔可夫模型(North等,2011; Nowak等,2016)。最新发布的方法使用梯度增强机(Gradient Boosting Machine)以高达85.1%的精度注释CDR主链构象(Long等人,2018)。但是,这些工具都没有使用自动更新的数据库,也没有提供用于大规模序列分析的Web界面和免费提供的软件包。
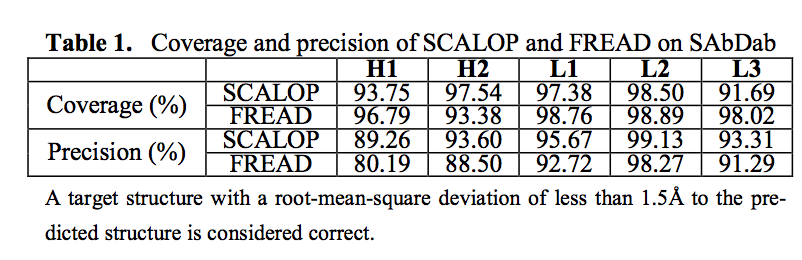
在这里,我们介绍了SCALOP,它在自动更新数据库中将H1,H2,L1,L2和L3 CDR聚类,并创建canonical form预测工具。 SCALOP可用于从单独序列(Krawczyk等人,2018)快速估计抗体结合位点的形状,最低准确度为89.47%(表1;补充材料)。 该工具可作为Web服务器使用,也可作为Python程序包用于批量处理。
2.2 算法
SCALOP将完整抗体链的一个或一组氨基酸序列作为输入。 然后用ANARCI对该序列编号(Dunbar and Deane,2016),并对提取的CDR序列针对适当簇的PSSM进行评分。 簇(cluster)命名法遵循Nowak等人的命名法 (2016年;补充材料)。 然后将输入CDR序列分配给具有最高得分高于得分阈值的cluster(补充材料)。 SCALOP返回分配的cluster的名称,并返回分配l类群的中间结构的PDB代码和链标识符。 如果框架的结构与CDR序列(补充材料)一起给出,则SCALOP可以返回结构模型。 该数据库每月更新一次,以前的数据库可从网站上获得。
2.2.1 建立PSSM
我们采用了Nowak等人开发的长度无关的CDR聚类方法。 (2016)。 使用截至2017年7月10日可用的SAbDab中的结构(Dunbar等,2014)(补充材料)。 我们仅使用每个cluster的唯一序列为其构建了PSSM:
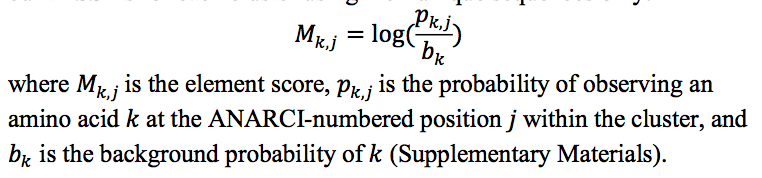
其中,Mk,j是元素评分,pk,j是在簇内ANARCI编号位置j观察到氨基酸k的概率,并且bk是k的背景概率(补充材料)。
2.2.2 Cluster assignment

2.3 Benchmark
我们使用留一法交叉验证协议(表1)和盲测集(补充材料)在训练集上评估了SCALOP的性能。 两者都取得了相似的结果。 我们还比较了FREAD的改进版本,FREAD是一种用于循环结构预测的准确数据库搜索方法(Deane和Blundell,2001年; Krawczyk等人,2018年;补充材料)。 此版本不会生成结构模型,但会返回其预测的PDB代码。 该方法的预测范围和精确度具有可比性(表1;补充材料)。
为了评估SCALOP和FREAD做出的预测的速度和一致性预测的一部分,我们在下一代测序数据集上运行了这两个预测因子,这些序列具有约800万个轻链和约500万个重链序列(Krawczyk等人,2018)。 两种方法(补充材料)中有98%的预测是一致的。 在单核上,使用FREAD预测100个序列需要227s,而使用SCALOP则需要0.29s。 这种快速的预测表明将SCALOP作为快速可靠的第一屏运行的可能性。
为了确保SCALOP始终提供最佳的覆盖范围,它使用自动更新数据库。 图1展示了使用L3作为示例的这种自动更新方法的优势。 我们根据规范形式定义的先前发布日期选择了代表年份(Al-Lazikani等,1997; North等,2011; Nowak等,2016)。 使用了直到年底的数据,即2016年的数据,使用了2016年底之前存放在SAbDab上的所有结构。 在1997年,只有一个L3集群。 到2016年,只有七个,非聚集数据的份额减少了一半以上。 使用1997年的数据集进行预测,我们可以达到与2017年的数据类似的精度(1997年为97.4%,2017年为94.0%),但覆盖范围却减少了约30%。
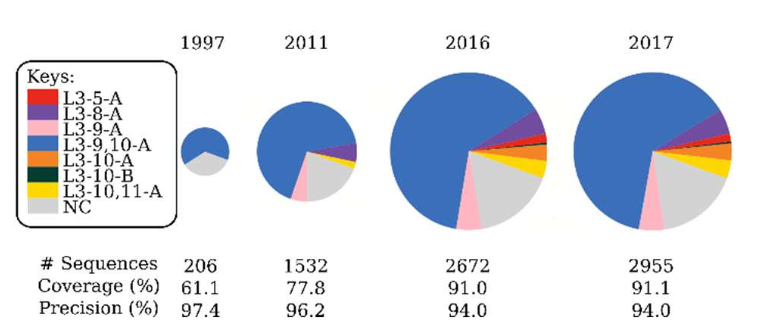
图1.过去20年中L3聚集的变化。 饼形图的半径与log(序列数)成正比。 1997年,仅存在一个L3集群,其成员均为length-9环。 2011年,存在四个簇,覆盖不同的序列长度。 在2011-2016年之间,一些长度为10的序列加入了2011-L3-9-A集群,该集群成为2016-L3-9,10-A集群。 丰富的知识可在保持精度的同时提高SCALOP的预测范围。 饼状图下方的数字是截至2018年7月1日的所有抗体的免检交叉验证(如有需要)(补充材料)。
参考资料
- Wing Ki Wong, Guy Georges, Francesca Ros, Sebastian Kelm, Alan P Lewis, Bruck Taddese, Jinwoo Leem, and C. M. D. (2018). SCALOP: sequence-based antibody canonical loop structure annotation. Bioinformatics, (May), 0–0. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6513161/
