【8.1.1.2】选择性肿瘤特异性抗原(Alternative tumour specific antigens)
基于新抗原的治疗性疫苗的历史背景:
鉴定单核苷酸变异(SNV)新抗原作为抗肿瘤免疫的靶标是理解肿瘤特异性抗原(TSA)疫苗疗法的重要的第一步。这一过程始于 SNV 新抗原可用于开发治疗性疫苗和细胞模式的理论。随后,SNV 新抗原治疗性疫苗的概念验证在临床前肿瘤模型中得到证实,为新抗原临床试验提供了框架:
- 小鼠模型中非同义体细胞点突变的识别和描述产生了候选靶点
- 肿瘤新抗原作为免疫检查点抑制剂疗法激活的 T 细胞的靶标起作用
- 外显子组和质谱相结合的方法鉴定出新抗原
- 小鼠肿瘤新抗原的表征表明,大部分识别是由 CD4 + T 细胞提供的
最近,人类新抗原治疗试验已在以下背景下进行:
- 黑色素瘤中的树突状细胞、肽和DNA 新抗原疫苗
- 低突变胶质母细胞瘤的新抗原疫苗
The most commonly studied class of TSAs are those derived from non- synonymous single- nucleotide variants (SNVs), or SNV neoantigens。最近对“替代 TSA”(此处定义为来自非 SNV 基因组来源的高特异性肿瘤抗原)进行了评估。这些选择性 TSA 包括源自突变移码、剪接变体、基因融合、内源性逆转录因子和其他过程的抗原。

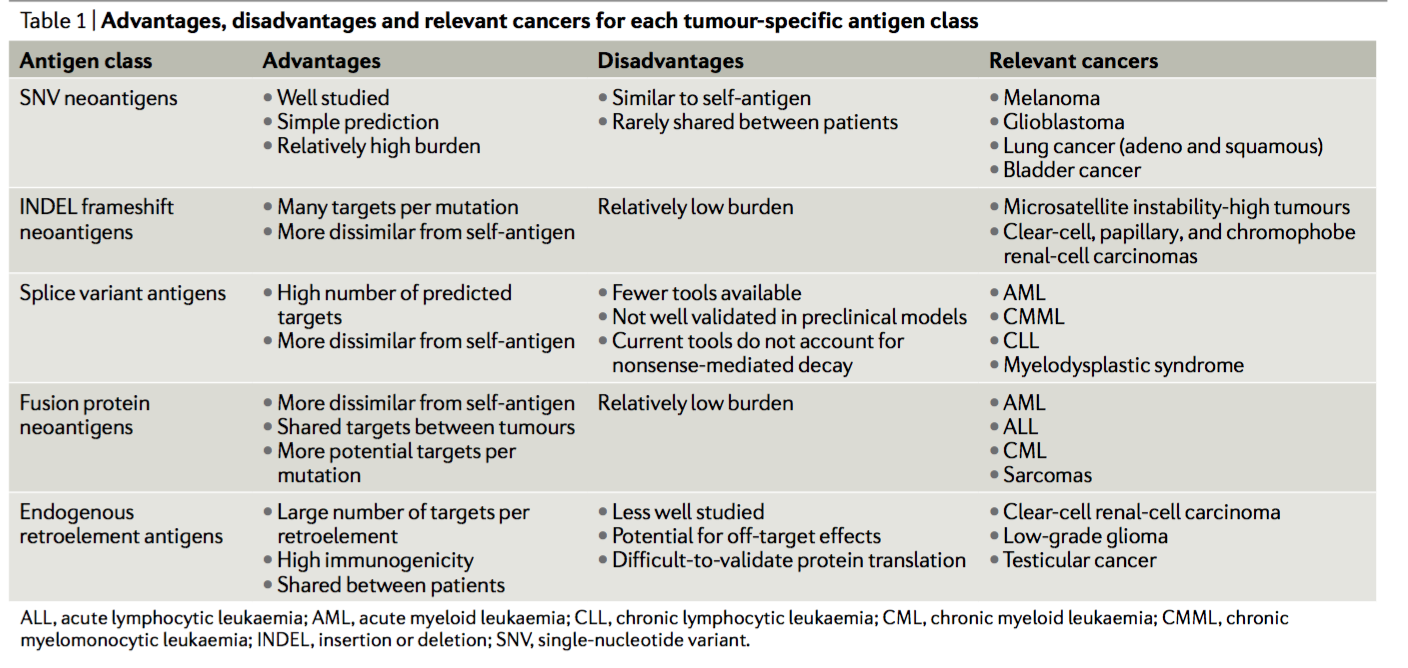
癌症基因组图谱 (TCGA) 癌症类型所识别的独特表位的数量
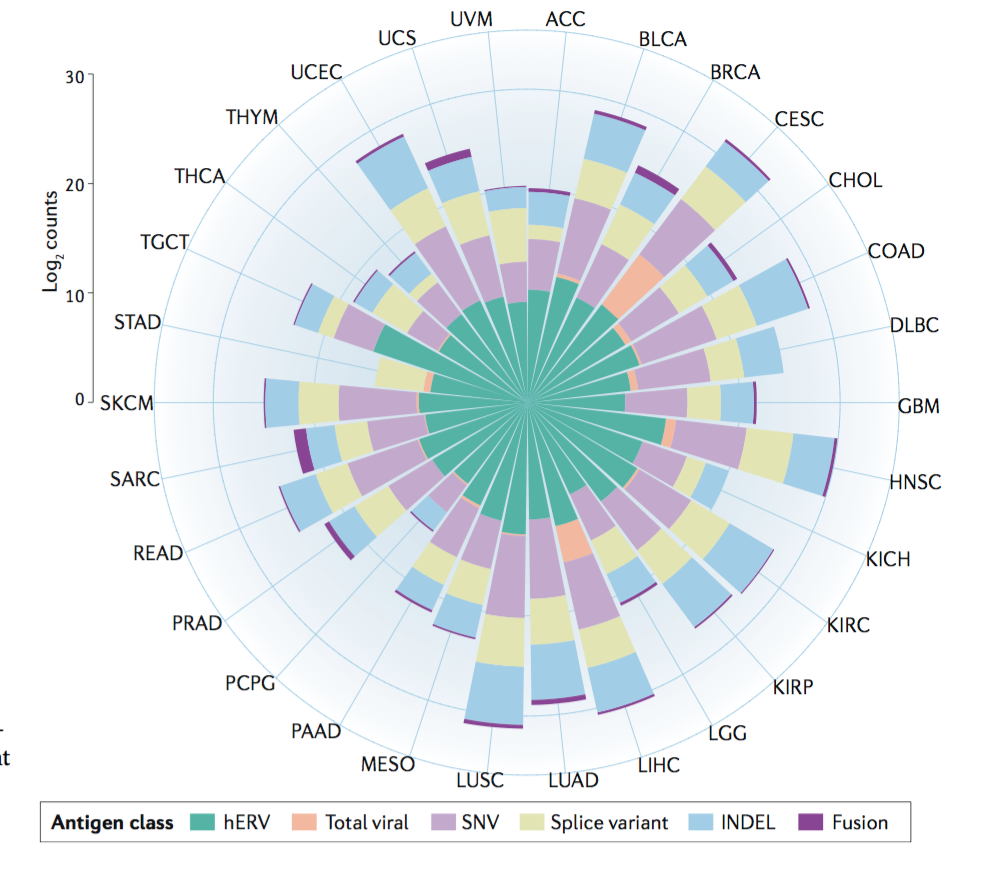
一、突变移码新抗原 Mutational frameshift neoantigens
INDEL 突变源自基因组中碱基对的插入或删除,能够产生非同义的新型开放阅读框,称为突变移码。INDEL 衍生的新抗原已被假设(但尚未证实)比 SNV 衍生的新抗原产生更强大的免疫反应,因为它们的序列与 INDEL 下游的种系序列完全不同。
同行评审出版物中至少有六种工具能够预测 INDEL 衍生的新抗原:pVACseq 、Neopepsee 、MuPeXI 、Epidisco 、Antigen.garnish 和 TSNAD
二、剪接变体抗原 Splice variant antigens
剪接变体抗原是由选择性剪接事件产生的转录后衍生的 TSA,包括 mRNA 剪接点突变、内含子保留或基因失调的那些肿瘤细胞中的剪接体机制。其他类型的转录后衍生 TSA 包括替代核糖体产物(例如,核糖体移码,非规范起始,终止密码子通读,反向转录和双联体解码 ) 和翻译后剪接 ,这两种机制很难应用于抗癌治疗,因为缺乏预测此类产物的工具。
两个工具,Spliceman 和 MutPred Splice ,预测带注释的剪接点周围的外显子变异干扰正常剪接的能力。其他工具提供选择性剪接事件的从头识别,包括 JuncBase 、 SpliceGrapher 、 rMATS 、 SplAdder 和 ASGAL 。其中许多工具(例如,SpliceGrapher、SplAdder 和 ASGAL)通过生成拼接图来预测替代拼接事件。
三、基因融合新抗原 Gene fusion neoantigens
尽管与 SNV 和 INDEL 突变相比,总体基因融合频率相对较低,但它们可以在不同肿瘤类型内部和之间共享,从而使它们可以通过靶向方法(例如荧光原位杂交)进行识别,并可能通过通用方法进行靶向(与患者特异性)基于新抗原的策略。
还开发了几种个性化基因融合新抗原识别管道,包括专为预测基因融合新抗原而设计的 INTEGRATE-neo
还描述了其他几种用于基因融合新抗原识别的工具,包括 pVACfuse(使用 INTEGRATE-neo 报告的融合变体执行新抗原表位识别)、NeoepitopePred 、Antigen.garnish 和Epidisco
四、内源性逆转录因子抗原 Endogenous retroelement antigens
。。。
五、其他选择性TSA
HLA体细胞突变衍生的新抗原
一些研究已经描述了肿瘤 HLA 中的体细胞突变,导致 T 细胞识别发生改变。发布的基于全外显子组的 HLA 分型软件 POLYSOLVER,能够以高预测能力调用 HLA 体细胞突变,并通过 RNA-seq 进行验证(估计灵敏度 94.1%,特异性 53.3%)。最近,HLAProfiler 改进了 HLA 体细胞突变调用的广度和准确性,并且能够仅根据 RNA-seq 数据进行工作 铁汉 10:15:00
翻译后 TSA
TSA 可以在肿瘤中翻译后出现,有可能成为治疗靶点,但用当前的计算工具很难预测它们。翻译后剪接可能发生在人类癌症中,导致多肽片段切除,随后将游离羧基末端与新肽的氨基末端连接。此外,第二类抗原,称为与肽加工受损相关的 T 细胞表位 (TEIPP,T cell epitopes associated with impaired peptide processing),被描述为存在于参与抗原加工 (TAP) 缺陷、MHC 低肿瘤的转运蛋白上,并被TEIPP特异性T细胞群。有趣的是,这些表位未突变且源自管家基因。然而它们并不存在于正常细胞上。TEIPP 特异性 T 细胞可以在野生型小鼠中逃避胸腺选择(但在 TAP1 缺陷型小鼠中则不然),这使它们成为抗肿瘤治疗靶点的有希望的候选者。
六、挑战和未来方向
阻碍替代 TSA 作为治疗方法广泛临床应用的挑战之一是需要提高表位预测的灵敏度和准确性。上述计算方法提供了从更广泛的基因组来源预测更多 TSA 的途径。然而,这些算法的上游和下游方法通常可用于提高所有 TSA 类别的预测性能。在这里,我们重点介绍几种可能普遍提高所有 TSA 预测的数量和准确性的策略:改进 MHC 表位结合预测、直接预测 TSA 生成和免疫原性的算法,以及提高 TSA 识别准确性的质谱方法。
6.1 MHC epitope calling
迄今为止,大多数 TSA 治疗性疫苗研究都集中在使用预测的 MHC I 结合表位,这很大程度上是由于经典假设,即 CD8 + T 细胞在抗肿瘤免疫中比 CD4 + T 细胞发挥更大的作用,并且具有更好的性能MHC I 表位预测算法的性能优于 MHC II 表位预测算法的性能。尽管如此,MHC I 和 II 预测算法都需要进一步改进,以识别更多数量的准确预测的 TSA。我们小组最近的一项分析表明,NetMHCpan 的 MHC I 结合亲和力预测的准确性因等位基因类型的不同而有很大差异,性能测量与训练数据表位“结合者”的比例密切相关(K D ≤ 500 nM, MHC 结合领域内普遍接受的截止值),而每个等位基因的总训练数据量则较少175。因此,训练集中“结合物”较少的等位基因的敏感性和特异性较差,这表明将 MHC I 预测因子应用于临床 TSA 预测需要更多高质量的数据。
关于MHC II预测,最近的临床前和临床研究表明MHC II结合新抗原在促进抗肿瘤免疫方面的重要性。Kreiter 等人的一项研究。是第一个描述 MHC I 预测的新抗原实际上呈现在 MHC II 上,随后触发 CD4 + T 细胞反应。Tran 等人的早期研究进一步支持了SNV 特异性 CD4 + T 细胞在抗肿瘤免疫中的相关性。输注 ERBB2 相互作用蛋白 (ERBB2IP) 突变特异性 CD4 + T 细胞群可在转移性胆管癌患者体内消除肿瘤生长 35 个月。Sahin 等人的临床研究。证实了 CD4 + T 细胞反应在人体试验中的重要性,为支持 MHC II TSA 的临床重要性提供了证据。尽管有这些证据,MHC II 表位的计算预测方法面临的一个主要障碍来自 MHC II 复合物的开放结合裂口结构。与 MHC I 相比,这种结构导致表位结合相对混杂,因此必须准确预测较长 II 类表位的结合核心,然后才能计算结合亲和力
MHC II 表位结合亲和力的计算预测最近取得了进展,这在很大程度上得益于在大型、经过验证的表位数据集上训练的机器学习算法的应用。许多早期算法侧重于表位结合核心的识别,并根据该肽核心与 MHC 复合体之间的相互作用进行预测。Neilson 及其同事首先描述了 NN-align,它提供了针对肽核心和侧翼区域特征进行训练的 MHC II 结合预测,从而显着提高了 MHC II 结合预测性能。尽管表位的结合亲和力主要由其肽核心决定,但侧翼区域特征也会影响结合亲和力。NN-align 后来被 Andreatta 等人改编为 NetMHCIIpan 的核心算法。这进一步提高了性能,并导致了替代表位-MHC 相互作用的描述。即使对 MHC II 结合预测进行了这些改进,最先进算法的性能特征仍然落后于 MHC I 结合预测器。虽然 MHC II 表位在促进抗肿瘤免疫方面的重要性主要是通过 SNV 新抗原观察到的,但预计替代 TSA 将同样适用于 MHC II 表位。因此,增加替代来源的可用 TSA 的广度和改进 MHC II 表位预测可以共同提供一致的策略,以倍增地增加肿瘤抗原的可靶向库。
6.2 直接预测 TSA 的产生和免疫原性 Direct prediction of TSA generation and immunogenicity
除了 MHC 结合亲和力预测之外,直接预测 TSA 生成和免疫原性的新方法可能有助于治疗表位的临床选择。目前大多数新抗原预测算法依赖于预测的肽-MHC 结合亲和力作为表位筛选的主要方法。然而,临床前和临床研究表明,所有候选新抗原中只有少数能够产生免疫反应。对于这种高假阳性率的一种解释是,当前的结合预测工具没有考虑 MHC 肽加工中涉及的其他步骤1。皮尔逊等人的一项研究。证明 MHC I 类相关肽(MAP;即表位)仅源自 B 淋巴细胞中表达的外显子序列的 10%,其中 41% 的蛋白质编码基因不产生 MAP。利用与有效 MAP 生成相关的转录本和蛋白质的特征,他们生成了一个逻辑回归模型来预测基因是否能够生成 MAP。
改进 TSA 预测的另一种方法是直接预测表位免疫原性。正如我们上面简要提到的,Neopepsee 是一种新工具,它结合了在 HLA 等位基因上训练的机器学习算法,该算法产生 T 细胞反应,以直接预测假定的新抗原的免疫原性。与传统的结合亲和力指标相比,Neopepsee 在两个外部验证数据集中预测了免疫原性,灵敏度和特异性显着提高,为直接免疫原性预测方法提供了支持证据。由于 Neopepsee 算法是在广泛的 HLA 等位基因上进行训练,而不是专门使用 TSA 表位免疫原性,因此训练集的自体新抗原和非自体表位之间的生物学差异可能是算法性能的限制因素。随着越来越多的临床试验收集新抗原免疫原性数据,未来专门针对 TSA 训练的算法可能会提供更好的预测能力。
6.3 质谱方法 Mass spectrometry approaches
除了计算 TSA 预测之外,基于质谱的肽组学方法也已应用于肿瘤抗原的鉴定。通过质谱法鉴定内源性表位始于 20 世纪 90 年代初。第一个肽抗原是通过串联质谱的手动解释发现的;然而,计算方法现在已成为比较串联质谱法和蛋白质组数据库上的肽序列的常规策略。虽然在概念上类似于基因组比对和测序,但串联质谱测序实际上更容易出错且灵敏度较低。使用来自充分表征的生物样品的复杂肽混合物进行的标准蛋白质组学实验通常只能识别蛋白质组学数据库中约 25% 的串联质谱。除了序列识别的计算困难之外,肽还可以在质谱仪中进行重排,产生原始生物样本中不存在的序列。尽管存在这些挑战,使用质谱法确认预测的新抗原仍取得了进展。免疫基因组学方法已用于从肿瘤测序数据生成虚拟肽组,并且已鉴定出新抗原。Laumont 等人最近的一项研究。193使用质谱方法观察到,从两种小鼠癌细胞系和七个人类原发性肿瘤中鉴定出的 TSA 中,约 90% 源自非编码区域,包括内含子、替代阅读框、非编码外显子、非翻译区域-外显子连接、结构变异和内源性逆转录因子。值得注意的是,这些非编码区并未通过当前的外显子组或基于转录组的测序方法来识别。这项研究强调了替代 TSA 的潜在重要性,并为其在治疗设计中的应用提供了强有力的证据,重要的是证明经典 SNV 新抗原可能只占 TSA 总量的一小部分。虽然这些研究已经实现了大规模新抗原的发现,但串联质谱比对的局限性和意外肽重排的可能性意味着可疑的新抗原应通过直接比较样品的串联质谱与合成肽的串联质谱来确认
参考资料
- Smith, C. C., Selitsky, S. R., Chai, S., Armistead, P. M., Vincent, B. G., & Serody, J. S. (2019). Alternative tumour-specific antigens. Nature Reviews Cancer, 19(8), 465–478. https://doi.org/10.1038/s41568-019-0162-4
