6.2 MetaMed--将微生物群功能与药物治疗联系起来
网页工具:http://metamed.rwebox.com
了解人类微生物组如何影响人类健康对治疗疾病和减少临床研究中的不良副作用具有影响。 在这里,我们介绍MetaMed( http://metamed.rwebox.com/index ),这是一种新颖的,系统性的,全系统的关联映射系统,可将细菌功能和药物疗法联系起来,为深入研究微生物疗法对人类健康的影响提供了新的假设 。 此外,还发现了生活在环境中的微生物与药物之间的全面关系,为发现具有巨大生物应用潜力的微生物群代谢产物提供了丰富的资源。
一、意见/假设
人类微生物群在治疗结果中的重要性已得到公认。破解微生物对人类健康的影响已成为临床研究中的一个具有挑战性的问题(1)。微生物通过各种途径影响人体,例如通过代谢产物的产生和生物活性成分的释放来调节宿主的新陈代谢和免疫力,从而影响健康状况以及疾病的发生和/或发展(1)。尽管这种微生物菌群与宿主相互作用的确切机制是复杂的,并且在很大程度上仍未得到充分探索(2),但通过利用现有药物的现有注释信息将微生物代谢物与现有药物或小分子联系起来,将提供有用的指导和新颖的假设来解密微生物对人体健康的影响(3)。
为此,我们引入了MetaMed(元基因组学医学作图系统),这是一种新颖的,系统性的,全系统的相关性作图系统,用于将细菌功能和药物治疗联系起来。在该系统中,微生物代谢物实体和药物实体之间的明确定义的相似性评分适用于将微生物功能与现有药物疗法联系起来。我们提供全面而可靠的证据,表明这种简单的链接策略可以帮助您准确预测微生物对人体的影响。此外,这样的预测也有助于推导出假说,这些假说将有助于发现具有重大药物应用潜力的微生物群代谢产物。
二、MetaMed的发展
如果微生物代谢物和药物分子具有相似的结构和扰动转录特征,则它们在人体中的功能可能相似。 在我们的研究中,
-
我们首先从MIBiG(4)收集了1,157个微生物生物合成基因簇(BGC,biosynthetic gene clusters)及其代谢产物
-
从DrugBank(5)收集了8,226种药物。 应当注意,MIBiG提供了全面的注释,以指示微生物产生的代谢产物的实验室条件。 还没有与微生物在人体中产生某些代谢产物的状况有关的其他信息。 因此,MetaMed旨在提供微生物产生某些代谢物的潜在能力,并鼓励对其进行进一步研究。
-
然后,我们通过考虑分子结构(6)和扰动转录表达谱(perturbation transcriptional expression profiles)(7)来定义相似性评分,以将微生物功能与可用的药物注释信息联系起来(请参阅补充材料中的文本S1)。
-
我们最终获得了1,193,324对,用于评估代谢物和药物实体之间的潜在联系,并按我们定义的相似性评分进行排名。
-
我们利用KEGG路径注释来证明我们提出的MetaMed评分方案。我们将代谢物和共享至少一个与疾病相关的KEGG途径的药物定义为具有相似的功能,并且将此类药物-代谢物对作为基准中的阳性样品,而其他不共享任何代谢物-药物的对与疾病相关的KEGG通路被作为阴性样本。
我们通过它们的MetaMed相似性评分对所有预测的药物-代谢物对进行排名,并评估高于特定MetaMed评分阈值的预测精度。因此,当MetaMed得分高于0.9(32/32)时,精度为100%。当MetaMed得分超过0.8(45/54)时,精度为83%;当得分超过0.7(95/199)时,精度为48%。当MetaMed得分超过0.6(41/735)时,精度下降到34%。这些数据表明,较高的MetaMed得分代表具有类似药物相关功能的可靠代谢物-药物对预测结果的可能性较高,如KEGG路径注释所示(见图2a)。综上所述,我们的MetaMed相似性评分可以有效地区分高潜力的代谢物-药物联系与阴性联系,可用于有效鉴定具有潜在治疗作用的代谢物。
通过整合来自MIBiG,LINCS,DrugBank,SIDER等的各种现有注释信息,我们进一步将微生物功能与相关药物作用相关联(8、9)(图1;文本S1),从而形成了系统的制图系统MetaMed 包括以下链接:微生物药物,微生物治疗适应症,微生物副作用以及微生物免疫状态的转变。
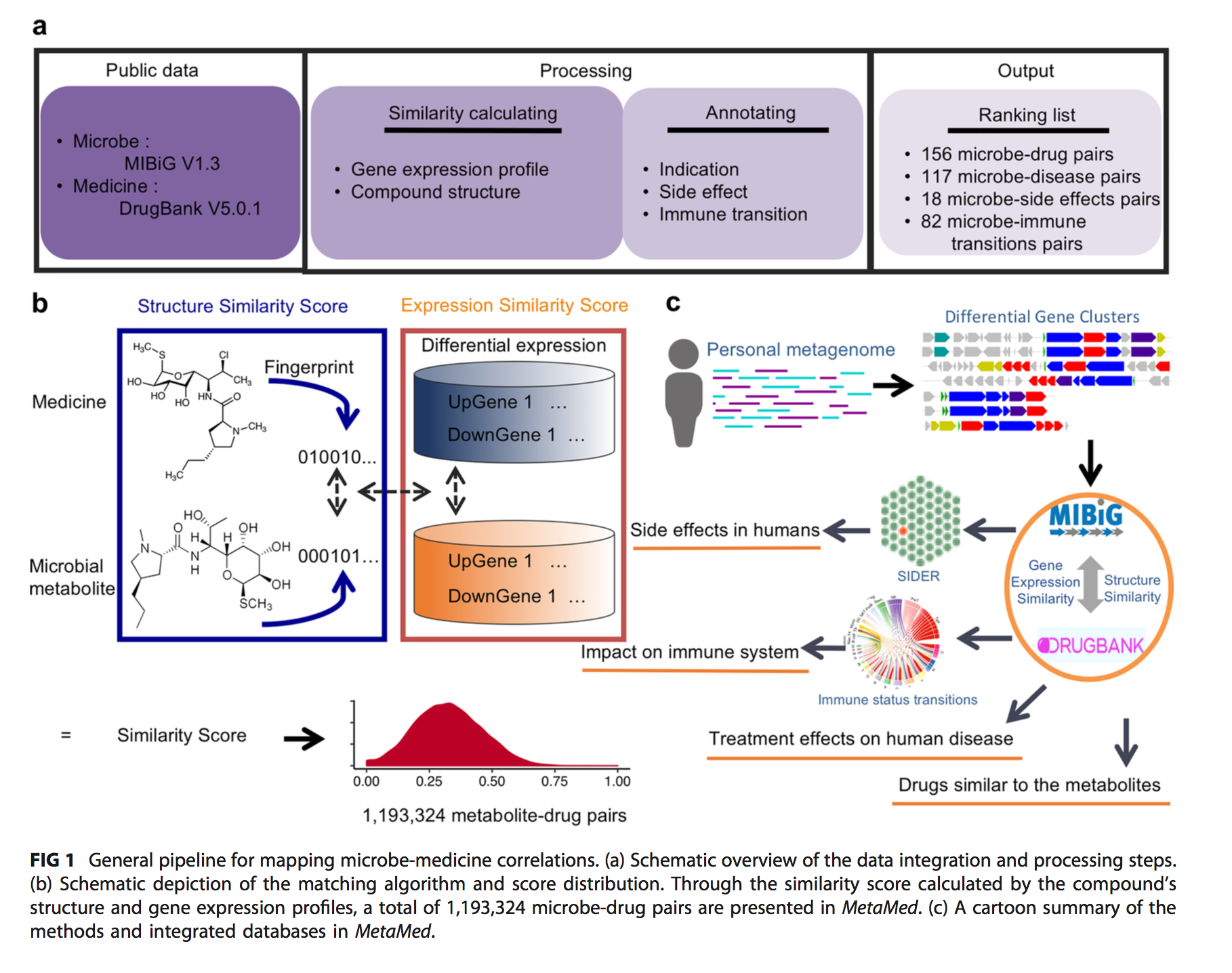
三、MetaMed提供的新发现和假设
3.1 放线菌和子囊菌是富含代谢产物的微生物的两种门类,对人类健康具有潜在的治疗作用。
通过应用我们开发的双峰聚类算法(定性双峰聚类算法QUBIC),得分截取为0.6(图2b)。 QUBIC有助于从两个角度同时清晰地解读微生物与药物之间关系的基本模式。我们发现具有相似门类的微生物聚集在一起,并与具有相似治疗作用的某些药物类别相关。显然,放线菌门菌已富集成为具有某些治疗适应症的微生物簇(图2b)。这些具有相似功能代谢物的成簇微生物为发现lead化合物提供了最重要的资源。此外,还提供了所有超过相似性阈值(相似性得分≥0.6,图2c)的预测连接的概述,以按治疗类别和门菌群将这些药物和微生物聚类。在门的水平上总共有九种微生物(图2c)。我们的分析表明,放线菌中的微生物产生的分子具有与抗感染,抗肿瘤和免疫调节药物相似的功能。这一发现与先前的研究(11)一致,即放线菌是新的次生代谢产物的有效生产者,这些次生代谢产物具有广泛的生物学活性,包括抗菌,抗真菌,抗癌,抗肿瘤和抗炎活性。此外,我们的分析为发现具有其他治疗功能的潜在细菌lead生产者提供了新的线索。例如,子囊菌中的微生物及其富集的代谢产物是潜在的来源,其功能类似于心血管系统,抗感染,抗肿瘤和免疫调节药物。我们基于与最多种类药物的相互作用来定义微生物枢纽,这些药物被认为对人类影响最大。另一方面,微生物岛的定义是基于与最少数量药物的相互作用(图2d;表S1)。

3.2 某些微生物产生次生代谢产物作为发现新药的潜在资源。
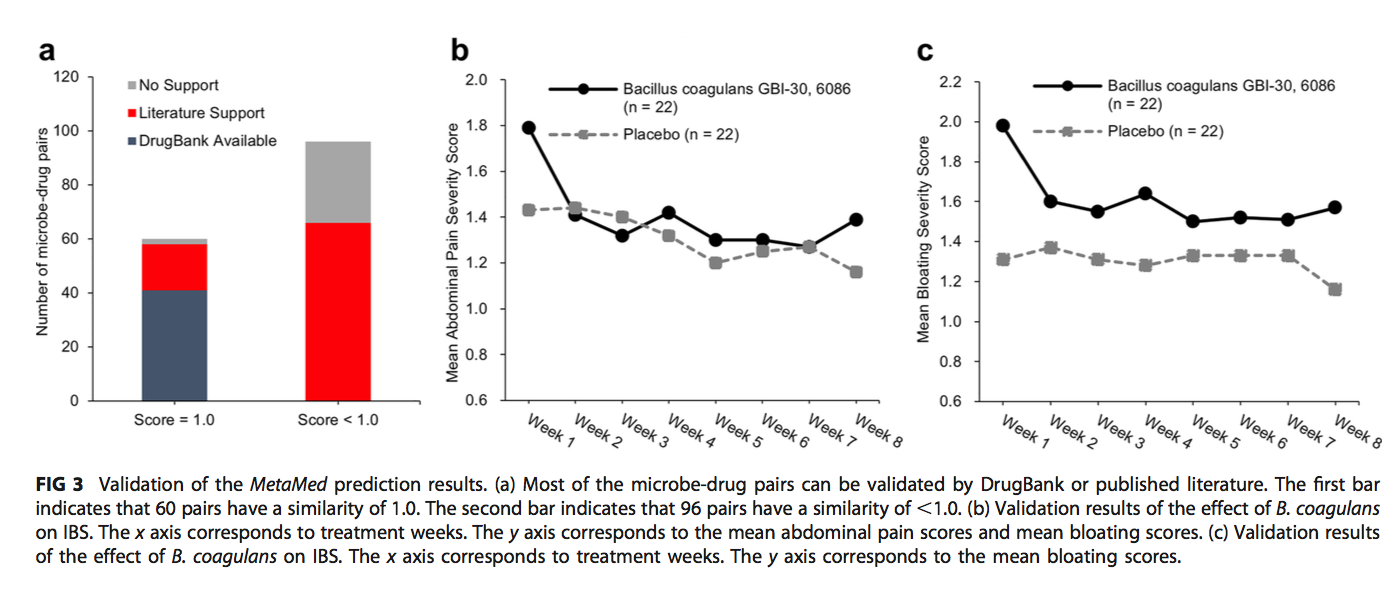
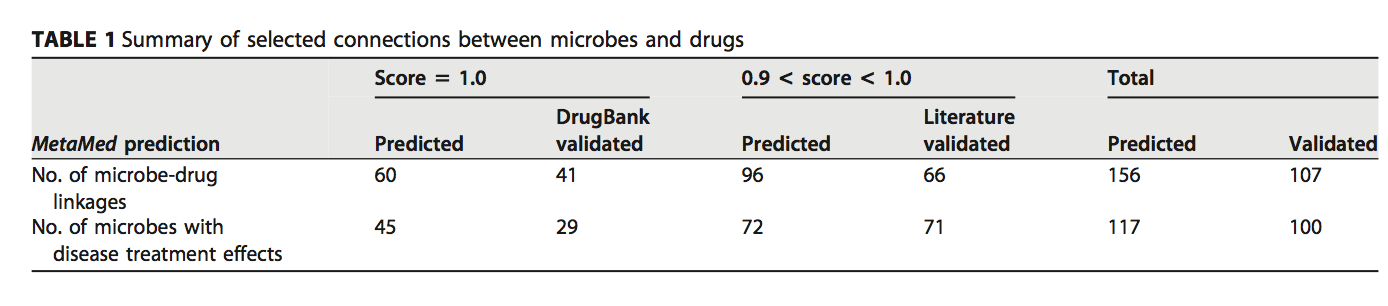
基于代谢物-药物对的相似性临界值0.9,MetaMed获得了156种有意义的微生物-药物联系(表1;表S2)。其中,60种微生物-药物对的相似性评分为1.0(38.5%,60/156),表明这些药物最初是从这些微生物中分离出来的,或者这些微生物可以产生与这些药物完全相同的次级代谢产物。在60种微生物中,已经有41种(68.3%,41/60)被标注为在DrugBank中生产相应的药物,而其余17种(28.3%,17/60)是由MetaMed标注为用于生产相应药物的新标识。对于另外96对微生物药物,它们的相似度得分低于1.0,而相似度仍保持在0.9以上。这些微生物中有66种(68.8%,66/96)具有与相应药物相似的治疗指征(图3a,表1和表S2)。我们的分析表明,这些微生物可以产生与相应药物相似的次级代谢产物。我们建议用户关注此类对,因为它们是发现新药的潜在来源。
3.3 MetaMed识别出具有疾病治疗效果的微生物
基于所识别的156种微生物-药物对,MetaMed通过利用药物注释信息直接预测117种具有疾病治疗效果的微生物(表1;表S3)(图1和文本S1)。其中,环境中存在113种微生物,它们无法在肠道中生存。但是,它们的代谢产物可以作为相应疾病的复合治疗剂摄入。对于某些无法摄取的微生物代谢物,需要进一步的研究以确定其摄取机理。在这113种微生物中,来自29种(25.7%,29/113)的代谢物已经在DrugBank中注明了其疾病治疗效果,因为这些代谢物与药物相同。另一类来自71个微生物的代谢产物(62.8%,71/113)也已通过基于文献的证据验证用于疾病治疗(表1;表S3)。总体而言,有证据支持100个微生物(88.5%,100/113)的次生代谢产物可用于治疗相应的疾病。 MetaMed预测,存在于人肠道中的另外两种内源性微生物,即凝结芽孢杆菌和大肠杆菌,可以分别以0.92和1.0的高分治疗肠易激综合症(IBS)。这些预测已通过两项临床研究得到验证(12、13)。一项研究用凝结芽孢杆菌(n = 22)或安慰剂(n = 22)治疗了44名IBS患者(12)。与对照组相比,IBS患者的IBS症状(如腹痛和腹胀)明显减轻(P all <0.01)(图3b和c)。另一项临床研究表明,大肠杆菌对IBS有治疗作用,尤其是对于肠道菌群改变的患者(13)。
3.4 MetaMed可以识别出具有副作用的微生物。
基于这156种已识别的微生物-药物对,MetaMed可以预测出18种具有副作用的微生物(图1,文本S1和表S4)。这些微生物通常存在于环境中,其次生代谢产物可能对人类产生副作用。在这18种微生物中,来自5种微生物的代谢物已经在SIDER中标出了副作用(8),因为这些代谢物与药物相同。根据已发表的证据,还证实了另一组源自两种微生物的与药物相似的代谢物具有潜在的副作用。 MetaMed预测,毒素链霉菌会产生与油性大便,腹泻和消化不良相同的副作用。这些预测在最近的一项研究中得到了成功的验证(14),其中毒链球菌的脂抑素的临床药物使用引起了不愉快的副作用,如油性大便,腹泻和消化不良。 MetaMed还预测淡紫色链霉菌会对听力产生副作用,例如听力障碍,高频耳聋,耳鸣和全耳聋。有趣的是,另一项研究(15)也报道了来自葡萄链球菌的卵霉素对内耳有副作用,必须注意预防耳毒性。这些细菌副作用的潜在机制是这两种微生物的次生代谢产物与具有相似副作用的药物相似。
3.5 MetaMed预测微生物会对免疫转换产生影响
MetaMed还利用最近发表的有关药物-免疫转变的信息,预测了82种微生物会对人类健康的免疫转变产生影响(表S5)(图9和图文S1)。其中,环境中存在81种微生物,它们无法在人体肠道中生存。但是,这81个微生物的代谢产物可以被视为对相应的免疫转变具有潜在影响的化合物。已经注释了来自35个微生物(43.2%,35/81)的代谢物,它们对免疫转换的影响(9),因为这些代谢物与药物相同。对于存在于人类肠道中的MetaMed鉴定的唯一内源性微生物,即凝结芽孢杆菌,MetaMed预测该微生物可以增加脾脏中的CD4 + T细胞。有趣的是,最近的一项临床研究(16)表明,给予凝结芽孢杆菌显着(P <0.001)会增加脾脏中CD4 + Foxp3 + T细胞的数量。
3.6 将内源性微生物改变信息与药物治疗联系起来,可作为药物组合预测的潜在策略
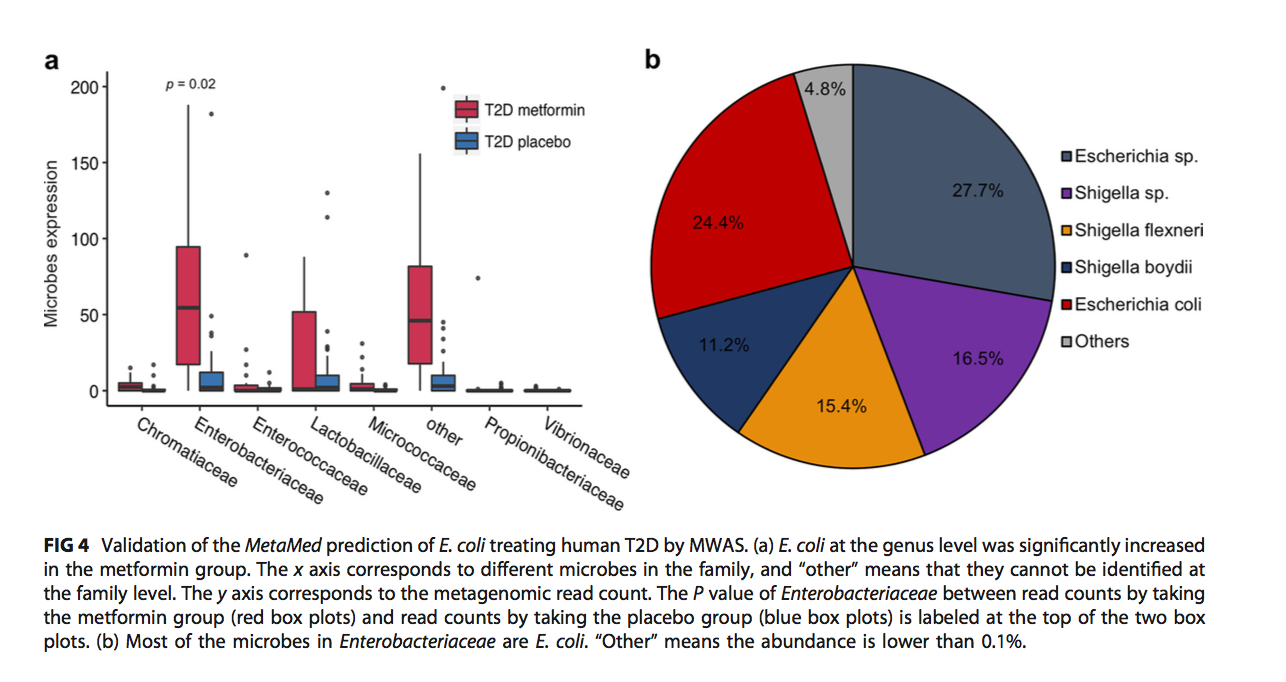
内源性微生物的变化也会影响疾病的治疗,我们进一步指出,将内源性微生物的变化信息与药物治疗联系起来,是预测用于治疗疾病的合适组合药物的一种潜在方法。我们通过以下两个案例证明了这一假设。 (i)最近,两项研究报道了肠道微生物参与调节抗CTLA-4和抗PDL1癌症治疗的疗效(17,18)。脆弱拟杆菌的生长与CTLA-4阻滞治疗伊立木单抗的黑色素瘤患者的疗效相关(17)。尽管这是一项相关性研究,但可以合理地推测脆弱的芽孢杆菌的代谢产物有助于改善依匹单抗的CTLA-4阻断治疗效果。在我们的研究中,MetaMed发现,氨基葡萄糖1-磷酸酯(得分= 0.73,在所有脆弱脆弱芽孢杆菌代谢产物中排名第一)和stibogluconate钠(得分= 00.70,在所有脆弱脆弱芽孢杆菌代谢产物中排名第二)与脆弱脆弱芽孢杆菌的相似度很高。代谢产物。有趣的是,一些研究报告说,stibogluconate钠可用于增强ipilimumab对CTLA-4的阻断作用(19)。 1-磷酸氨基葡萄糖的潜在免疫治疗效果有待进一步验证,尽管其潜在的免疫抑制作用和抗癌活性在其他地方已有报道(20、21)。 (ii)我们还从全基因组关联研究(MWAS)数据中分析了人类疾病中差异表达的微生物(22,23)。肠杆菌科是唯一两个在两个大小相似的亚组中表现出高度表达的细菌家族,这些亚组患有2型糖尿病(T2D),接受(n = 20)或未接受(n = 33)二甲双胍治疗的妇女(图4a),大肠杆菌是这种情况下最丰富的属(图4b)。在MetaMed中,大肠杆菌被确认与T2D治疗效果相关,这与MWAS结果一致。 MetaMed还确定了利那洛肽是与大肠杆菌代谢产物最相似的药物(评分= 1.00)。我们推测该药物可能是治疗T2D的潜在药物组合候选药物,尽管迄今为止尚未发表任何报道。我们研究了第二种最相似的药物,即利西拉来,它与大肠杆菌代谢产物相似,相似度为0.68。出人意料的是,据报道该药物可降低T2D患者的血糖水平(24),并与二甲双胍一起使用以改善T2D的治疗(25)。总之,尽管证据有限,但该证据表明,将临床治疗引起的内源性微生物变化与MetaMed的药物注释相关联的信息是预测组合药物作用的有前途的方法。
四、结论
应该注意的是,MetaMed利用来自MIBiG的微生物BGC产生的代谢物作为数据源,这可能导致排除某些没有BGC信息的微生物产生的已知药物以及相应微生物代谢物的实验注释。对于据报道由DrugBank中的微生物生产的所有93种药物,MetaMed在其中发现了41种。尽管MIBiG对BGC的注释有限,但MIBiG数据库提供了关于微生物BGC在特定条件下如何产生某些代谢物的综合实验注释,这是我们在此进行的主要研究。微生物可能具有产生药物的基因组能力,但仅在某些治疗或实验室条件下才会发生。由于MIBiG数据库提供了从公开文献中精选出来的某些微生物产生的代谢物的实验室条件的全面注释,因此选择该数据库作为我们的数据源。其他数据库(26,27)收集了更多的微生物,但是缺少此类注释。因此,我们认为,基于从MIBiG数据库收集的微生物代谢物的实验验证注释建立的MetaMed,可以为整个社区提供宝贵和可靠的资源。
为了促进对带有药物注释的微生物群功能的系统映射的全面探索,我们构建了网络系统MetaMed V1.1( http://metamed.rwebox.com/index ),以允许用户浏览各种微生物功能和药物注释链接信息,以及直接从个性化宏基因组学测序数据(Metapipe, https://github.com/adamtongji/metapipe )计算此类链接。通过分析这种联系可以得到新的发现和假设,而未来的方向将通过实验研究探索它们之间的因果关系(28)。总之,MetaMed首次尝试将微生物群功能与药物治疗联系起来,为深入研究微生物治疗对人类健康的影响提供了新颖的观点和假设。我们研究的目的是提出一种新的计算策略,以破译微生物对人类健康的影响,并且我们仅利用已知的文献证据验证了所确定的关系。我们鼓励将来对这些推导的假设或预测结果进行实验验证和研究。这些知识无疑将促进基于微生物群的营养疗法的成功应用,从而使微生物群相互作用药物在人类中的功效最优化,并有助于发现具有巨大药物应用潜力的微生物群代谢产物。
六、数据可用性
有关微生物与药物之间的相关性,微生物信息和药物注释的数据,请访问 http://metamed.rwebox.com/index 。 直接从个性化宏基因组学测序数据直接计算微生物与药物之间这种联系的代码可在 https://github.com/adamtongji/metapipe 获得。
参考资料
- mSystems. 2019 Sep-Oct; 4(5): e00413-19. Published online 2019 Oct 8. doi: 10.1128/mSystems.00413-19. MetaMed: Linking Microbiota Functions with Medicine Therapeutics 链接:https://msystems.asm.org/content/4/5/e00413-19
