【3.4.4】具有复杂域间几何的蛋白质域(domain)定义的数据库
蛋白质结构域是理解进化和蛋白质折叠所必需的,并且基于功能和序列的结构域变化广泛。尽管存在各种结构域数据库,但定义某些蛋白质的结构域并非易事,其域边界的定义不可用。在这里,我们提出了一个新的数据库,其中包含基于SCOP“多域蛋白”(multi-domain proteins)类( http://prodata.swmed.edu/multidom/ )的代表性蛋白质组,手动定义的结构域。
- 我们认为我们的domain是移动进化单位,可能在蛋白质进化过程中重新排列。
- 另外,它们可以被视为结构紧凑并且可能独立地折叠单元。我们还发现,将域表示为进化和折叠单元并不总是导致唯一的域定义。
但是,与现有数据库不同,我们在仔细检查结构相似性,功能位点和自动域定义方法后,保留并优化这些“alternate”域定义。我们为众所周知的数据库(如SCOP和CATH)中不会尝试分裂的蛋白质提供域定义,包括实际残基边界。我们的alternate域定义适用于自动化方法的序列和结构搜索。此外,该数据库可用于训练和测试域划分算法。由于我们的域代表结构紧凑的进化单元,因此该数据库可用于研究域属性和进化。
一、前言
虽然蛋白质结构域通常代表蛋白质功能的单位,但它们也可以被视为结构紧凑的半独立构建模块。因此,从历史上看,各种标准已被用于定义domain,即功能,顺序,进化,结构和折叠考虑因素。基于这些不同考虑因素的域定义并不总是彼此一致。例如,结构紧凑的单元可能不对应于已知生物功能的单元,因为功能位点通常容纳在结构域之间。在本文中,我们提出了一个结构定义的蛋白质域的数据库,并充分考虑了进化机制。我们选择的结构包括一些已知的结构最复杂的蛋白质,并在SCOP的“多结构域蛋白”类中分类。可靠的人工数据库如SCOP和CATH仅为某些结构中的域提供了残基边界。
结构域的特征在于存在分离的疏水核心。此外,域内残基接触比域之间更广泛。 残基接触中的这种差异可以指示折叠过程中的成核区域。 因此,结构域也可以称为独立折叠单元。 或者,在蛋白质进化过程中,可能发生通过插入或缺失的一级序列的分子重排( modular rearrangements )。 这些分子模块也可以作为结构域,并且可以基于与不同蛋白质中的其他结构域共存来定义。 这种分子性的逻辑扩展是维持域的序列连续性。 我们的工作利用这些观点来定义域。
可靠的参考域集的一个主要好处是在相似性搜索中,其中参考域可以用作查询。其次,它们也可以是自动域定义方法开发的训练集。这些参考域通常从SCOP [5]或CATH [6]等现有数据库中获得。 SCOP提供了一个手动矫正的蛋白质分类数据库,该数据库主要由同源性定义。例如,如果多肽链的一段存在于几种蛋白质中,但是与不同的非同源区段连接,则认为它是一个结构域。类似地,CATH基于架构(整体形状),拓扑(折叠)和同源性提供分层域分类,但是利用自动和手动过程的组合。许多自动化方法也可用于定义域。更可靠的是使用相似性检测,尤其是使用查询域的主要和三级结构搜索。但是,使用这些相似性来定位相关结构仅适用于较大的域或更高的身份,并且可用的可靠查询域定义。在本文中,我们将域定义与从自动化方法Domak 和PDP中获得的域定义进行比较。两种方法都使用inter residue接触来定位紧凑的结构区域。此外,Domak使用二级结构包装和来自一组参考域的统计参数,如文献中所述,用于域划分。或者,PDP通过最小化空间上接近的残基之间的链断裂来分解结构域。然而,尽管存在大量现有数据库和自动化方法,但仅在最简单的蛋白质拓扑结构中才观察到它们之间的域定义的一致性。
作为这项工作的结果,我们开发了一个手动域定义数据库,其中指定的残基范围主要基于结构相似性,紧凑性和序列连续性的组合概念。 这些域名定义可在 http://prodata.swmed.edu/multidom/ 在线获取。 我们的域定义不仅有助于相似性搜索和自动化方法的开发,还可能有助于研究域属性和进化。
二、结果
我们提供了一个新的手动定义域数据库,每个域的残基边界,一组拓扑复杂蛋白( http://prodata.swmed.edu/multidom/ )。 在仔细研究相关文献和检查现有自动化方法定义的域之后,我们的域定义基于结构,功能,序列和进化因素。 我们提供可下载的PyMol 脚本,以便轻松查看每个链的域定义,以及每个域的域序列和3D坐标。 结构域定义用于SCOP(版本1.73)中来自“多域蛋白”类的PDB 链的代表性组(40%序列同一性,157个总链)。 数据库中的PDB链分为53组同源蛋白质。 这些组对应于"多域蛋白"类中的SCOP"折叠"。 将更大和更多样化的组(例如聚合酶)分成亚组。
2.1 替代域定义
我们数据库中的域代表紧凑的进化模块,它们可以融合到其他域的主序列中或插入其中。我们认为域的融合或插入是由单个进化事件引起的。对于某些蛋白质,模块化域的这种进化考虑可以支持替代域定义(替代进化模块)。基于末端延伸的结构重叠,可以将同一对结构域可视化为末端融合(序列连续)或一个在另一个内插入(序列不连续)。对于具有大量结构域的蛋白质,对于仅少数或甚至仅一对组成结构域,这种替代定义是可能的。此外,域模块化概念禁止某些结构紧凑的区域由单个进化事件引起。通常,这些包括结构紧凑的插入其他域的组。我们不是从数据库中省略这些区域,而是将它们定义为另一个备用域(alternate domain)定义(复合域,composite domains)。这些备用域定义解释如下。
替代进化模块(Alternate evolutionary modules)。在多结构域蛋白质结构中常见的情况是在与相邻结构域相互作用的结构域的N-或C-末端存在相对短的非球状延伸。已知这种末端延伸稳定相关结构域(图1a,来自祖先结构域2的蓝色N-末端α-螺旋延伸与融合的粉红色祖先结构域1相互作用)。这种情况导致每个域的连续序列(图1a蓝色段,后面是粉红色段)。或者,扩展可能来自祖先结构域末端附近的域插入事件(图1b蓝色插入域分裂粉红色祖先结构域),在祖先结构域中产生序列不连续性(图1b蓝色区段分裂粉红色区段)。因为这些场景中的每一个在进化术语中同样是可能的,所以终端扩展是单独定义的,并且提供了替代域定义:“按序列(By sequence)“定义包括作为序列连续域的一部分的扩展(图1a),而"通过结构(‘by structure)“定义包括扩展作为结构相互作用域的一部分(图1b)。
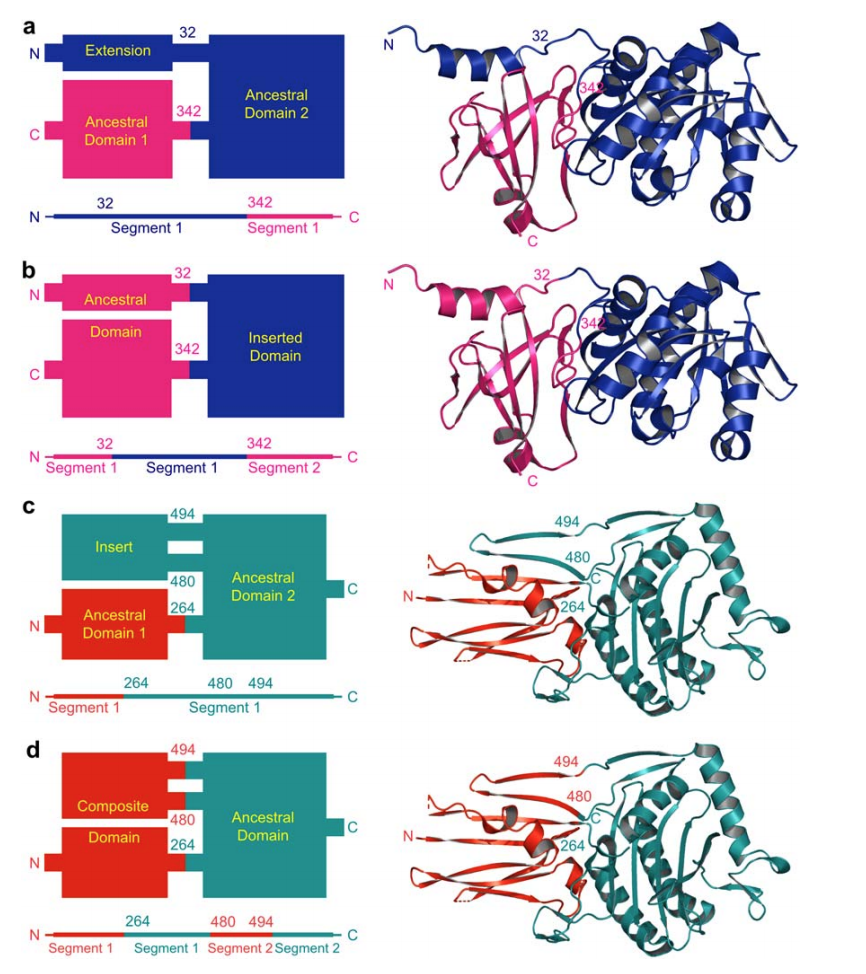
图1. 域定义类别。 表示域定义类别策略的框图域结构示意图显示在左侧,右侧显示相应的结构(a和b为1amu; c和d为1qme)。 表示多肽链中结构域位置的示意性序列视图显示在每个方框图下方。 残基编号在连接域的连接子上标记,N和C标记末端。 为清楚起见,仅显示了蛋白质结构的一部分和相应的示意图。 虚线表示从结构中省略的域。 从一个域突出但与另一个域交互的终端扩展被定义(a) 通过序列接近(‘‘通过序列’')或(b)通过结构接近(‘‘通过结构’')。 与相邻域相互作用的突出域插入通过(c)序列接近或(d)结构邻近来定义导致复合域。
复合域(Composite domains)。除了终端扩展之外,一些域还包括从域内突出的相对较短的非球状扩展(非终端扩展),但与另一个域交互。例如,从青霉素结合蛋白26中的profillin样α/β/α夹心结构域的中间延伸的β-发夹延伸N末端结构域的β-片(图1c和d)。该β-发夹可能作为对profillin结构域的插入而起作用,其稳定与N末端结构域的预先存在的相互作用(图1c中的蓝色结构域插入到祖先结构域2与红色祖先结构域1相互作用)。 保留具有突出域的插入保持两个域的序列连续性(图1c红色片段随后是青色片段)并遵守域模块化的进化假设。或者,插入可以在结构上定义为属于与其相互作用的N-末端结构域。这样的分配导致“复合”域,其只能通过多个插入事件来解释并导致序列不连续(图1d红色插入到teal域中完成红色复合域)。此外,还观察到一些相邻的非球状结构域形成具有共用疏水核心的紧密球状单元。我们的数据库还为这些域群集提供了复合域定义。因此,多结构域蛋白质数据集包括由插入和结构域的各种组合组成的复合结构域(插入+插入,插入+结构域或结构域+结构域)
总之,我们的数据库提供了四类定义:“按结构”,“按顺序”和复合域以及扩展。
- “由结构”(by structure)定义更适用于结构相似性搜索,因为与空间最接近的域相关联的终端扩展在寻找远程同源物中可能是重要的。
- ‘序列’(By-sequence)定义将序列连续多肽片段的末端延伸(类似于SCOP)归因于序列搜索策略更有用; 因为这样的区域可能含有保守序列基序。
- “扩展”(extensions)类别指定扩展区域(终端扩展,插入和连接),区分“按结构”和“按顺序”定义。
- 复合域(composite domains)不代表进化单元,纯粹是几何的,但可能在收敛进化的研究中有用。
2.2 与其他手动和自动域定义方法进行比较
我们数据库中手动定义的域有三个替代类别(613’按结构,612按顺序,58’复合’)。此外,数据库还单独定义了83个插入,链接器和终端扩展,它们也是域定义的一部分。 CATH 是使用手动和自动程序创建的数据库,以及两种自动化方法,即PDP和Domak,分别为我们数据库中的结构定义了273,443和297个域。但是,当该项目完成时,CATH尚未分配58个PDB链(157个)中的域。我们为多域蛋白质类中的每个链定义了至少两个结构域(图2a中的结构),每个链定义的结构域数量逐渐从2减少到6。手动定义也将13个链分成7个或更多个结构域,并且主要是参与复制和转录的蛋白质,特别是聚合酶。尽管SCOP 并不总是为多域类比较提供域范围,但数据库很少在前四个结构类中为每个链定义三个以上的域(图2a SCOP)。 CATH域号最接近我们的手动方法(图2a CATH),具有2-6个域的链的数量非常相似。然而,CATH确实将14个链定义为单个域,并且很少链具有7个或更多个域。自动域定义方法,尤其是DOMAK(图2a DOMAK),倾向于分配更少的域。然而,PDP执行接近手动定义,每个链的域数较少(图2a PDP)。与我们的定义不同,PDP确实将链定义为单个域,而具有7个或更多个域的链的数量非常相似。
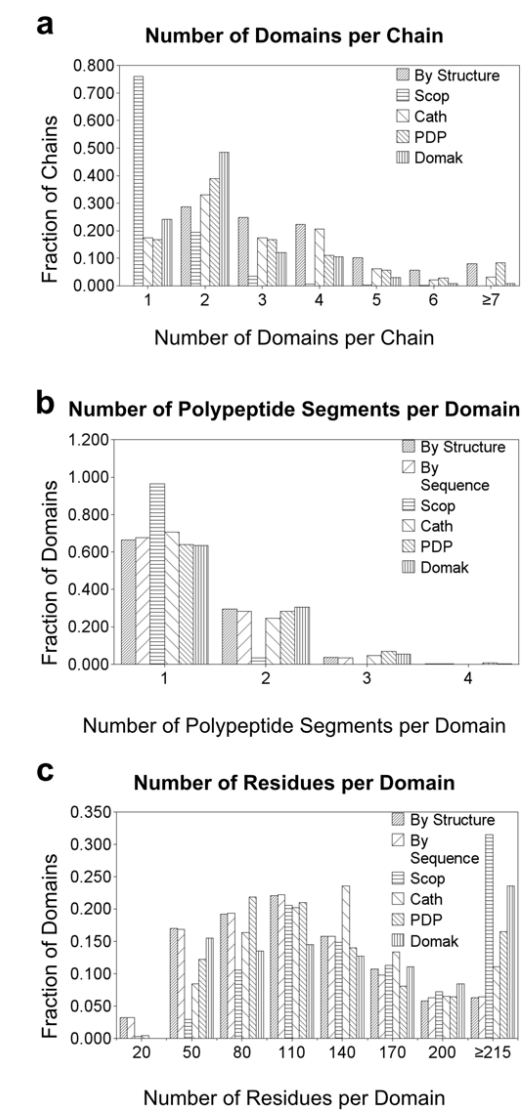
图2.域定义比较。 “按结构”和“按顺序”将类别定义(见图1a,b)与CATH和自动化方法“PDP”和“DOMAK”进行比较,用于我们的结构数据集(参见方法)。 SCOP的数据是从SCOP类1到4中的PDB链生成的。垂直轴上的数据被标准化为相应数据集中的PDB链或域的总数。 (a)每种方法按链定义的域数。 “按顺序”的数据与“按结构”相同,未显示。 (b)包含每个结构域的多肽片段的数量。 (c)代表确定域的残基长度的直方图。 为清楚起见,仅显示长达215个残基的结构域
尽管与自动化方法相比,手动定义单个链的更多结构域,但手动定义产生每个结构域的多肽片段数量略低(图2b)。手工结构域序列连续性的增加是由于对分裂施加了进化假设:由多个插入片段组成的紧凑区域不能代表单个进化事件,而是在特殊类别的“复合”域中处理。手动“按顺序”和“按结构”类别定义之间每个结构域存在的多肽片段数量的差异表明含有末端延伸的结构。因此,终端扩展被分配序列连续(按序列)的域具有比通过结构邻近(通过结构)分配终端扩展的域略低的段数。与手动和自动域定义相比,SCOP很少在前四个结构类的分类中引入序列不连续性(图2b SCOP)。 CATH定义的每个域的段数比我们的定义略高。
对于结构域大小的分析,我们的手动定义与PDP,SCOP和CATH相当,定义的结构域的百分比在95和215个残基之间,DOMAK向更长的域移动(图2c)。 然而,在此范围内,CATH定义了更多的长度范围为125-185个残基的结构域。 尽管所有方法在每个结构域显示约95个残基的峰,但CATH在140附近显示峰,然后结构域大小突然下降。 与其他相比,我们的数据库包括大量具有少于50个残基的结构域,并且很少定义超过300个残基的结构域。 我们的手动方法确定了自动化方法未检测到的非常短的结构域(例如通过检查半胱氨酸和组氨酸而发现的锌指)。 此外,我们的定义倾向于提供更小的紧凑域。
在我们的视觉评估期间,我们发现自动化方法无法为数据集中的大多数链提供一致的域定义。自动化方法仅在非常简单的情况下达成共识,其中结构紧凑的域显示很少的域间交互。几乎在所有情况下,即使域号及其一般位置正确,这些程序的域边界也需要进一步细化。从CATH获得的域定义虽然不完整,但更可靠。然而,我们确实注意到类似蛋白质之间的结构域定义存在杂散不一致。域定义的这些问题和局限性已被其他人充分记录[18]。但是,我们发现即使这些有限的定义也很有用,例如建议使用潜在的域核心。文献有助于我们对几个领域定义的功能考虑,例如:在1ecr [21]中,分析了非紧凑功能区域和域界面处的残基,以便将它们分配到正确的域。
2.3 进化考虑:DNA / RNA聚合酶中区域描绘的一个例子
对于具有大量可用结构的蛋白质家族,在定义结构域时将结构相似性与进化考虑相结合是特别有用的。 我们的模块化域可以通过保守域的结构相似性,各种相关域的插入位置和一般拓扑以及它们之间的相互作用来进行正确识别和边界细化。 我们以DNA / RNA聚合酶为例说明了我们的一般思路和对结构域定义的困难。 聚合酶的常见结构包括通常称为“‘手掌’”,“拇指”和“手指”的三个功能域,其中普遍存在的手掌结构域为酶提供催化活性。 我们基于结构对齐的容易性和三个共同结构域的相似性将聚合酶细分为四组(图3,I-IV)。
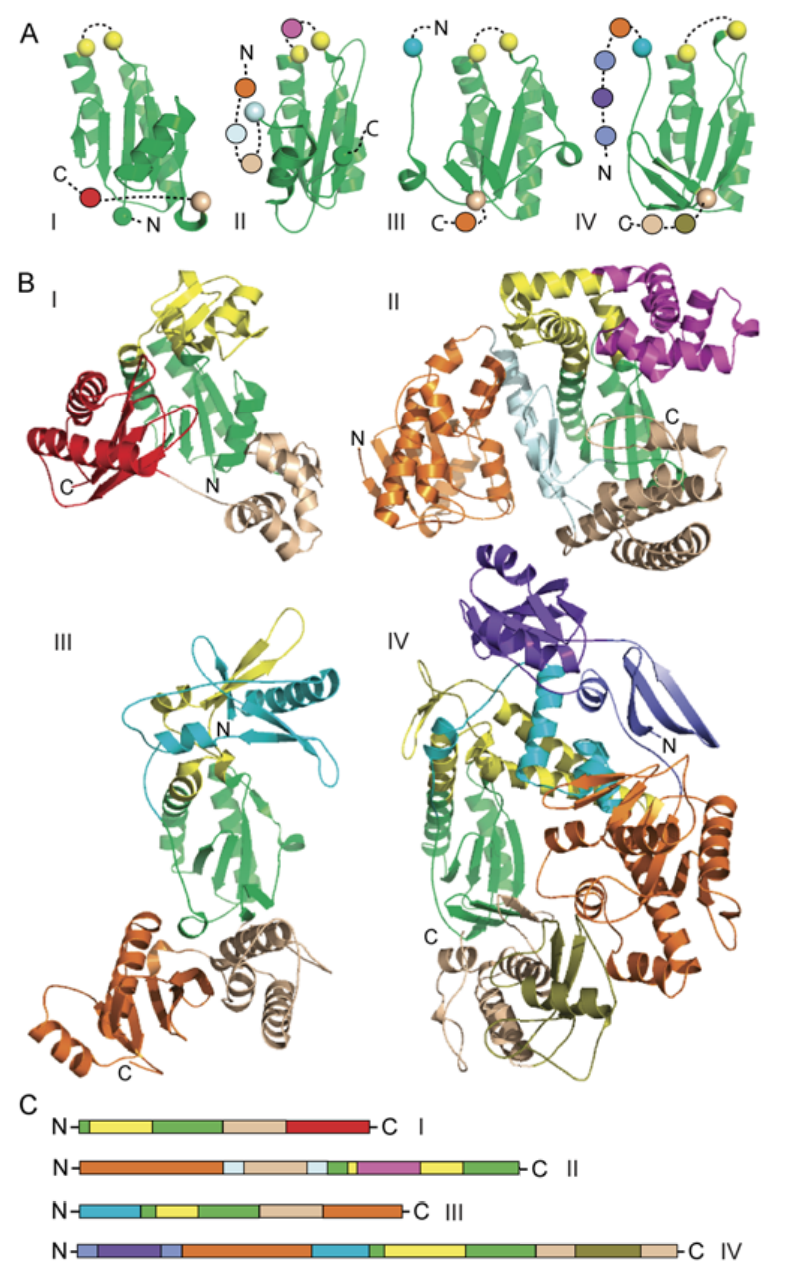
图3. 聚合酶中的模块化结构域。显示不同复杂性和连接性的组织的不同聚合酶结构被分成四个标记的亚组:i)Y家族DNA聚合酶,ii)Klenow DNA聚合酶/T7噬菌体聚合酶,iii)逆转录酶/ RNA依赖性RNA聚合酶,和iv)DNA聚合酶I. a)所有聚合酶结构都具有同源催化手掌(Palm)结构域(绿色卡通模型)。来自每个聚合酶子集的代表的手掌结构域以相似的方向从左到右描绘(i 1xx4; ii 1u4b,iii 1vrt和iv 1tgo)。彩色球体标记手掌域边界:插入的手指域(黄色),N-末端域(青色和浅蓝色)或C-末端域(小麦)。另外的域表示为通过虚线从N-连接到C-末端的有色球体。 b)Sulfolobus solfataricus DNA聚合酶IV(i)嗜热脂肪芽孢杆菌DNA聚合酶I,(ii),HIV-1逆转录酶(iii)和Thermococcus gorgonarius B型DNA聚合酶(iv)的卡通结构模型。如在A中着色.c)定义的域的序列连续性,表示为从N-到C末端的块
在来自所有组的聚合酶结构中,催化手掌(palm)结构域掺入了铁氧化还原蛋白样折叠(图3a,绿色),其由许多其他结构域可变地修饰。铁氧化还原蛋白样折叠本身包含针对保守的β-片包装的各种额外的二级结构元件(图3a):组I中的手掌结构域包括C-末端α/β延伸,组II包括N-末端螺旋延伸和III组和IV组包括C末端β-发夹。我们的域在大小和球状上显示出相当大的变化(图3b:青色和橙色域)。另外,这些不同的域可以在单个结构内进行各种组合。观察到许多域插入;因此,尽管所有域都是紧凑的和模块化的,但并非所有域都是序列连续的(图3c)。 例如,绿色手掌域中的序列不连续性是由黄指结构域的插入引起的。我们的进化考虑因素可以解释这些域插入中的每一个都是由不同的单个进化事件引起的。此外,还观察到'‘嵌套’插入,并且可能由于域插入的级联而发生(例如,插入黄色结构域的结构紧凑的洋红色结构域,其中黄色结构域本身插入图3II中的绿色结构域) 。
虽然手掌结构域是最保守的,因此代表了聚合酶结构的进化核心单元,但拇指和指状结构域表现出显着的拓扑变异。相对于手掌(图3b,小麦),拇指结构域的结构位置是保守的,尽管它的顺序位置不同(图3c,小麦)。因此,Thumb域定义基于相对于手掌的位置。第1组,第III组和第IV组拇指融合到手掌C-末端,而第II组拇指在手掌的N-末端作为嵌套插入物发现(图3,浅蓝色)。另外,尽管保留了主要的α-螺旋二级结构组合物,但拇指结构域在组之间显示出低结构相似性并且包括α-螺旋的不同大小和拓扑结构。与拇指域相反,基于相对于手掌域的顺序位置来定义手指。手指域的一个组成部分(图3,黄色)总是插入相同位置的手掌域。然而,手指域的二级结构组成,拓扑以及与相邻域的交互是变化的。
在聚合酶中也观察到复合结构域。 与N末端结构域(图3,青色)一起,来自III组和IV组的插入形成复合球状结构,其具有广泛的域间相互作用和共享的疏水核心。 复合结构域是序列不连续的(图3C,青色,绿色和黄色),并且其形成不能通过单个插入事件进化解释。 因此,我们不认为它是一个进化模块。 因此,复合域代表一种特殊情况,其中一级序列排列和进化考虑指示两个独立的域,而结构建议组合域定义。
除了手掌,拇指和手指之外,聚合酶结构还包括各种其他结构域。 一个这样的结构域类似于核糖核酸酶H样(RNAseH样)折叠拓扑结构(图3b,橙色)。 发现该RNAseH样结构域位于聚合酶中的一级序列(图3c,橙色)中。 组II和组IV中的结构包括常见聚合酶组分N末端的RNAseH样结构域,而组III中结构域是C末端。 组I中的结构一起缺乏RNAseH样结构域。 这些变化说明了聚合酶之间的结构域的模块化重排,并提供了进一步的域边界线索。
四、讨论
虽然生物学家认为蛋白质由结构域组成,并且组成结构域的分析对于研究整个蛋白质是重要的,但是对于各个结构域本身的性质和定义存在广泛的分歧。 因此,必须解决域(什么是域?)的概念以及在给定蛋白质中描述它们的方法(如何找到域边界?),并且是自动分析各种蛋白质性质的先决条件。 另外,基于一些一般原理,出现了关于为蛋白质提供一致的结构域定义的可能性的问题。 这项工作基于一组拓扑复杂的蛋白质,代表了迈向这些重要目标的一步。
4.1 什么是域?
领域研究中最大的困难可能是缺乏对问题的统一答案:什么是域?有趣的是,可以提出几个领域的“概念”。其中,以下五个广泛的概念值得注意。
- 功能结构域的特征在于功能独立性,并且形成足以进行某种主要酶活性的单位。由于蛋白质结构域的功能表征通常是主要目标,因此蛋白质结构域通常被认为是功能结构域。
- 序列结构域是多肽链的区域,其可以通过序列相似性检测,并且可以与其他序列结构域组合发现。这些结构域广泛用于序列分析,其中不正确的边界意味着对保守区域的错误描绘。由于基于序列分布的搜索方法(如PSI-BLAST 和其他用于同源性推断的传递策略)依赖于保守区域,因此正确描绘域边界是必不可少的。
- 进化结构域是可以通过重组,转座,外显子改组和其他突变事件在蛋白质之间移动的模块。因此,它们在不同的域上下文中的出现,提供的同源性可以通过序列或结构相似性来检测,形成进化域定义的基础。
- 作为折叠单元的域在蛋白质研究的早期被定义[2]。该概念意味着分离的结构域能够独立折叠,或者至少具有可以引发蛋白质折叠的折叠核。
- 结构域通过结构紧密性,疏水核心的存在以及域内而不是域间更广泛的氨基酸相互作用来几何定义。这些域对结构相似性搜索至关重要。
对于许多蛋白质,例如那些看起来像“字符串上的珠子”的域彼此分开的域,所有5个概念都可能导致相同的域定义。然而,用于制定这五个领域概念的标准是完全不同的,并且这些概念始终如一地并非总是可能的。当域更密切地相互作用时,不同的概念不可避免地导致不同的定义。这项工作试图在蛋白质链的一些最拓扑复杂的例子中协调这些领域概念中的几个。
4.2 替代域定义 Alternate domain definitions
域可以被认为是可能在蛋白质之间混合的进化单元,并且是生物多样性研究的自然观点。 当来自不同蛋白质的结构域之间的序列相似性高时,即使结构域结构不同,这种进化考虑提供了对结构域识别的简单指导。 然而,当难以确定与其他蛋白质的序列或甚至结构相似性时,结构域定义变得特别困难。 这种困难在域边界特别明显,其中域定义倾向于仅在额外考虑结构紧凑性和残差之间的相互作用等几何特性时才有可能。
由于生物复杂性和进化推论的不确定性,单个域定义可能无法解决域的起源的真实过程。另外,由于当代自动序列和结构分析方法的限制,甚至可能不需要这样的单域定义。相反,我们将域定义视为3类: 其中两个(“按序列”和“按结构”)表示基于相同的进化考虑的域起源的不同合理机制。第三类“复合”域仅基于几何属性,而不是基于我们的进化考虑。我们还提供了另一类“扩展”,以列出致病多肽片段,这些片段区分“按序列”和“按结构”定义。尽管采用了这种特殊处理,但由于没有扩展,大多数域在“按顺序”和“按结构”定义上没有区别。 尽管如此,我们在数据库中单独列出它们以便于自动化方法的使用。因此,我们的工作设法从许多拓扑复杂蛋白质的进化角度提出了完整和一致的域结构图。
结构域的明显固定的相对排列对于许多蛋白质的功能是必不可少的。然而,通过它们之间的单个连接器实现这种固定定位可能是困难的。因此,一个域经常包括到达相邻域的终端扩展,通过附加交互提供稳定性。此扩展可以在结构上可视化,作为交互域的一部分。从进化的角度来看,包括作为交互域的一部分的扩展假定了域插入的场景。还可以设想替代方案,其中扩展可以从突出域演化,提供序列连续域定义。在大多数情况下,导致这种情况的真正进化机制仍然不清楚:即域插入与其中一个域的扩展的添加。这种不确定性的一致解决导致我们的替代域定义;例如"按结构"和"按顺序”。术语“按顺序”和“按结构”指的是终端扩展到域的归属,而不是域识别的方法。首先,在定义'‘by structure'‘中,扩展按结构分配给交互域。这导致“祖先”域的不连续序列(图1b),并暗示一个域被插入另一个域。即使这种进化方案不准确,结构定义仍然有意义,因为扩展经常占据结构中的位置,该结构在结构相似的单结构域蛋白中容纳相同的二级结构元件。因此,将扩展作为相互作用域的一部分处理对于基于结构的相似性搜索和远程同源推断可能是有用的。其次,在定义'‘by sequence'‘中,扩展被分配序列连续到它扩展的域。此定义导致域内的链断裂更少。因此,“按顺序”定义导致最小的紧凑和球状结构域比我们的“结构”定义。保持序列连续性和具有无间隙的结构域序列使得“按序列”的结构域定义适合于序列分析。许多SCOP 域定义,在多域蛋白质类之外,与我们的“序列”定义相匹配,因为SCOP倾向于最大化域的序列连续性。第三,我们在数据库中单独列出所有这些终端扩展,以便于识别和分析。
几种延伸和插入可以结合在一起并形成紧凑区域,具有明确限定的疏水核心。这些区域通常可以包含紧密相互作用的二级结构元素,甚至可以共享单个β片。根据我们的说法,这些区域不构成域,因为它们不是进化上的移动单元,而是仅仅是由于对其他域的扩展而产生的几何关联。这些紧凑区域在视觉上很容易识别,并且通常通过自动域定义方法定义为域。因此,我们建立了第四类,称为复合域,以适应这些区域。除了由扩展和插入形成之外,一些复合结构域还包括插入的非球形结构域。在所有情况下,复合域是序列不连续的,但形成紧凑的空间体,其不能源自单个进化事件,例如域插入。因此,这些领域纯粹是几何的,在我们的定义中,不具有进化意义。我们之所以对它们进行定义只是因为它们出现在稳定的构象中,似乎可以将类似的拓扑结构视为未来其他蛋白质中的进化域。
我们的备用域定义(alternate domain)从一些一般原则提供了对域源的不同观点。 由于域来源的真实证据要么无法实现,要么不完整,我们选择不将自己局限于单一的最佳域名定义。 此外,计算资源的最新发展表明,从不同的查询集合开始合并更准确的序列和结构搜索在不久的将来不会受到限制。
4.2 手动域定义
我们从域定义的角度选择了一组最具挑战性的蛋白质:整个SCOP ‘‘多域蛋白'‘类。 SCOP将这些蛋白质统一在一个类别中,因为它们在结构和功能上都包含彼此紧密相关的拓扑不同的结构域(例如全α,全β,α/β和α+β)。因此,这些蛋白质的“折叠”被定义为多结构域结构,即使可以将各个结构域归因于其他SCOP折叠。因此,SCOP不为这些蛋白质提供结构域边界,尽管它经常提到结构域类型及其可能的分类。 SCOP域定义是可靠的参考;然而,考虑到缺少残基边界,将多结构域蛋白质类中的结构视为单结构域是不正确的。我们的数据库为这些拓扑复杂蛋白的代表性集合提供了一组精心定义的域边界。我们的数据集中的结构代表数与SCOP层次结构的“蛋白质域”级别的条目数紧密匹配。因此,我们相信我们的数据库涵盖了SCOP多域蛋白质类别中编目的所有潜在域名安排。
独立的人类专家同样定义了大多数域[24]。 这一观察意味着具有生物学意义的域定义需要经验丰富的判断,并与自动化方法的结果之间明显的不一致形成对比。 我们在当前的工作中观察到了这种分歧。 在我们的域定义中,我们偏向于较小的域而不是将几个多肽片段合并成更大的域。 对于新颖和不寻常的域,我们强调较小尺寸的重复和具有确定的疏水核心的小(20-40个残基)几何形成。 然而,我们没有将诸如Rossmann折叠单元之类的众所周知的区域分成更小的部分,例如, 形成双重伤口折叠的两个重复。
在我们的手动域定义中,我们重复执行某些步骤。尽管并非所有这些步骤都是定义每个域所必需的,但是观察它们的频率足以引起特别提及。我们按它们被调用的近似顺序讨论它们。
1》 使用结构和序列方法检测结构相似的蛋白质。类似但不相同的蛋白质(同系物)显示出不同的结构域排列。这些同源物的比对表明至少一些结构域的结构域边界,使得剩余结构域的描绘更容易。有时观察到不同蛋白质(具有低相似性得分)含有相似的结构域,然后我们可以对齐并描绘。一些同源结构在不同的生长阶段显示出额外的结构域。为了保持一致,我们仅在疏水核心可辨别时才定义这些额外的结构域,否则将多肽片段作为环处理。 2. 找出给定蛋白质中可能的重复。结构重复可能通过重复形成,因此代表单独的进化结构域。 3. 尝试识别罗斯曼(Rossmann)折叠,铁氧化还原蛋白折叠,4-螺旋束,免疫球蛋白,SH3和转基因桶等规范域。这一步受到我们经验的限制。但是,频繁转介相关文献缓解了一些问题。解决这些限制的唯一方法是实际拥有一组参考域,这正是当前工作朝着解决方向迈出的一步。甚至可以在上面步骤1中的结构搜索期间使用这样的参考集。 4. 根据单独的β表的存在识别潜在的域。各个域通常包含完整的β sheets。在极少数情况下,域包含多个片,然而,序列相互缠绕(例如,beta sandwich)。 5. 基于α-螺旋的空间聚集识别潜在的域。通过步骤4和5识别域核心通常很容易。然而,域接口的位置有时不清楚。内部和域间残基接触之间的差异有时难以察觉。在这方面,残基的侧链取向有一些帮助。在极少数情况下,我们求助于结构相似性检测(步骤1和3)。 6. 通过注意序列连续性和域插入事件将域视为进化模块。我们的域定义的先决条件是所有序列不连续域只能由于其他域插入而起源。在此步骤中观察到大多数自动化域定义方法失败。 7. 将任何未分配的肽片段归属于已识别的域核心。短(20个残基)片段归因于现有域。较长的区段被定义为新的区域或基于存在感知的疏水核心而归因于现有区域。 8. 基于侧链取向和相互作用以及结构相似域的叠加来精确确定域边界。
4.3 域数据库的潜在应用
可以使用我们的域数据库的几个应用程
- 数据库可用于训练和测试自动域定义算法,因为它是唯一为拓扑,最具挑战性的蛋白质链提供域定义的数据库。
- 数据库可用于研究域交互,接口和拓扑。此外,域组合的变化允许数据库成为可能的域架构的参考。
- 我们的手动域定义标准,观察和陷阱可能有助于设计将模块化进化单元视为域的自动域定义算法。
- 我们的结构域的紧凑性表明可能的折叠单元或核,并且它们的分析可以增强对蛋白质折叠和结构预测技术的理解。
- 对我们数据库中编目的特定蛋白质感兴趣的生物学家可以推断出可以分离用于生化研究的功能和进化单位。
- 虽然我们的复合结构域是由几个不建议移动进化单位的序列片段组装而成的,但这样的组装结构域可能与其他蛋白质中的进化结构域相似。作为未来的工作,这些发现可能产生类似的结构域对,可用于分析蛋白质进化的收敛
五、材料和方法
5.1 数据集准备
结构数据集。 代表SCOP'‘多域蛋白'‘类(版本1.73)的157个PDB 链的数据集被用于这项工作。 每个结构代表都是从ASTRAL 提供的单个聚类集中选择的。 ASTRAL从SCOP中的PDB链开始提供这些聚类集合,序列同一性低于40%。 PDB文件中的修饰残基名称(由MODRES记录标识符指示)被转换为标准遗传密码的相应残基名称。 二级结构信息由PALSSE生成并合并到PDB文件中。
推定的域数据集(来自CATH和自动化方法)。获得CATH(版本3.0)定义的域边界以供参考和比较。 PDP 和Domak 的结构域分配也有助于快速识别潜在的域。 Domak运行了几个非默认参数。基于人工观察Domak为来自PDB的100个原始选择的结构定义的域来选择参数。这些参数中的四个用于增加残留物覆盖并且设置为相同的值(MIN_PEAK_BLO_C = MIN_PEAK_SS_ ONLY_BLO_C = MIN_PEAK_BLO_DC = MIN_PEAK_SS_ ONLY_BLO_DC = 80)。增加了域内联系的最小分数(“分割值”)以获得更多不同的域(MIN_PEAK_C = MIN_PEAK_DC = MIN_PEAK_MC = 15)。最后,二级结构接触所需的二级结构含量的最小分数(与残余物接触相反)将被专门使用增加(MIN_SS_PER = 0.8)。
5.2 检查域特征
视觉检查结构坐标对于识别对域定义重要的属性至关重要。 许多结构特征有助于定义域边界。 这些特征中最重要的是二级结构填充和拓扑,球状和疏水核心。 此外,评估结构相似性和进化模块的评估有助于定位保守域并定义域边界。 将得到的手动域定义与从可用数据库,自动化方法和已发表的文献中获得的定义进行比较,有助于完善我们对困难案例理解。
5.3 手动域定义过程
我们的方法可以大致分为两个步骤; 识别域的数量和一般位置的初始步骤(步骤1),以及(步骤2)域边界的后续细化。 由于域边界的细化(步骤2)还涉及对域的结构扩展的分配,因此该步骤影响了我们域的顺序排列和模块化表示。 由于我们对域的能够进行模块化重排的观点,这种顺序重排有时需要改变域号(步骤1)。 因此,我们通过上述两个主要步骤遵循域定义的迭代方法。
识别域的数量和一般位置。推定的域数据集(如上所述)有助于告知我们潜在的域。在大多数情况下,结构上不同的紧凑区域通过自动化方法容易地分裂成域。我们将这些推定的域视为域核心,并尝试重新定义这些核心产生的域。但是,自动方法有时会在域检测中显示出明显的错误。罗斯曼(Rossmann)和PIN域等大而容易识别的折叠有时会过分分裂。对于这些可识别的折叠,我们对域核心的考虑与自动化方法所建议的相比有所改变。在补体中,通过自动化方法合并较小的褶皱,例如铁氧化还原蛋白,RNaseH,4-螺旋束,免疫球蛋白,SH3和OB桶,并且多个倍数被指定为单个结构域。对于这些折叠,我们依赖于二级结构填充的视觉评估和疏水区域的感知存在,用于识别结构紧凑和球状域核心。通过金属离子和近端组氨酸和半胱氨酸残基的存在检测到小锌结合结构域。关于拓扑结构仔细检查相邻折叠以获得重复的证据。结构中的重复域通过手动观察和序列和结构比对来定义
细化领域边界。对领域边界进行了细化,以按顺序和结构分配模块化域。考虑了这些模块在蛋白质进化过程中重排的可能性,并在我们的定义中表达。通过比较我们的数据集中相同SCOP [5]超家族中的所有相似域来辅助边界细化,我们认为这与进化相关。通过DaliLite 产生的结构比对评估了潜在结构域的相似性,其中我们考虑了比对的残基来定义结构域的保守区域。因此,域边界被设置为在结构上相似的和可能与进化相关的域之间是一致的。只要没有相似的结构域,通过观察多肽骨架和残基侧链几何结构确定结构域边界。我们假设模块结构域在蛋白质进化过程中重排,在确定结构域边界方面起了作用。在结构域插入位点,研究多肽骨架的几何结构以将空间上近端残基分配到祖先结构域。此外,我们假设在域内而不是域间残基之间存在更大的侧链相互作用。
序列连续性和备用域(alternate domain)定义。我们对模块化域定义的遵守确保了各个域的序列连续性;除非还存在其他插入的域(本节后面描述的复合域除外)。因此,对于插入结构域A的n个结构域,结构域A被定义为由n + 1个多肽片段组成。然而,对于一些相邻域对,观察到一个域包含与相邻域相互作用的末端结构延伸。在这些扩展被识别为二级结构元素(螺旋或b片)的任何地方都提供了替代定义。我们通过结构'‘确定终端扩展’',确保域核心扩展的结构紧凑性。这种考虑导致了一个域被插入另一个域的定义。此外,我们通过序列'‘确定终端范围'‘确保每个域的序列连续性。该替代方案导致一种定义,其中结构域通过序列终止融合,两者中没有不连续性。复合域构成了我们域定义的第三类,仅基于结构紧凑性。在极少数情况下,二级结构延伸和向域的插入,以及非球状结构域形成结构紧凑的区域,其中可以感知疏水核心。我们将这些区域定义为复合域,尽管它们是序列不连续但不包含插入的域。
参考资料
- April 2009,PLoS ONE.《A Database of Domain Definitions for Proteins with Complex Interdomain Geometry》 http://prodata.swmed.edu/Lab/MultiDom_PLOS109.pdf
