【3.2.2】检测蛋白质构象是否合理--Ramachandran plot(拉氏图)
Ramachandran plot(拉氏图)是由G. N. Ramachandran等人[1]于1963年开发的,用来描述蛋白质结构中氨基酸残基二面角ψ和φ是否在合理区域的一种可视化方法。同时也可以反映出该蛋白质的构象是否合理。
在介绍Ramachandran plot前有必要简单说明一下二面角。
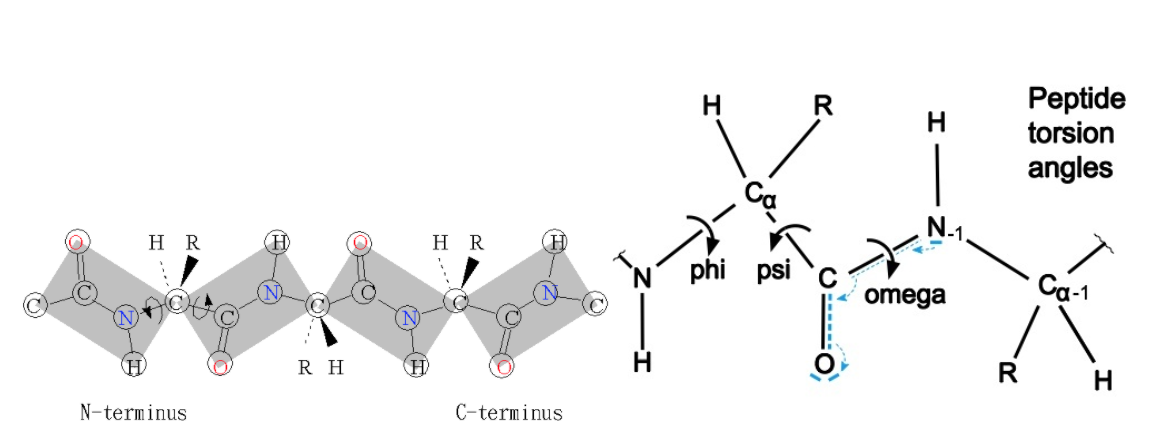
图1 蛋白质链上肽平面之间形成的二面角以及 两个残基脱水缩合后的 ψ,φ和W(图片来自swiss-model)
图一的左图中,Calpha 分别连接的两个肽平面是可以自由转动的,但是由于周围原子的影响,在转动过程有一定的障碍。 右图中phi(CNCalphaC)和psi(NNCalphaCN)较为自由,而omega(OCNH)由于肽平面是刚性的一般是180度(极少数是10度)。
一、拉氏图(Ramachandran plot)
其展示了在一个蛋白质结构中所有残基中的phi和psi, 一般是将gly和pro氨基酸的除去,另外处理。我们可以对单个氨基酸做拉氏图,也可以对一条蛋白质链做拉氏图,甚至是多个蛋白质链。分别如下:
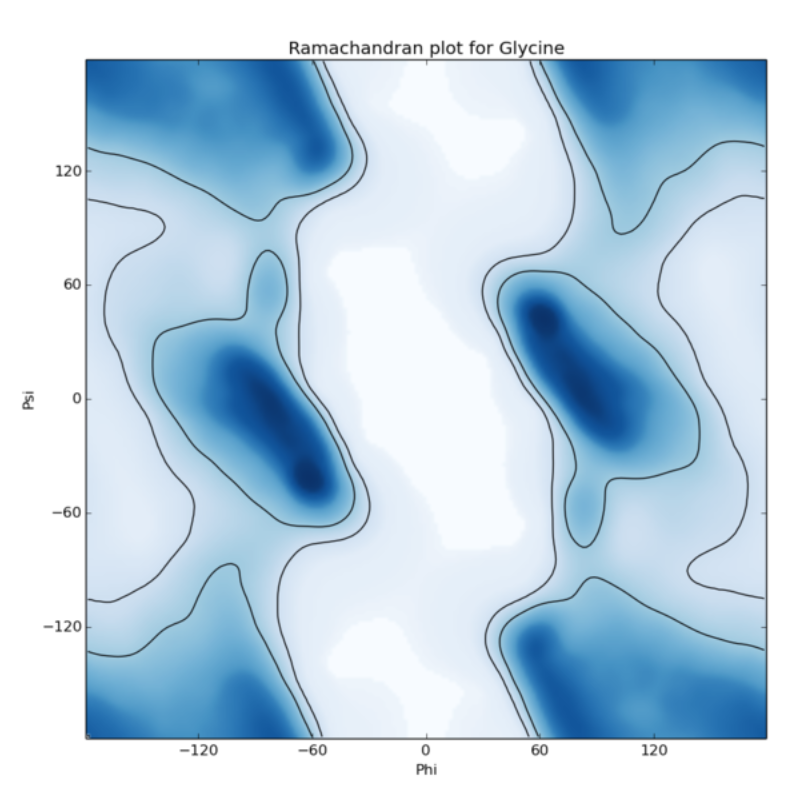
图2. 甘氨酸的拉氏图, 蓝色、浅蓝色、白色分别为构象完全允许、允许、不允许区域

图3. 用散点做的gly的拉氏图,并且标注了alfa,beta,Lalfa二级结构 。而且点集中在完全允许区域,允许区域次之,不允许区域非常少。
拉氏图中关于不允许区域的点: 不允许区域可以理解为高能区域, 只有能量是无限高才会引起完全不允许。所以这些不是无限高的区域中是可以偶尔出现一些点的。换句话说,蛋白质可以花一些能量来驱动残基进入这一区域。
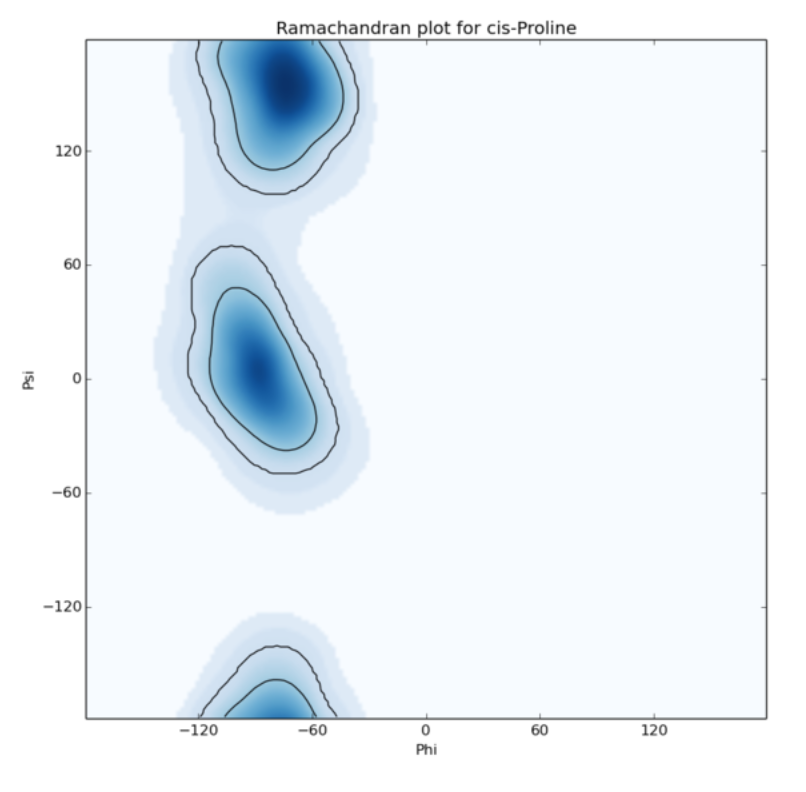
图4、脯氨酸(pro)的拉氏图

图5、出去gly、pro其他氨基酸的拉氏图

图6、2erl(40aa)蛋白质的拉氏图
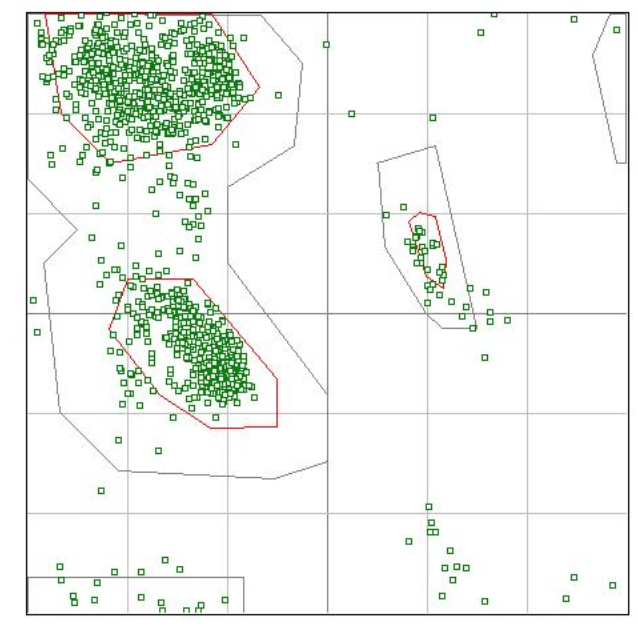
图7. 1epw的拉氏图
三、可以做拉氏图的软件procheck
可以在线预测, 也可以下载单机版预测。
在线版本只需要http://www.ebi.ac.uk/thornton-srv/databases/pdbsum/Generate.html
分别上传PDB格式的蛋白质文件和个人邮箱。等待结果回传到邮箱。 点击进入结果页面,点击右上角的procheck按钮可以进入如下图页面:
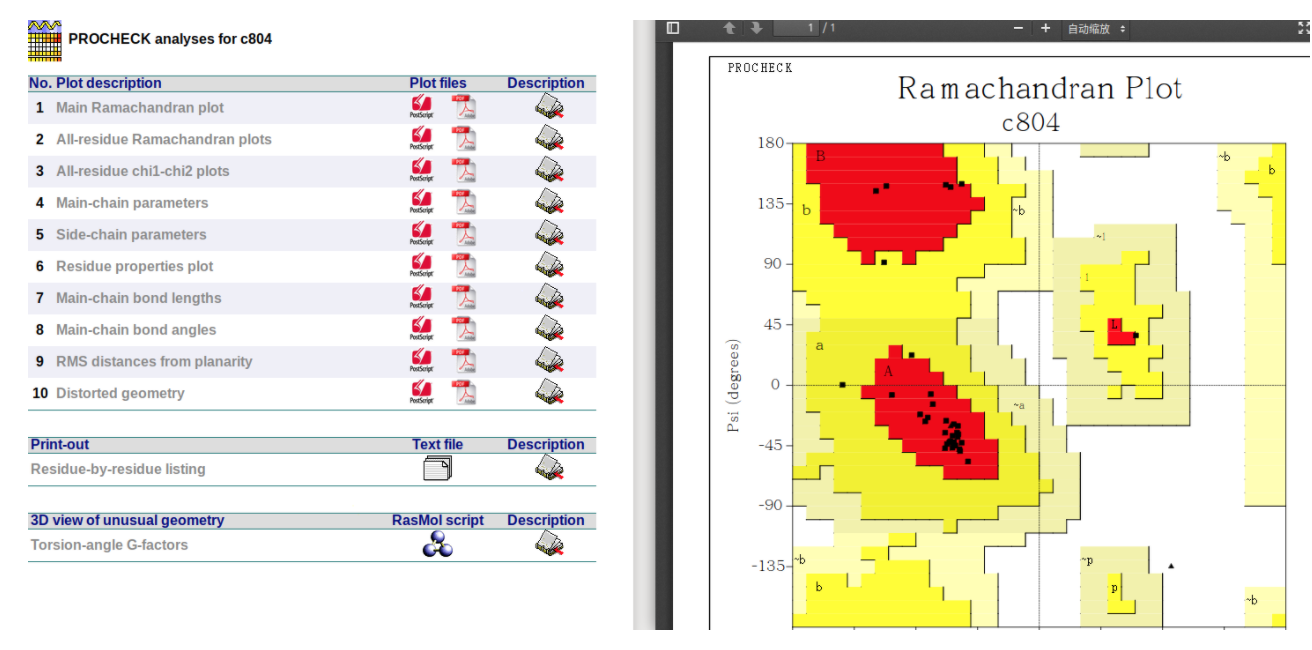
点击左图第一个main Ramachandran plot即可得到右图的拉氏图。下面对右图进行说明
其中,该图是pdb名为2erl的蛋白质结构中所有残基(除去gly,pro)拉氏图,其横纵坐标分别为phi和psi。其中深色区域(红色)代表的是core区域,残基较喜欢的构象区域。理想的,我们希望散点90%以上落到这些区域,这样这些残基具有较好的构象空间。这些区域分类来自Morris et al. (1992).
这些区域标记如下:
A - Core alpha L - Core left-handed alpha
a - Allowed alpha l - Allowed left-handed alpha
~a - Generous alpha ~l - Generous left-handed alpha
B - Core beta p - Allowed epsilon
b - Allowed beta ~p - Generous epsilon
~b - Generous beta
下载需要登录procheck网址后:
进入网址后下载5个文件,解压打开procomp.scr文件,将f77修改为相应的fortran的编译器名称,我所安装的为gfortran。保存后,在终端中键入:cshprocomp.scr,即开始安装。
参考资料
- https://blog.csdn.net/MCANDML/article/details/80672174
- Laskowski R A, MacArthur M W, Moss D S, Thornton J M (1993).PROCHECK - a program to check the stereochemical quality of protein structures.J. App. Cryst., 26, 283-291.
- Morris AL, MacArthur MW, Hutchinson EG, Thornton JM (1992). Stereochemical quality of protein structure coordinates. Proteins, 12, 345-364.
