【6.2.2】CHOPCHOP-v2
在短短3年间,CRISPR基因组编辑已改变了生物学,其流行性和效力不断增长。继续有新的CRISPR 用于定位最佳靶标的effectors和规则的报道,这突显了对计算CRISPR靶向工具的需求,以整合这些规则并促进靶标选择和设计。 CHOPCHOP是用于基于CRISPR和TALEN的基因组编辑的最广泛使用的网络工具之一。它的总体原则是提供一个既直观又强大的工具,可以为新手和有经验的用户提供服务。在此重大更新中,我们介绍了用于下一代CRISPR技术进步的工具,包括Cpf1和Cas9切口酶。我们支持许多新功能,这些功能可以提高CHOPCHOP的定位能力,可用性和效率。为了增加靶向范围和特异性,我们为定制长度的sgRNA提供了支持,并且我们使用了来自多个大规模研究的模型来评估整个sgRNA及其周围区域的序列组成。这些和其他新功能,以及用于提高可用性和支持不断增长的生物体列表的更新界面,使CHOPCHOP成为CRISPR基因组编辑的主要工具之一。可以在 http://chopchop.cbu.uib.no 找到CHOPCHOP v2
一、前言
用于基因组编辑的CRISPR细菌系统的发现和采用引发了生物学的一场革命:现在,在许多生物体中都可能进行靶向突变,包括许多以前不适合进行基因操作的生物。这既改变了我们回答生物学问题的方法,也消除了纠正人类遗传疾病的可能性。
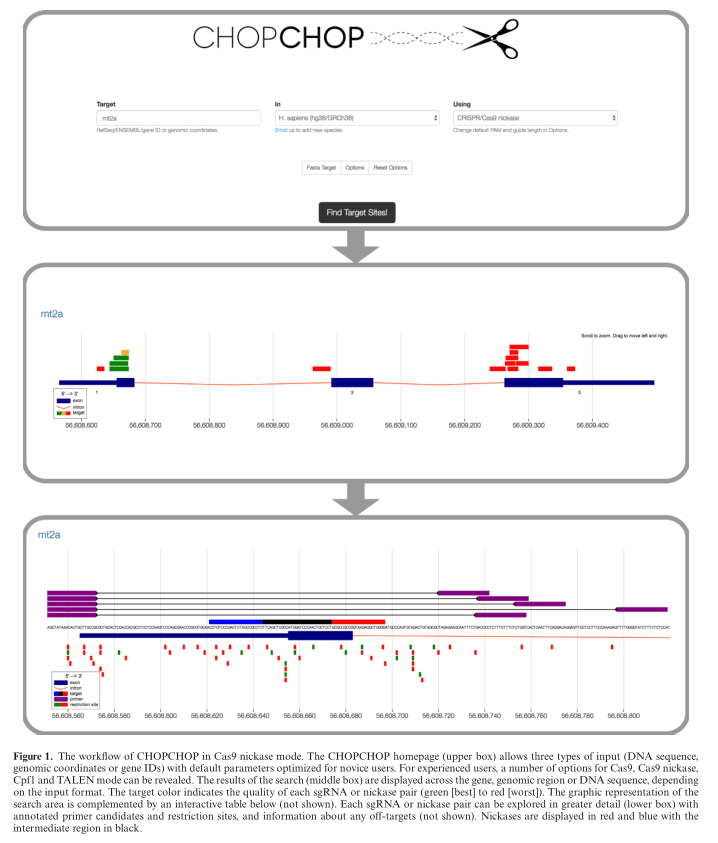
最初利用化脓性链球菌( Streptococcus pyogene)II型系统(1-3)进行CRISPR基因组编辑是基于两组分系统:Cas9核酸酶和单个指导RNA(sgRNA),其将核酸酶引导至基因组中的特定位点。在存在sgRNA的情况下,Cas9定位目标位点并产生双链断裂(DSB)。 DSB通过宿主的非同源末端连接途径进行修复,但修复通常不完善,会形成插入缺失,并在许多情况下产生移码突变。自该技术问世以来,改善技术的研究集中在两个主要挑战上:优化切割效率和切割特异性。为给定基因设计的大部分sgRNA产生的切割率较低或为零,许多sgRNA具有在基因组中混杂结合的能力,这可能导致脱靶诱变(4-10)。为了解决这些问题,研究集中于鉴定有助于有效(和无效)sgRNA的序列特征(11-16),以及开发新的CRISPR变体,以扩大核酸酶的靶向范围和特异性(17-16)。由于众多因素对最佳sgRNA靶标选择产生了影响,因此有必要使用软件来帮助选择CRISPR靶标进行实验。 CHOPCHOP(21)提供了一个直观的在线目标选择环境,根据最新的大规模研究,可以优化效率和特异性(efficiency and specificity),并通过友好的图形界面来进行引物设计和限制性酶切位点识别(图1) 。 CHOPCHOP的这一新更新提供了sgRNA设计新选项,以及对sgRNA目标进行评分和排名的其他指标,从而提供了更大的灵活性。
二、2016版的改进
CHOPCHOP接受多种生物体的多种输入格式(基因标识符,基因组坐标和粘贴序列),并提供即时,可视化输出以及可下载的数据(GenBank,文本表和FASTA文件)。 在此新版本中,用户还可以单击UCSC浏览器中的输出数据,从而可以在带注释的基因组特征的背景下查看结果, 例如转录因子结合位点和染色质结构及可及性(图2)。

CHOPCHOP results can be exported to the UCSC browser with a single click. Here, the sgRNAs (in this example in promoter-targeting mode) are viewed in the context of the genome. The tracks displayed in this example are DNase sensitive regions, common SNPs and CpG islands.
CHOPCHOP可以灵活地靶向蛋白质编码和非编码基因的子区域,包括编码区域,UTR,剪接位点和单个外显子。 在这个新版本中,我们还提供了一种启动子靶向模式(图2),用于分别使用catalytically dead Cas9(dCas9)或transcriptionally active dCas9(例如dCas9-VP64)下调或上调基因表达的实验(23–25) 。 CHOPCHOP使用Bowtie(26)确定所有sgRNA的潜在脱靶位点,并使用Primer3(27)自动生成目标位点的引物。 可以指定引物的长度和退火温度,以及扩增子的大小。 CHOPCHOP在动态视觉界面中可视化所有元素,其中包括有关限制位点的信息,可用于下游验证。
除了这些改进之外,CHOPCHOP的新版本还引入了以下主要新功能。
2.1 支持新一代CRISPR效应器 Support for a new generation of CRISPR effectors
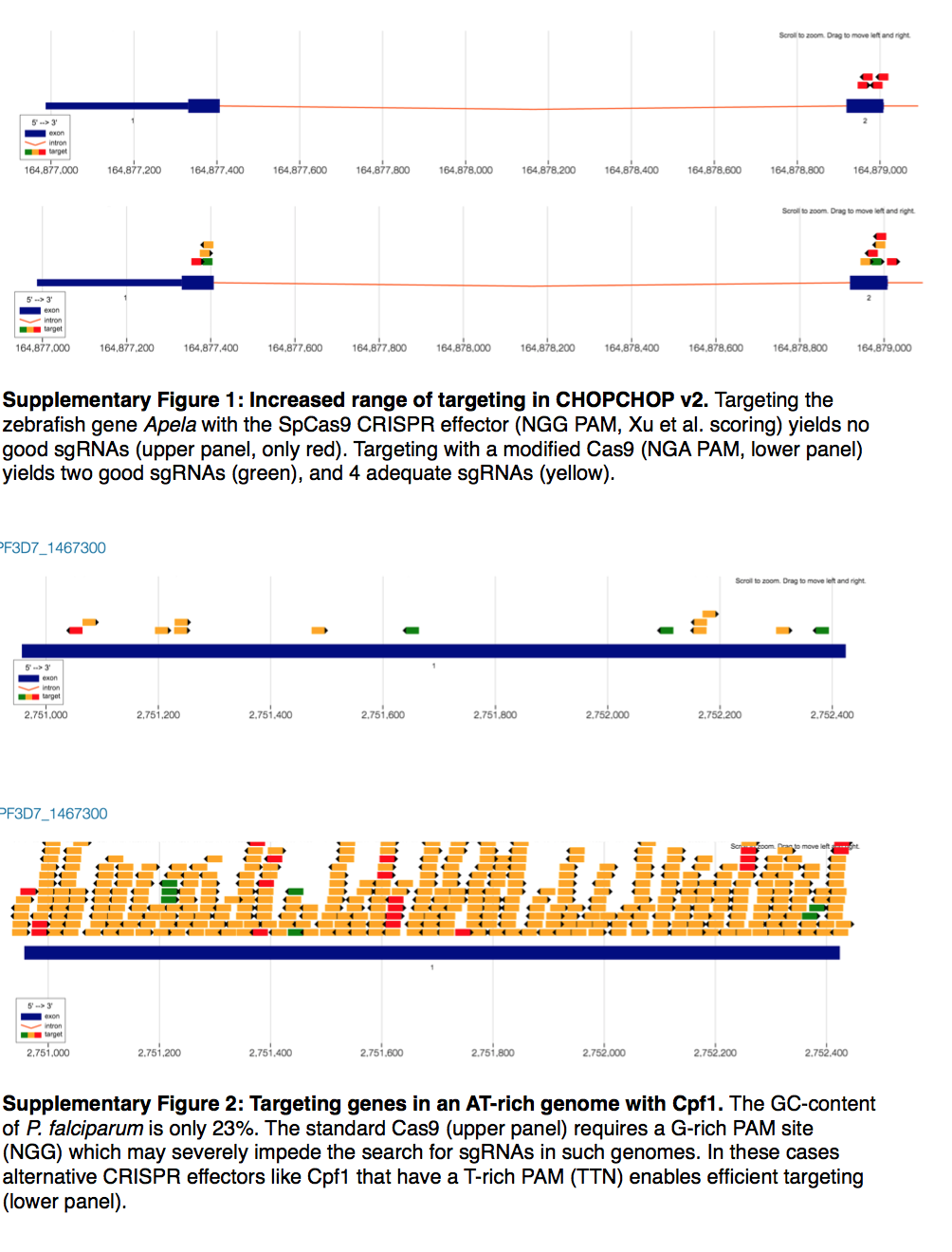
使用最广泛的CRISPR效应子(effector)是Cas9,衍生自II型化脓性链球菌(type II S. pyogenes)系统。虽然RNA介导的Cas9靶向在选择靶位点方面具有很大的通用性,但限制因素是需要与靶标相邻的NGG原间隔子相邻基序(PAM)基序。这种基序的出现在大多数基因组中并不罕见,但是它施加了一个限制,对于实现某些实验或靶向小基因所需的高基因组精度可能是不利的。新一代的CRISPR效应器通过提供替代的PAM motif(补充表S1,补充图S1和2),极大地扩展了可行靶标的范围。 CHOPCHOP现在为其他CRISPR效应子提供支持,包括来自Acidaminococcus的Cpf1,其利用富含AT的PAM(17)和化脓性链球菌,嗜热链球菌,金黄色葡萄球菌和脑膜炎奈瑟氏球菌的Cas9同源物(28)。此外,CHOPCHOP还接受用户定义的自定义PAM,它们可以锚定到sgRNA的5'(Cpf1)或3'(Cas9)末端。该字段接受标准IUPAC核苷酸字母(29),包括歧义码。因此,CHOPCHOP为任何当前已知的CRISPR效应子的序列需求提供支持,并能够立即采用任何新的CRISPR效应子。这大大增加了可以用CHOPCHOP设计的CRISPR实验的靶向范围,包括改善了对富含AT的基因组(如恶性疟原虫)的靶向性(补充图S2)。

2.2 优化切割效率的新规则 New rules for optimizing cutting efficiency
CRISPR sgRNA可以按2个标准进行排名:
- 效率-特定sgRNA促进切割的可能性,
- 特异性-sgRNA结合脱靶位点的可能性。
根据实验研究,CHOPCHOP的初始版本提供了两个简单的效率指标:
- 首先,sgRNA的GC含量(理想的是介于40%和80%之间)
- 其次,sgRNA是否在位置20处含有G(11,30)。
自从CHOPCHOP首次发布以来,已经提出了一些改进。 Doench等人的研究。产生了一个大型数据集来计算各种sgRNA的效率(14),最近,同一小组进一步完善了计算辅助sgRNA设计的规则(13)。 Moreno-Mateos等进行了类似的筛选发现,取决于鸟嘌呤富集和腺嘌呤较少的sgRNA稳定性,是sgRNA效率的主要决定因素(12)。
Chari等进行了一项利用慢病毒整合到转录活性区域中的偏见的研究,该研究:
- 发现可及的(accessible)DNA更适合用Cas9切割;
- 分离了DNA可及性和序列组成对sgRNA效率的影响。
CHOPCHOP用户现在可以在DNase I超敏位点的背景下,在UCSC浏览器(22)中查看结果,以预测可访问的DNA区域(图2)。最后,徐等人的meta研究,编译了多个数据集的序列特异性,以构建聚合模型(15)。我们在新版本中实现了所有这些指标,以便为用户提供广泛的指标选择(默认值为Xu等指标)。使用这些方法,CHOPCHOP现在可以使用特定于位置的评分矩阵或支持向量机对每个sgRNA进行评分,这些矩阵考虑sgRNA的每个单独位置以及PAM下游和结合位点上游的序列。在结果表中,该分数被报告为“效率分数”。
其他因素在sgRNA是否可能切割其预期靶标方面也起作用。最近,我们和其他人表明,sgRNA的自身互补性可以抑制其有效整合到效应复合物中(12,31)。现在,CHOPCHOP包含了Thyme等人的self-complementarity score研究(31),该方法计算了sgRNA内以及sgRNA与骨架之间潜在的4 bp茎数。因此,用户可以使用该选项选择避免具有自身互补性的sgRNA。
2.3 提高特异性的策略 Strategies to increase specificity
CRISPR实验中的一个重大挑战是在预期靶位以外的位点诱导裂解的可能性。缓解切口问题的一种新兴工具是配对切口酶(paired nickase)方法(32)。与天然CRISPR效应子不同,切口酶(nickases)已被修饰为仅切割一条DNA链。为了创建DSB,必须将一对切口酶靶向相反的链,并彼此结合10-31 bp之内(32)。这些要求极大地降低了创建脱靶DSB的可能性,CHOPCHOP现在增加了对配对切口酶实验的支持。在此模式下,指定距离内(默认或用户定义的)相对链上的位点将配对为潜在的切口酶位点。对于这些site,除了默认的脱靶搜索外,还会评估每对站site脱靶情况,在这种情况下,绑定和切割会导致DSB。通过围绕“断裂”区域的两个CRISPR靶标使切口酶位点可视化(图1)。
最近的研究强调指出,在确定偏离目标时需要搜索两个以上的错配(10),因此CHOPCHOP现在最多可以计算三个错配的偏离目标。尽管已经报道了三个以上错配的脱靶位(10),但证据表明几乎不对四个错配或更多错配的预测位点进行切割(10),因此,在选择sgRNA时需要进行搜索,绝大多数此类预测位点都会产生误导,并且不必要地浪费时间。
已显示出减少脱靶裂解的另一种策略是使用截短的sgRNA(10,20)。除了增加特异性外,通常20 bp的5’缩短也增加了靶向范围。因此,新版本的CHOPCHOP支持用户定义长度的sgRNA。
因此,此版本的CHOPCHOP支持许多新功能:
- 提高靶向更广泛序列的能力,
- 更彻底地预测基因组中潜在的脱靶位点。
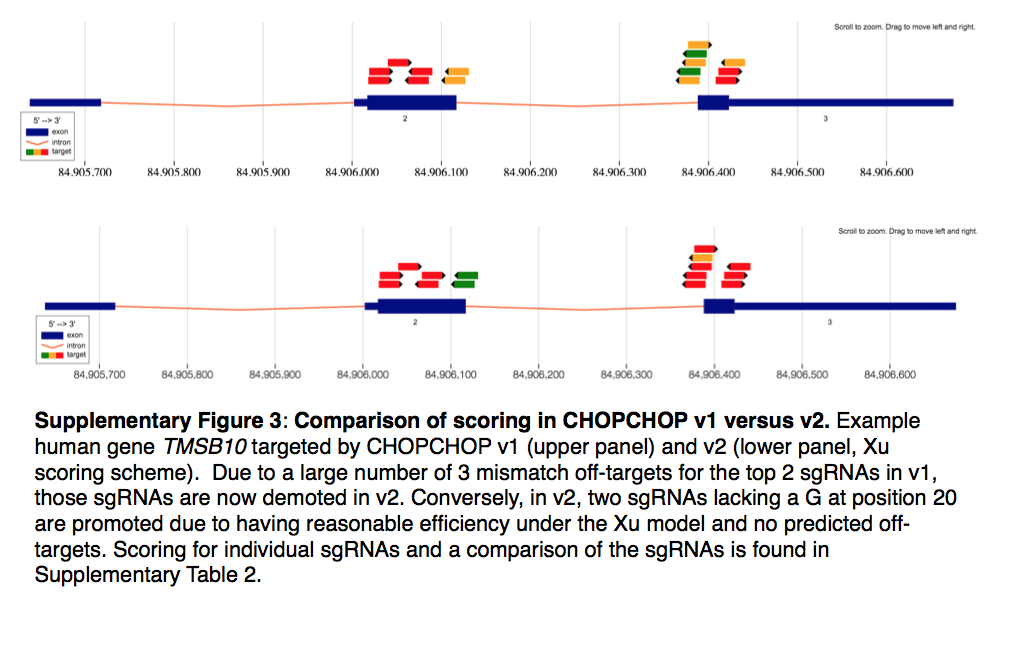
有关扩大目标范围以及在新旧版本CHOPCHOP之间增加评分系统的示例,请参见补充图S3和表S2。

2.4 新基因组
除了一系列新功能外,CHOPCHOP还努力满足对新基因组和基因注释集的所有要求。 到目前为止,我们已经合并了所有收到的询问,CHOPCHOP现在支持总共32种生物。 此外,所有基因组均已更新至最新装配,可通过主页上的链接轻松提交有关新物种的建议。
三、讨论与未来发展
CHOPCHOP的首要原则是提供一个既直观又功能强大的工具,可以为首次使用和有经验的用户提供服务。基本模式为基本用户提供了优化的默认值,而更高级的用户则可以根据相关性和实用性从文献中精选出的各种选项中进行选择。所有选项均以列表和组织的方式呈现,以帮助用户在设计CRISPR实验时快速可视化和评估选项。
此版本保留了先前版本的总体布局,但将视觉配置文件更新为现代外观并容纳了新功能。该网站现在可移动和平板电脑使用,为了简化用户体验,我们使用cookie来记住物种的选择和针对后续搜索的目标选项。所有已报告的错误均已修复,现在已针对将来的开发对实现进行了优化,以促进快速采用任何将来的效应子和大规模研究中的新靶向数据。这次重大更新使CHOPCHOP保持为最易用,功能强大的CRISPR靶向工具之一。
我们要感谢Alexander F. Schier和Michele Clamp的支持,以及许多为改进CHOPCHOP和此手稿提供有用建议的用户,特别是Etsuko Moriyama,William C. Cheng,Miguel A. Moreno-Mateos和Antonio Giraldez。我们特别感谢Maximilian Haeussler协助将CHOPCHOP结果整合到UCSC基因组浏览器中。
参考资料
