【4.2.3】CHOPCHOP-v3
CRISPR-Cas系统是功能强大的基因组编辑工具,可在多种生物和细胞类型中起作用。最初开发该技术是为了诱导DNA的定向突变,但是CRISPR–Cas现在已针对多种用途进行了改造,可以靶向核酸。 CHOPCHOP是用于识别CRISPR–Cas单向导RNA(sgRNA)靶标的网络工具。在CHOPCHOP的主要更新中,我们将工具箱扩展到了剔除(knockouts)之外。我们介绍了Cas13靶向RNA的功能,其中包括对替代转录异构体和RNA可及性预测的支持。我们整合了新的DNA靶向模式,包括CRISPR激活/抑制,针对基因的靶向富集以进行长时间阅读测序以及预测Cas9修复结果。最后,我们扩大结果页面的可视化范围,以揭示其他同工型和下游ATG位点,这将有助于用户避免截短(truncated)蛋白的表达。 CHOPCHOP Web工具现在支持200多个基因组,并且我们发布了一个命令行脚本,用于运行更大的任务并处理不受支持的基因组。可以在 https://chopchop.cbu.uib.no 上找到CHOPCHOP v3
一、前言
CRISPR-Cas的使用在现代分子生物学中无处不在。首次作为一种工具引入基因组中的修复诱导突变,效应蛋白的催化死亡或融合形式的出现将CRISPR–Cas转变为用于靶向的通用工具。例如,CRISPR–Cas已被用于将新序列引入基因组(1-3),激活(4,5)或抑制(6)转录,作为测序的富集工具(7,8),用于靶向超突变(9)作为诊断工具(10),执行全生物谱系追踪(11),靶向RNA分子以进行破坏(12)或编辑(13),并追踪活细胞中的转录本(14)。
所有CRISPR–Cas应用都使用sgRNA将CRISPR效应蛋白(Cas)定向到其靶标。从理论上讲,CRISPR-Cas靶向仅要求sgRNA及其核酸靶标之间具有互补性,但许多研究表明,有效靶向遵循更复杂的规则(15-21)。例如,特定核苷酸在靶序列中的位置,靶位点的可及性及其侧翼区的序列都可以影响效率。已经在大规模研究中测量了Cas9和Cas12a / Cpf1的靶向效率,并结合了基于机器学习的方法来优化切割效果(15,21)。
还有其他因素可以影响或阻止无效突变体的产生。例如,将移码突变引入到距离起始密码子太近的位置会允许在下游ATG处进行翻译起始,从而导致意外的蛋白质产生。在其他情况下,靶向仅存在于同工型子集中的外显子可以防止无效突变的产生。最后,由于转录适应或遗传补偿,CRISPR-Cas基因编辑可产生混淆的表型。例如,最近的一项研究表明,突变mRNA的降解可能导致相关基因的上调(22)。在这些情况下,删除启动子可能是产生敲除的更可靠的方法。
许多CRISPR-Cas应用要求产生移码突变来破坏基因功能,这需要DNA修复事件,其中插入或缺失的核苷酸数目不是三的倍数。出人意料的是,已证明双链断裂(DSB)修复不是随机的:使用相同sgRNA的Cas9诱导的DSB通常会产生相同的突变(23)。最近,这已被纳入预测DSB修复是否会引起移码突变的模型中(24)。
总而言之,CRISPR–Cas系统已经适应了广泛的用途选择,并且多种因素会影响每种模式,因此必须存在用于目标选择的直观软件。 CHOPCHOP网络工具的这一新更新结合了新的CRISPR–Cas靶向模式,并在改进的用户友好界面中预测了移码突变频率。
二、新版本中的改进
像以前的版本一样,CHOPCHOP处理来自(i)基因和转录物标识符,(ii)基因组坐标和(iii)粘贴序列的输入,并以多种输出格式提供结果。 界面很简单(图1),要求用户在默认模式下进行四个选择:(a)靶基因/isoform/区域,(b)生物,(c)CRISPR效应子(例如Cas9,CasX或Cas13), (d)目的(例如,击倒,击倒,镇压)。 高级用户可以通过单击“选项”按钮来调整默认设置(图1)。
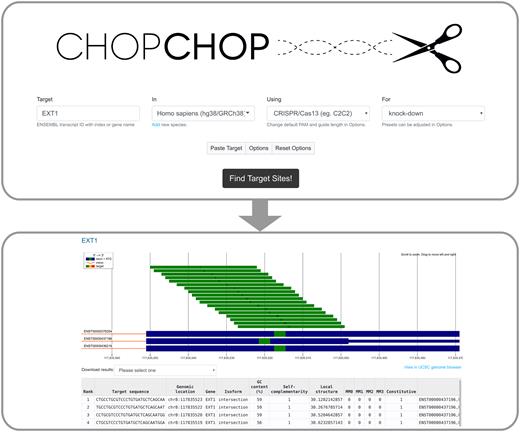
The workflow of CHOPCHOP when targeting RNA for knock-down. The CHOPCHOP homepage (upper box) require four types of input: (i) target, (ii) species, (iii) CRISPR effector and (iv) the purpose of the experiment. Default options will be adequate for most users, but advanced options can be revealed using the ‘Options’ button. The results of the search (lower box) are displayed along with all isoforms of the target gene. The target color indicates the quality of each sgRNA or nickase pair (green [best] to red [worst]). Below the graphic representation an interactive table allows for exploring each guide in greater detail.
尽管CHOPCHOP查询通常会在几秒钟内运行,但繁忙的流量会导致高峰时段的拥塞。因此,我们引入了一个排队系统,以确保所有用户的优先级。新系统在初始查询后最多保留48小时的结果和计算结果,如果稍后进行相同的搜索并与协作者共享结果,则可以快速访问(通过缓存)。为了提高大型查询的速度,CHOPCHOP还支持通过:
- GC含量(默认值为10–90%)
- sgRNA中存在自我互补性来(self-complementarity)对sgRNA目标进行预过滤。
这样可以大大减少计算时间。
已经进行了一些调整,以改善结果页面的可视化效果。值得注意的是,现在在同工型可视化中为框内下游ATG着色(图1),以帮助用户避免下游翻译的启动。此外,现在可以使用任何条件对结果表进行排序。
除了这些改进之外,最新的CHOPCHOP还引入了以下新的主要功能。
2.1 靶向转录组
CRISPR-Cas的最广泛用途是将突变引入DNA。但是,CRISPR–Cas系统现已设计为靶向RNA。例如,CRISPR–Cas13已被用于转录本敲除(25),活细胞转录本成像(14)和RNA碱基编辑(13)。
CHOPCHOP现在允许CRISPR–Cas13靶向,并通过在整个转录组而不是基因组中搜索脱靶来实现此功能。 RNA靶向的一个重要方面是避免具有高结构的区域,该区域会降低Cas13的可及性 (accessibility)(12、14、26)。 CHOPCHOP根据已发布的建议(14),使用ViennaRNA程序包中的RNAfold(27)计算RNA可及性。简而言之,在70个核苷酸的窗口中计算accessibility,从而获得转录本中给定核苷酸位置未配对的可能性。对于每个靶标,我们采用sgRNA靶向的每个位置的平均结构概率(14)。
类似于DNA靶向模式中的原间隔子相邻基序(PAM, protospacer adjacent motifs),我们支持任何5’或3’原间隔子侧翼序列(PFS)进行RNA靶向。默认为Cas13a PFS(目标3’端的H)。与PAM一样,PFS应该存在于DNA或RNA靶序列中,而不能存在于sgRNA中。 CHOPCHOP提供适当的sgRNA序列,确保在订购sgRNA寡核苷酸时用户不包括PFS。
2.2 靶向基因组的新模式
CHOPCHOP v3扩展了DNA靶向模式的数量。这些包括:
(1. 纳米孔富集模式。靶向测序是一种用于在特定目标区域中获得高质量测序读数的方法。用于富集基因组区域的基于PCR的方法有一些限制,例如可以富集的区域的最大长度。相比之下,CRISPR–Cas提供了一种在牛津纳米孔技术(ONT)进行扩增和测序之前切除基因组区域的强大方法(7,8)。 CHOPCHOP中的纳米孔富集模式允许用户通过排除低效指南来鉴定大区域(最大40kb)侧翼的gRNA对。该模式会过滤所有具有预测的自我互补性的sgRNA,因为这可能会对整体Cas9活性产生抑制作用(8,28和ONT个人交流); 2. 敲入模式。这可以鉴定出与敲除模式相同的sgRNA,但设计的同源性臂可达2 kb,根据最近的研究(1-3),该臂可用于靶向插入。 3. 激活/抑制模式。设计这些模式是为了与Cas9融合蛋白一起使用,目的是激活或抑制基因。具体来说,这些模式根据(29-31)中指定的指导原则靶向启动子区域及其侧翼位点,以使激活/抑制域与转录起始位点紧密接近。
对于Cas9基因敲除模式,我们现在创建DSB修复结果的预测(24)。该模型估计给定sgRNA导致移码突变的可能性。此外,我们增加了Cas12a / Cpf1的效率得分(21),并将“ Doench”效率得分更新为最新版本(15),现在这是Cas9基因组定位的默认得分指标。
2.3 扩展基因组和靶向
CHOPCHOP现在支持200多个基因组,包括用于基因组靶标的基因注释以及用于RNA抑制的三个转录组(人,小鼠和斑马鱼)。虽然以前版本的CHOPCHOP需要选择特定的isoform进行靶向,但该新版本允许用户靶向整个基因。在其默认的“交叉”模式下,CHOPCHOP v3仅搜索每个isoform中存在的sgRNA(图1)。可以通过选择“联合”模式来禁用此模式,该模式将显示所有转录本中的所有sgRNA,以及指示该sgRNA是否靶向组成性外显子的列。
2.4 命令行版本
除了Web界面之外,我们还提供CHOPCHOP命令行版本的代码,该代码可以在本地运行,适用于较大的查询或显示。除了用于大型实验的其他功能外,该工具还包括网络界面的所有功能,例如设计与基因组中任何序列都不匹配的对照sgRNA的能力。 CHOPCHOP的命令行版本与ampliCan兼容,后者是用于基于序列的突变评估工具(32)。
三、讨论与未来发展
自成立以来,CRISPR领域经历了持续不断的创新,需要并行开发适应新发现和新技术的生物信息学工具。 在短短几年内,CRISPR–Cas已成为一种强大的靶向工具,可在多种情况下沉默和激活DNA和RNA,每种情况都需要应用特定规则。 CHOPCHOP的最新版本通过添加反映不断扩展的CRISPR工具箱的新功能来应对这一挑战。 到目前为止,我们已经容纳了200多个研究小组最喜欢的物种,随着我们继续改善CHOPCHOP的功能,我们将继续容纳新的转录组和基因组。
总之,这一重大更新扩展了CHOPCHOP工具箱,保持了其作为最易用,用途广泛的CRISPR-Cas靶向工具之一的地位。
The CHOPCHOP (version 3) server is available at https://chopchop.cbu.uib.no; the python code for local installation is available at https://bitbucket.org/valenlab/chopchop.
参考资料
