【8.3.5.3】IRESbase
http://reprod.njmu.edu.cn/cgi-bin/iresbase/index.php
- 当前版本的 IRESbase 包含 1328 个 IRES,包括分别来自 11 个真核生物和 198 种病毒的774 个真核 IRES和 554个病毒 IRES 。
- 由于 IRESbase 仅收集具有功能证据的最小长度 IRES,因此 IRESbase 中 IRES 的中位长度为 174个核苷酸。
- 通过将 IRES 映射到人类 circRNA 和长非编码 RNA (lncRNA)、2191 个 circRNA 和 168 个 lncRNA 被发现包含至少一个完整或部分 IRES 序列。
- IRESbase 可在 http://reprod.njmu.edu.cn/cgi-bin/iresbase/index.php 获得
两个 IRES 数据库 IRESdb 和 IRESite 分别于 2002 年和 2005 年建成[15]和[16] 。此外,Rfam数据库收集 IRES 作为一种顺式调节 RNA 元件[17]。然而,这些数据库仅包括 mRNA 中的 IRES。最近,IRES 元件也在环状 RNA (circRNA) 和长链非编码 RNA (lncRNA) 中被发现[18]。发现circ-FBXW7、circ-ZNF609、circ-SHPRH、circPINTexon2和circβ-catenin等 circRNA在 IRES 元件的介导下被翻译成多肽[19],[20]、[21]、[22]、[23] 。lncRNA meloe中两个小的开放阅读框(ORF;120个核苷酸和 141 个核苷酸长)的 翻译是通过 IRES 依赖性机制实现的[24]。由IRES元件驱动的circRNA和lncRNA翻译的鉴定最近引起了更多关注
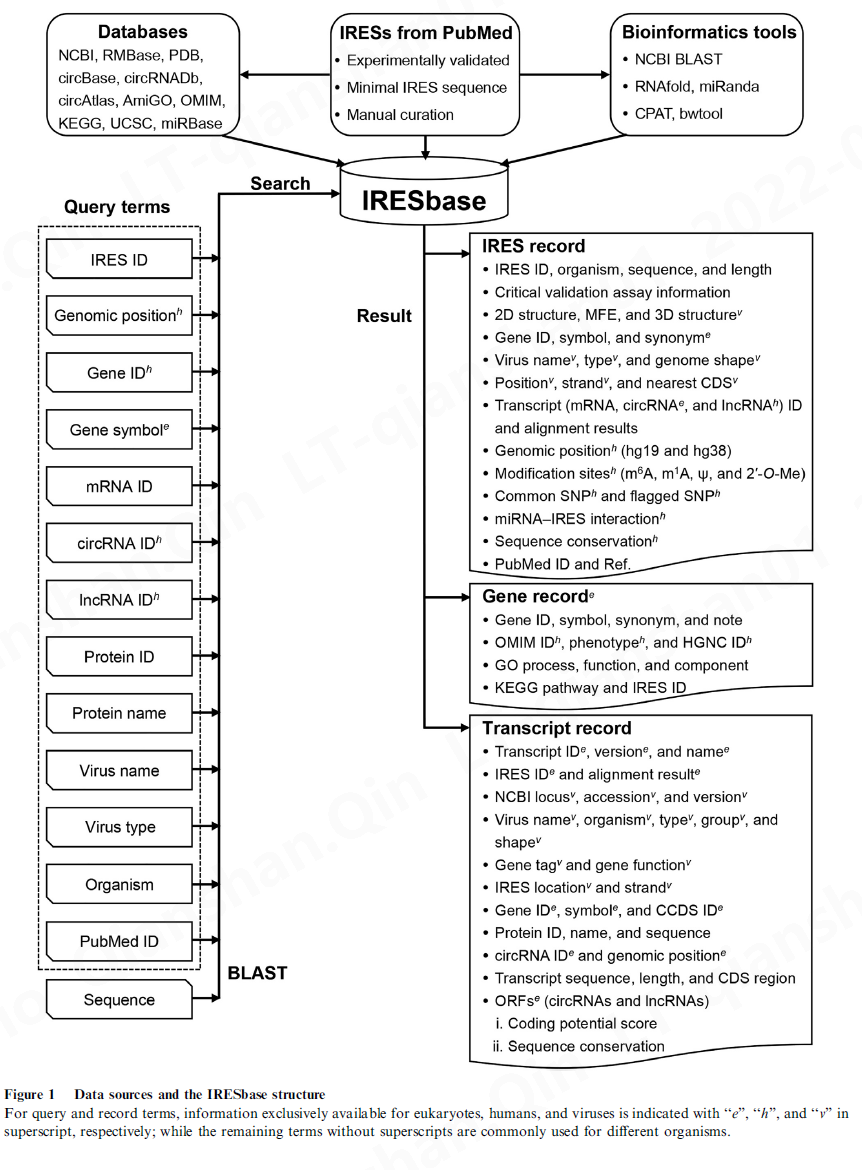
IRESdb 数据库只有 30 个病毒 IRES 和 50 个真核 IRES,而 IRESite 数据库总共包含来自 43 种病毒和 70 个真核 mRNA 的 125 个 IRES。Rfam 数据库构建了 32 个 RNA 家族,其中大约有 11 个病毒 IRES 和 21 个真核 IRES。在这项研究中,我们通过人工管理,开发了一个新颖的非冗余公共数据库,并将其命名为 IRESbase。该数据库包含更新的经过实验验证的 IRES,包括 554 个病毒 IRES、691 个人类 IRES 和 83 个来自其他真核生物的 IRES。
该数据库包括有关基因组位置、序列保守性、单核苷酸多态性 (SNP)、核苷酸修饰、靶向microRNA (miRNA)、宿主基因、宿主转录本(mRNA、circRNA 和 lncRNA)、GO的信息术语(生物过程、分子功能和细胞成分)、KEGG 通路注释和验证测定信息
二、数据收集和处理
2.1 Curation of IRES elements
- 使用 PubMed 从 2019 年 10 月 14 日之前发表的文献中手动搜索经过实验验证的 IRES 序列
2.2 IRES 序列的集合
由于可用的IRES序列相关信息在不同文献中有所不同,我们使用不同的方法手动提取IRES序列(图S1)。这些方法大致可分为三类:(1)直接从其二维(2D)结构图中提取IRES序列;(2)通过宿主转录本和位置信息间接提取IRES序列;(3)利用文献报道的正向和反向引物,利用NCBI BLAST工具提取IRES序列。请注意,如果在文献中实验验证了多个重叠序列包含 IRES 活性,则仅选择其中的最小功能序列(图 S2)。此外,将不同文献的IRES序列进行成对比较,如果序列同一性超过90%,则只选择最短的一个。
参考文献
- IRESbase: A Comprehensive Database of Experimentally Validated Internal Ribosome Entry Sites. https://doi.org/10.1016/j.gpb.2020.03.001
