【4.1.3】几种基于核酸序列构建三维结构的工具
本文将介绍几种基于核酸序列构建DNA/RNA三维结构的工具,可以用于做分子动力学模拟、分子对接等目的。虽然还有很多其它程序也可以构建,如HyperChem等,但本文提供的这些就已经足够用了,且都是免费的。这些工具在产生核酸结构时只需要指定一条链的序列,从5’端到3’端,对于产生双链结构的情况,另一条链的序列总是自动按照规范DNA中标准碱基配对方式自动确定的。这些程序都可以保存成常用的pdb文件格式,并且原子名是规范的。
1 在线工具DNA Sequence to Structure
地址:http://www.scfbio-iitd.res.in/software/drugdesign/bdna.jsp
输入DNA序列以及DNA结构类型,即可立刻返回产生的pdb结构,例如:
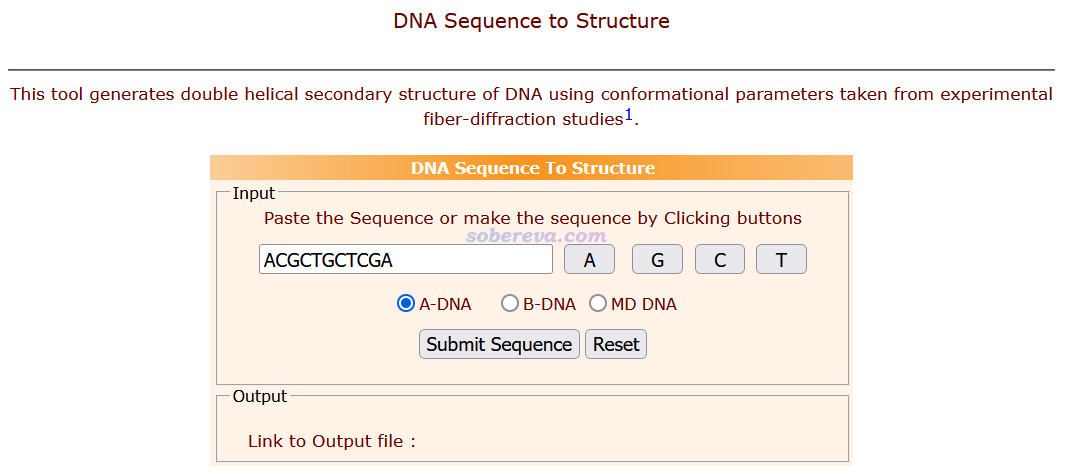
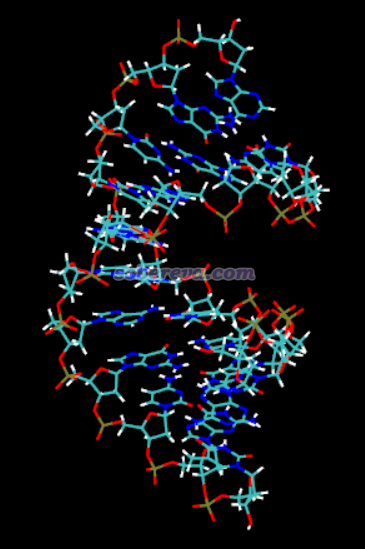
返回的结构用VMD查看:
2 在线工具web.x3dna.org
地址:http://web.x3dna.org
进入后,选Rebuilding - combination of A-, B-, or C-form DNA models。之后可以输入DNA序列由几段构成,比如设了3,点next,若三段内容分别按下面这样设,那么DNA序列就是AAACCCCGGG,且其中AAA部分是A-DNA形式、CCCC部分是B-DNA形式、GGG部分是C-DNA形式。

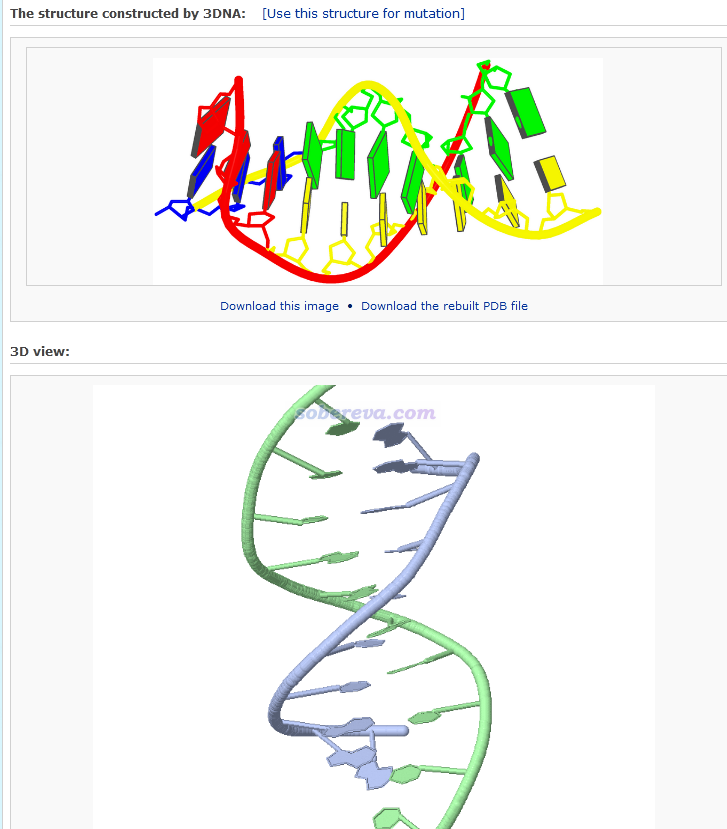
提交之后,过一会儿(有可能时间挺长),看到下图,可以点击链接下载pdb文件
3 AmberTools的NAB
AmberTools程序包可以在 http://ambermd.org 下载,NAB是AmberTools中的组件,AmberTools装好后NAB就可以直接用了。最简单的运行方式为nab test.nab -o test.out,这里test.nab是NAB程序的输入文件(后缀必须是nab)。NAB就像编译器一样会编译出名为test.out的可执行程序,然后运行./test.out即可使里面的指令生效。
NAB可以用于创建DNA和RNA序列。例如创建一个序列为gcgttaacgc的B-DNA结构,就创建一个文本文件比如叫genDNA.nab,里面写以下内容
molecule m;
m = fd_helix("abdna","gcgttaacgc","dna" );
putpdb( "sobDNA.pdb", m );
之后运行nab genDNA.nab -o genDNA,当前目录下就出现了名为genDNA的可执行文件。再输入./genDNA运行之,当前目录下就出现了sobDNA.pdb,是我们要的DNA的结构,DNA的骨架顺着Z轴。
从上面例子可见fd_helix函数里面跟了三个参数,第一个参数控制产生的核酸类型,第二个参数是序列,第三个参数写dna就是生成DNA、写rna就是生成RNA。

4 Gabedit
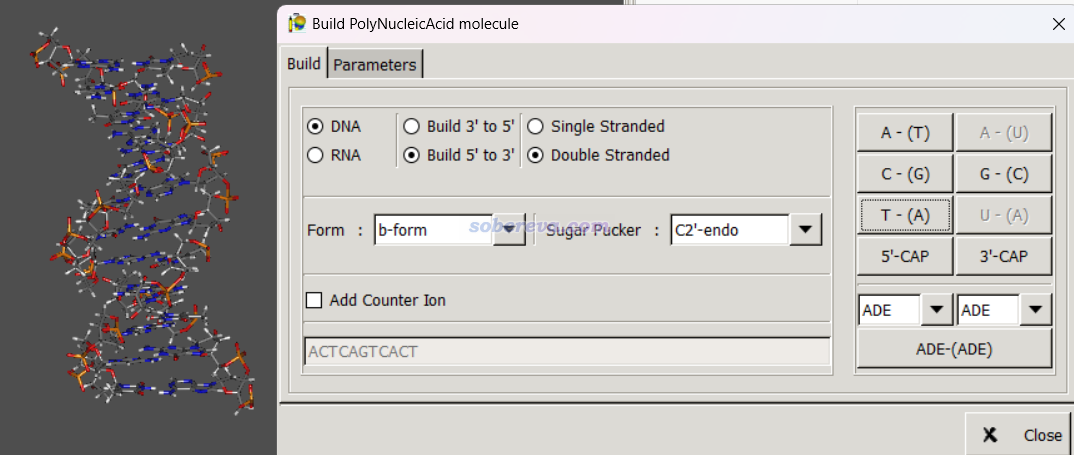
Gabedit是一个免费的可视化程序,可以在 http://gabedit.sourceforge.net 下 载。启动后点击菜单栏Geometry - Draw,然后点右键选Build - polyNucleic Acid,之后一边点击碱基名字的按钮,三维结构一边不断产生,如下图所示。可见核酸类型和结构形式都可以自己定义。如果选上Add Counter Ion,产生的核酸结构的磷酸基旁边还会自动加上Na+作为抗衡离子。构建好后,在图形窗口上点右键选Save as,就可以选择保存成pdb格式。
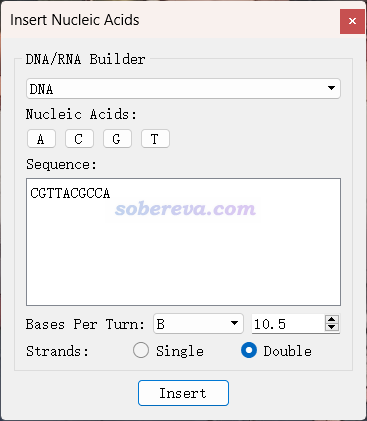
5 Avogadro
Avogadro可视化程序可以在 http://avogadro.cc 免费下载。启动Avogadro后,点击菜单栏的Build - Insert - DNA/RNA,就蹦出了如下窗口。然后一边点击按钮输入核酸序列,一边图形窗口里就可以看到生成的核酸结构。DNA和RNA,单链和双链,结构形式都可以自由选择。
参考资料
