【6.2.3】二级蛋白质结构数据库-CATH(结构分类数据库)
根据结构域的空间特征可以对结构域进行分类。CATH 和 SCOP 是两个重要的蛋白质结 构分类数据库。CATH 数据库( http://www.cathdb.info/ )由伦敦大学 1993 年创建。CATH 这个数据库的名字 C、A、T、H 是数据库中四种结构分类层次的首字母。也就是,所有蛋 白质结构域在 CATH 中被首先分成 4 种 CLASS,这就是 C。四种 CLASS 分别是全α型,全β 型, α +β型,低二级结构型。比如图 1 中第一行这三个蛋白质,很显然左边是全α的,右 边是全β的,中间是 α +β的。

每一个 Class 中的结构域又被具体分为不同的 architecture,也就是 A。A 这一层是按照 螺旋和折叠所形成的超二级结构排列方式分类的。比如α +β这个 class 下的结构可以进一步 分为桶状的,三明治状的,还有滚轴状等 Architecture。每种 Architecture 里的结构域,又可 以根据二级结构的形状和二级结构间的联系更进一步分为不同的 topology,也就是 T。最后 再通过序列比较以及结构比较确定同源性分类,划分出不同的 homologous superfamily,也就 是 H。这样每个结构从粗到细,即从 A 到 H,会有四个层次的分类。注意结构分类是以结 构域为单位进行的,而不是针对整个蛋白。所以 PDB 中的一个蛋白质结构可能对应 CATH 中多个结构域分类。CATH 在分类时既使用计算机程序,也进行人工检查。
CATH 为每一层的每一种结构分类命名,并用数字代号代表这一分类。因此每个结构域 会具有一个分类代码。第一个数字是 C 这一层的分类代码,第 2 个数字是 A 这一层的分类 代码,第 3 个数字是 T 这一层的分类代码,第 4 个数字是 H 这一层的分类代码。
目前 CATH 已为 PDB 数据库中 10 多万个蛋白质结构所涉及的 30 多万个结构域进行了 结构分类,这些分类可以归入两千七百多个蛋白质超家族中。此外,CATH-Gene3D 还为超 过 500 万条来自公共数据库的蛋白质序列进行了结构分类预测。Gene3D 里的信息为绝大多 数还未解析 3D 结构的蛋白质提供了重要的功能研究依据。
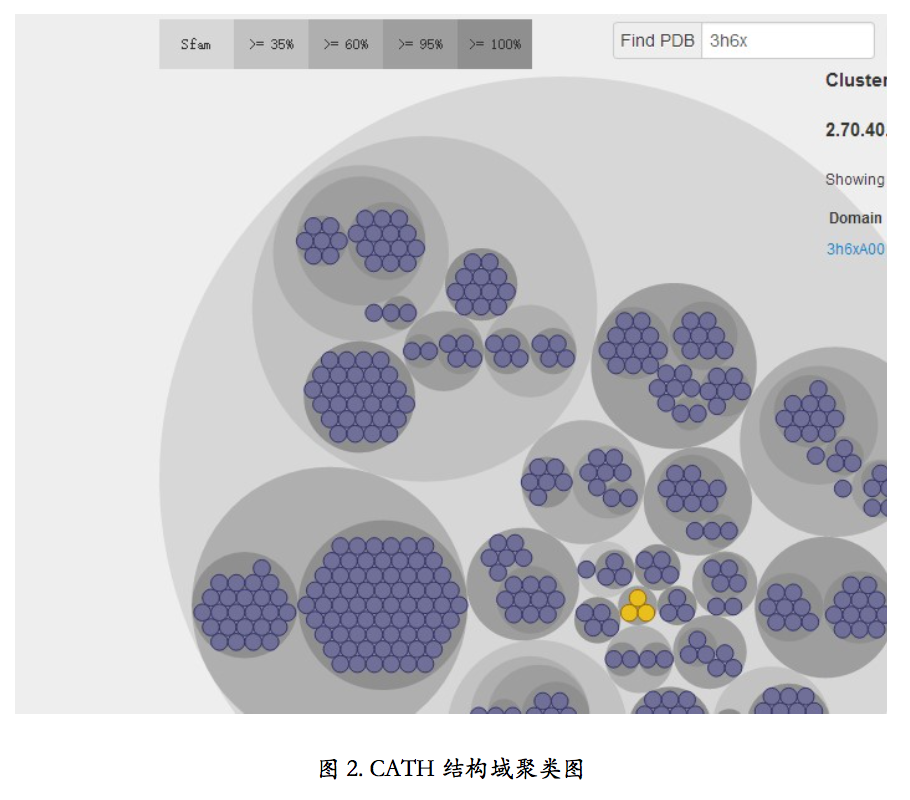
接下来,我们从 CATH 数据库搜索一个 PDB 结构的分类信息。搜索条输入 3H6X,这 是我们在 PDB 数据库里查看过的 dUTPase 的结构。结果显示 dUTPase 蛋白的结构分类代码 是 2.70.40.10。点击这个分类代码,可以获得各层次具体的结构分类信息以及各种结构 相关分析信息。结果页面的下半部分还提供聚类图。这里,CATH 把所有拥有 2.70.40.10 结构分类的结构域,根据他们的序列相似度不同,进行了聚类(图 2)。不同深浅的圈代表 不同的序列相似度。通过这张图,我们可以了解到具有相同结构分类的蛋白质他们在序列水 平上的亲缘关系远近。
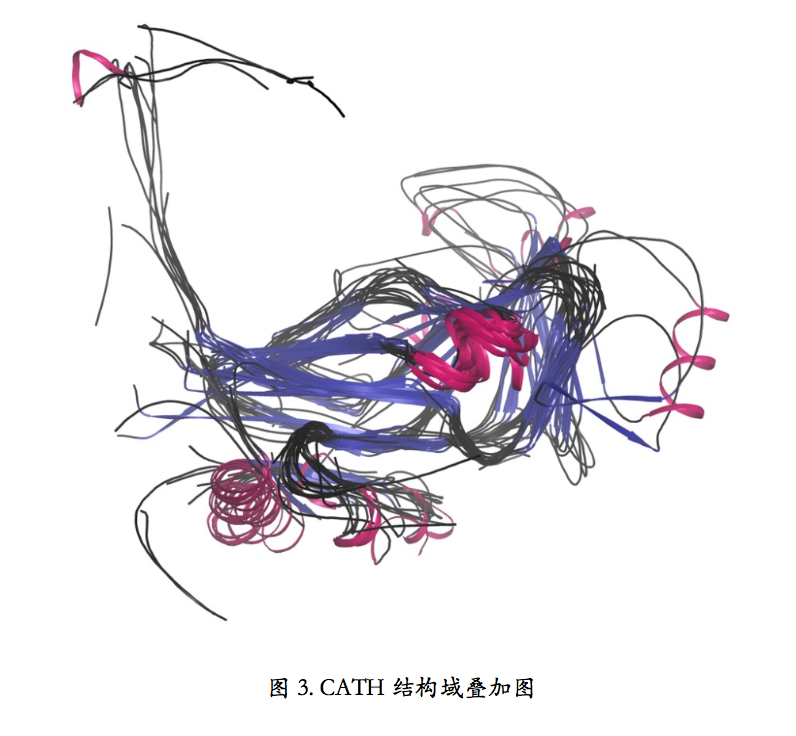
此外,CATH 还从 2.70.40.10 这个结构分类里挑出了 19 个有代表性的结构域,并且 把他们的 3D 结构叠加在了一起(图 3)。从这个图上,我们可以看到这个结构分类的总体特 征以及差异产生的位置。
参考资料
- 山东大学 生物信息学课题组荣誉出品 http://www.crc.sdu.edu.cn/bioinfo 巩晶老师课件
