【3.9】一级核酸数据库-tcga
一、简介
肿瘤基因组图谱 (The Cancer Genome Atlas,TCGA)计划由美国 National Cancer Institute(NCI)和National Human Genome Research Institute(NHGRI)于2006年联合启动的项目。
- 目前收录了来自11000个病人,33个癌症的数据,2.5P的数据量。
- 但是TCGA只对授权的用户开放Level1-Level3数据访问的权限,而普通用户只能访问Level3的分析结果。即TCGA数据库的普通用户无法用Level1的数据进行个性化的高级分析。同时,这些用户也不能有效结合重要的临床信息进行数据的深入挖掘,严重限制用户对数据的有效利用。
收录的癌症类型,详见:https://cancergenome.nih.gov/cancersselected
1.1 TCGA个部门分工
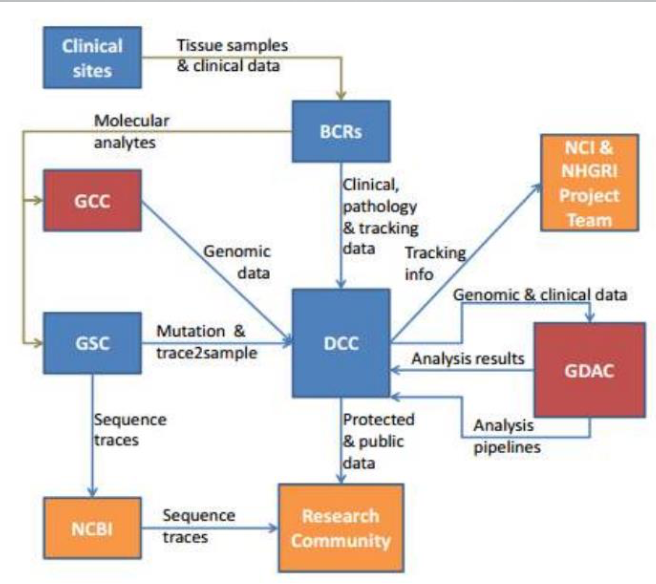
- TCGA 基因组分析中心(GCC)比对肿瘤和正常组织,寻找异常的基因重组现象。
- 高通量测序中心(GSC)分析与各癌症或者亚型相关的基因突变、扩增或者缺失。
- 资料分析中心(GDAC)进行资料的整理、汇总、并提供图表报告给全体研究团队。
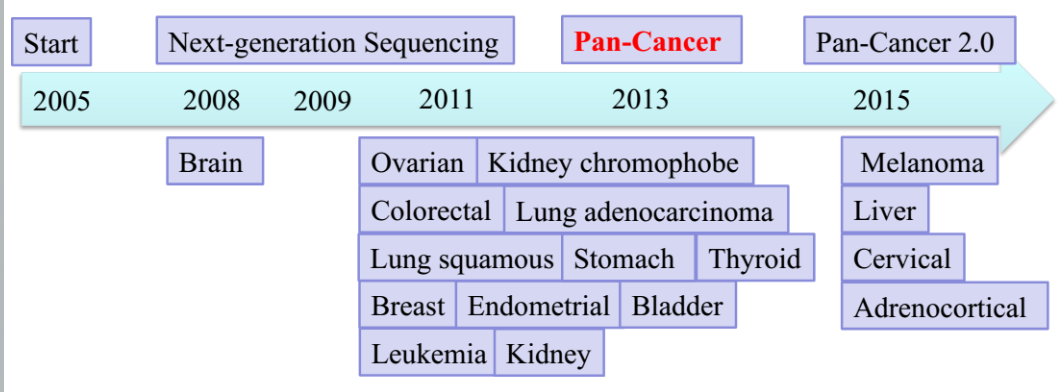
1.2 TCGA history
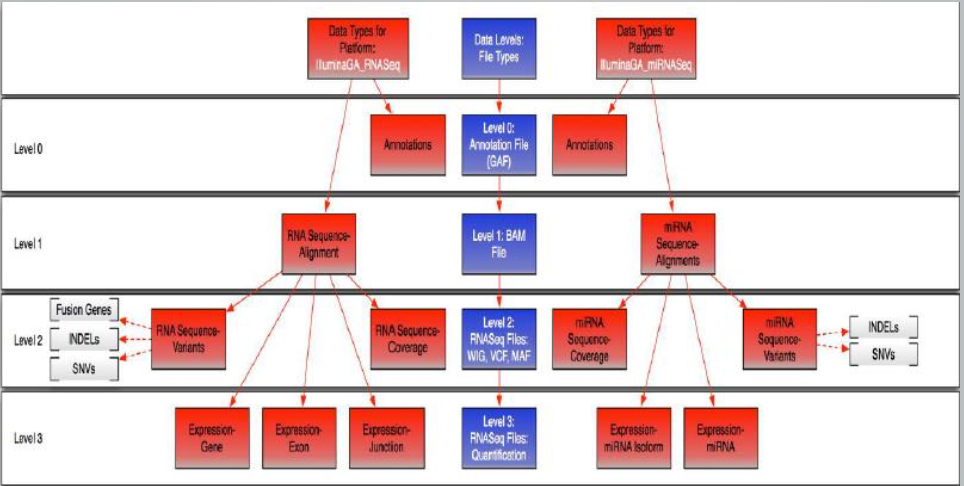
1.3 TCGA数据资源与类型
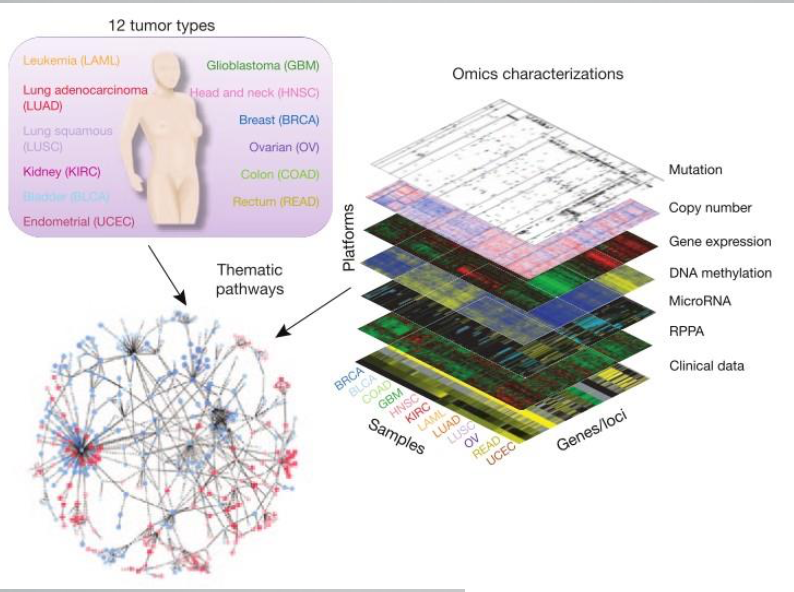
数据类型包括:Clinical Data,Images,Microsatellite Instability (MSI),DNA Sequencing,miRNA Sequencing, Protein Expression,mRNA Sequencing,Total RNA Sequencing ,Array-based Expression ,DNA Methylation, Copy Number
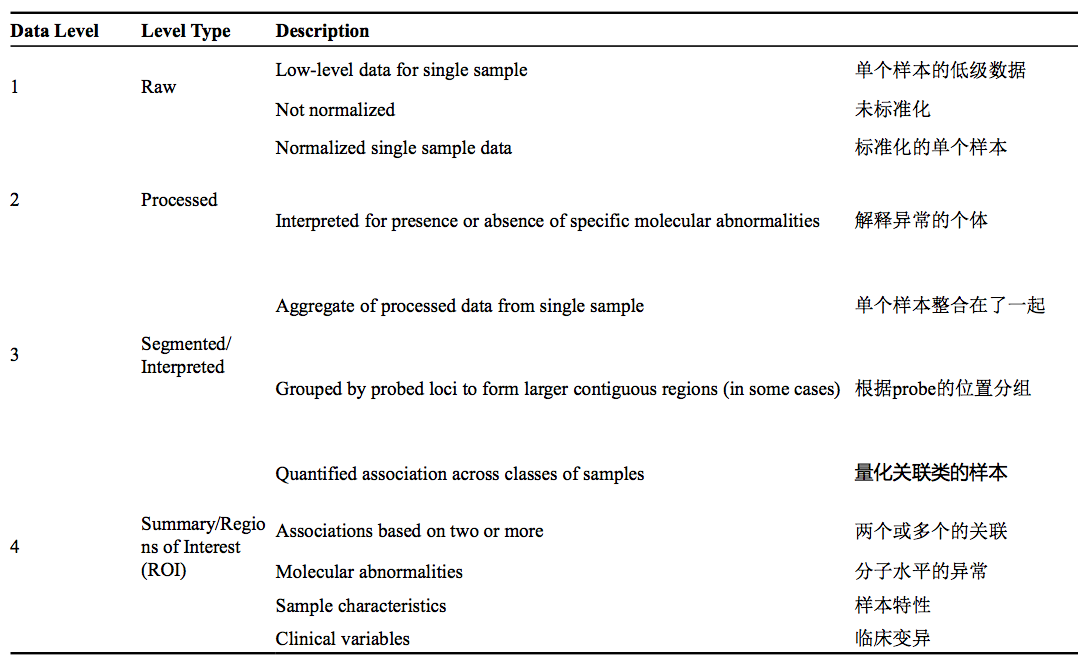
更详尽的关于数据类型和数据等级,参见:https://cancergenome.nih.gov/abouttcga/aboutdata/datalevelstypes
数据概况:
- 癌症种类丰富,样本量大
- 34 kinds of cancer 104+ samples
- Whole-exome sequencing
- 10% whole-genome sequencing (30-50 x)
- 20% low-pass whole genome sequencing (6-7X)
- RNA-seq (mRNA + miRNA)
- SNP arrays
- Methylation (Illmina arrays)
- Proteomics (RPPA)
1.4 Platform Design
- Applied Biosystems Sequence data
- Agilent 244K Custom Gene Expression
- Agilent SurePrint G3 Human CGH Microarray Kit
- Affymetrix Genome-Wide Human SNP Array
- Agilent 8 x 15K Human miRNA-specific microarray
- Illumina Genome Analyzer DNA Sequencing 等等
更多阅读:https://cancergenome.nih.gov/abouttcga/aboutdata/platformdesign
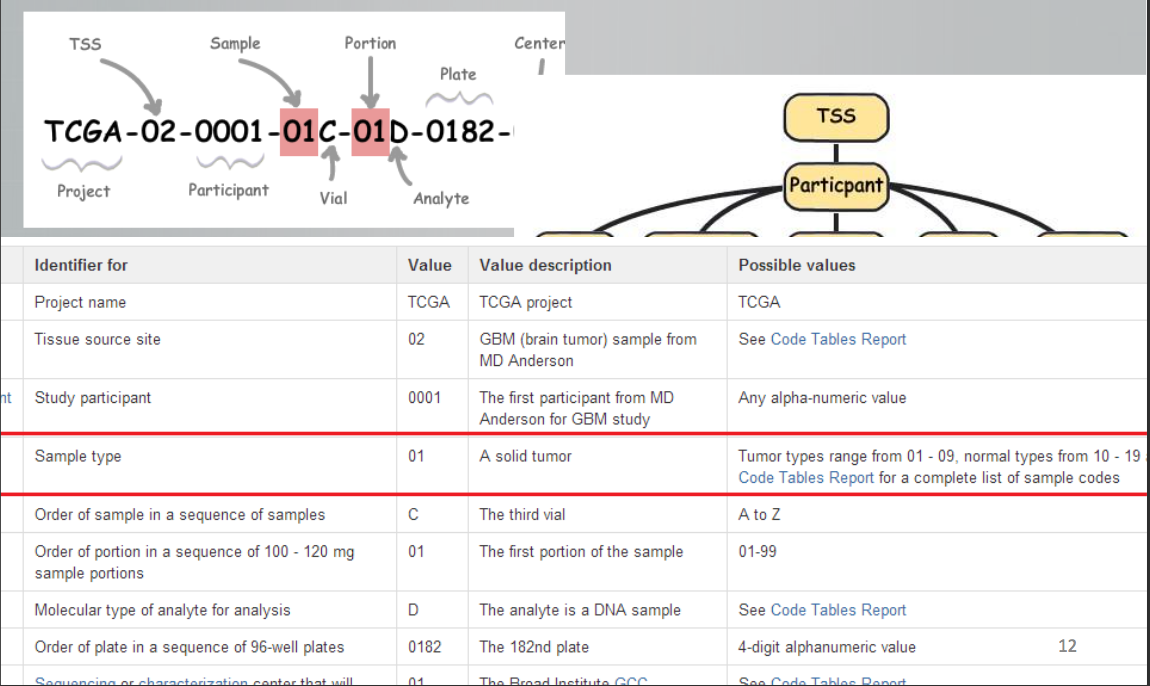
1.5 TCGA barcode
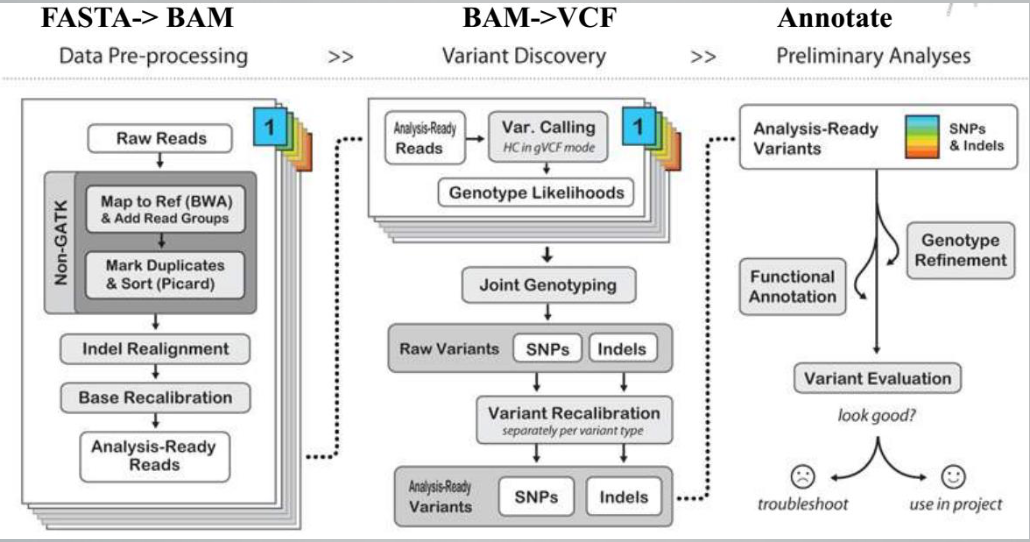
1.6 Exom-Seq
TCGA RNA-Seq
Gene Expression Platform

1.7 癌症样本组织处理
-
癌症病人自愿捐赠肿瘤组织及正常组织样本,由人类癌症生物标本核心资源库承担癌症组织标本和正常组织标本的采集、处理和分配工作。
-
组织样本经过严格标准处理(处理标准根据不同后续分析类型而异,具体标准请参见),确保质量可以用于进一步分析及测序,并由相关中心采用高通量测序技术进行基因和基因组排序。
-
获得的临床资料中,可以识别病人身份的信息去掉。
TCGA 整合分析

二、数据下载
虽然在TCGA中直接下载数据的方法较为繁琐,但是有多个网站提供TCGA数据(包括表达和临床等)完善的整理:GDAC, Cancer Browser和cBioportal是其中整理最为完整和可靠的。GDAC由美国MIT和Harvard共建的Broadinstitute运行,UCSC运行着Cancer Browser 和Xena, cBioportal由MemorialSloan-Kettering Cancer Cente建立,提供较为完善的TCGA数据为基础的各类信息检索服务。

下载的数据分为两个权限:
1.公开的数据
这部分数据不涉及个人信息,下载这部分数据不需要用户认证,包括的数据
- De-identified clinical and demographic data
- Gene expression data
- Copy number alterations in regions of the genome
- Epigenetic data
- Summaries of data compiled across individuals
- Anonymized single amplicon DNA sequence data
2.受控的数据
因为这部分信息设计到个人信息,所有需要用户申请,包括的数据:
- Primary sequence data (BAM and FASTQ files)
- SNP6 array level 1 and level 2 data
- Exon array level 1 and level 2 data
- VCFs
- Certain information in MAFs
3.资料分享
- 资料综合中心(DCC)集中处理各个团队产生的资料,定期公开于网络上供全世界研究人员利用。
- 提供公开的资料下载网站入口以方便进行资料搜索和下载
下载途径
1.GDC
自2016年7月15日起,TCGA(The Cancer Genomic Atlas) DATA PORTAL不再提供数据服务,所有数据将转入GDC(Genomic Data Commons) DATA PORTAL。 GDC网站下载TCGA数据,图形界面,操作简单。
GDC提供两种数据下载方式:
(1)对于少量数据,在购物车内点击download,选择cart可以直接下载购物车内的数据
(2)对于大量数据,从购物车中直接下载易出现错误。我们可以点击download下的manifest,然后利用GDC Transfer Tool (gdc-client),在Terminal内输入如下命令进行批量下载: ./gdc-client download -m manifest_xxx.txt
Workflow Type 一般选择 ** HTSeq - FPKM-UQ
(需要授权认证)
2.gdac和firehose
网站是:https://gdac.broadinstitute.org/
客户端工具是firehose_get ,https://confluence.broadinstitute.org/display/GDAC/Download
这里的数据也来源于 portal.gdc.cancer.gov,经过了简单的合并,将每种癌症相同类型的数据合并到了一个文件中(例如443个胃癌样本的RNA表达量数据都合并到了一个文件中,非常适合用R进行后续的分析)
更多阅读:
http://www.biotrainee.com/thread-822-1-1.html
http://www.biotrainee.com/thread-822-1-2.html
3. cgdsR和cbioportal
cbioportal地址是:http://www.cbioportal.org/
他们给网站做了一个R包的API为cgdsR,整合和简化了包括TCGA,ICGC以及GEO等多个癌症基因组数据库的内容,提供友好可视化的界面,可供下载。
主要展示基因的somatic 突变谱,拷贝数变化,mRNA&miRNA表达量变化,DNA甲基化以及蛋白质表达的情况,并结合患者的临床资料,展示了KM生存曲线。
更多阅读:http://www.biotrainee.com/thread-824-1-3.html
4. Synapse
Synapse是需要注册的,但是是免费注册的,很简单,用谷歌账户注册即可。
https://www.synapse.org/#!Synapse:syn300013
这里面存放的就是一系列TCGA大文章的数据,一些人整理好的,所以非常方便的可以使用! 比如,我们可以获取 Lung Squamous Cell Carcinoma的生存分析数据
https://www.synapse.org/#!Synapse:syn1446127/version/3
三、常用分析工具
1. cBioPortal(cBio Cancer Genomics Portal)
是一个基于TCGA数据库,进行可视化分析的网页。
a. 首先进入这个网页(http://cbioportal.org),然后可以看到下面这个界面,首先选择你想要分析的数据库和具体的数据
b. 接着勾选你要分析的数据到底都是啥,主要可以分析的是MUT(Mutation,突变),CNA(Copy Number Alterations,拷贝数变化),EXP(mRNA Expression,mRNA表达)和PORT/RPPA(Protein/ phosphoprotein level,蛋白表达或磷酸化变化)。但要注意的是,并不是所有数据都具备这四个选项,大多数只有MUT和CNA这两组数据,有些具有EXP数据和PORT数据。接着,要选择你要研究的基因,有一个下拉菜单可以给你参考,比如会有类似信号通路上的明星分子集合这类,你可以按照需要选择。当然,也可以自己输入基因名
c. 确认后就可以进入结果页面了,主要是显示样本中较为直观的变化,比如突变、缺失、RNA表达、磷酸化变化等等。
2. 用TANRIC来探索癌症中lncRNA功能
更多阅读:http://www.biotrainee.com/thread-999-1-2.html
3. TCGA2BED-可以从TCGA数据库提取数据成bed格式
官网:http://bioinformatics.mdanderson.org/main/TANRIC:Overview
更多阅读:http://www.biotrainee.com/thread-1056-1-1.html
4. TCGA可视化数据库GEPIA
参见 GEPIA 的博文
5. TCGA生存分析oncolnc
官网:http://www.oncolnc.org/
这是一个整合了TCGA的各种RNA数据和患者临床数据,提供生存分析的网站,灰常简单好用。
6. 基于TCGA的蛋白芯片分析神器TCPA
官网:http://www.tcpaportal.org/tcpa/
更多阅读:http://www.biotrainee.com/thread-1293-1-1.html
7. UCSC的cancer genome browser探索TCGA的level3数据
可以对任何癌种,根据任何临床指标进行分sub-group之后进行任何形式的生存分析,比较分析,还有相关分析。
更多阅读: http://www.biotrainee.com/thread-1086-1-1.html
8. Immunophenogram
网站是:https://tcia.at/home
TCGA的数据挖掘大文章类型,从细胞群里里面区分各种免疫细胞
9. 基于TCGA的甲基化神器mexpress
官网:http://mexpress.be/
整合了TCGA中的DNA甲基化,表达量及临床数据,主要用来探索甲基化,基因表达和临床表型之间的关联
10. oncomine
Oncomine是目前最大的癌症基因芯片数据库 更多阅读:http://www.biotrainee.com/thread-1242-1-1.html
更多工具
- UCSC CGHub (https://cghub.ucsc.edu/)
- Broad firehose (https://confluence.broadinstitute.org/display/CGATools/Firehose)
- UCSC Cancer Browser Portal (https://genome-cancer.ucsc.edu/)
- TCGA pan-cancer analysis (http://www.nature.com/tcga/)
讨论
用TCGA数据的时候,需要考虑肿瘤亚型
参考资料
- http://www.biotrainee.com/thread-1080-1-1.html
- http://www.biotrainee.com/thread-306-1-1.html
- http://www.biotrainee.com/thread-307-1-1.html
- http://www.biotrainee.com/thread-827-1-1.html
- http://www.biotrainee.com/thread-1290-1-1.html
- http://paper.dxy.cn/article/511878
- https://cancergenome.nih.gov/abouttcga/overview
- 上海宇道生物技术有限公司 《实用肿瘤生物信息学讲座》
