【6.2.5】DeepDDG-- 使用神经网络预测蛋白质点突变的稳定性变化
网页地址: http://protein.org.cn/ddg.html
- 准确预测由于突变引起的蛋白质稳定性变化对于蛋白质工程和理解蛋白质错义突变的功能后果很重要。
- 我们开发了 DeepDDG,一种基于神经网络的方法,用于预测由于点突变引起的蛋白质稳定性变化。
- 神经网络在超过 5700 个手动策划的实验数据点上进行了训练,并且能够在三个独立的测试集上获得 0.48-0.56 的 Pearson 相关系数,优于其他 11 种方法。
- 输入特征的详细分析表明,突变残基的溶剂可及表面积是最重要的特征,这表明隐藏的疏水区域是蛋白质稳定性的主要决定因素。
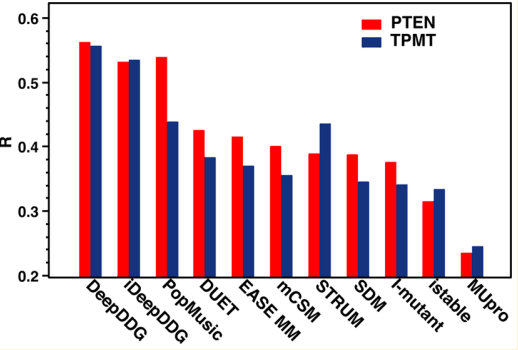
各种方法的比较
网络训练的方法:

我们接下来调整了 SRP (shared residue pair) 网络来预测突变的ΔΔ G值(图2)。突变的残基及其N 个最近的相邻残基中的每一个(基于 C α -C α距离)形成一个残基对,这是 SRP 网络的基本输入。相邻残基数N设置为 15,这在我们之前的研究中证明了蛋白质设计的最佳性能。SRP 网络的权重在所有残差对之间共享,类似于卷积网络中的权重。一对残基的特征包括:
- 几何特征,例如骨架二面体、溶剂可及表面积、二级结构、氢键数量、突变残基与相邻残基之间的距离和方向;
- 序列特征,例如来自 PSI-BLAST 搜索的 PSSM 值、成对适应度得分 (PFS;参见方法)) 来自多序列比对,以及基于我们的蛋白质设计神经网络的目标残基的野生型和突变氨基酸的概率;
- 目标氨基酸类型和邻近残基的一般特征。

参考资料
这里是一个广告位,,感兴趣的都可以发邮件聊聊:tiehan@sina.cn
![]() 个人公众号,比较懒,很少更新,可以在上面提问题,如果回复不及时,可发邮件给我: tiehan@sina.cn
个人公众号,比较懒,很少更新,可以在上面提问题,如果回复不及时,可发邮件给我: tiehan@sina.cn

