【2.5.4】Parker亲水性预测
预测网址:http://tools.iedb.org/bcell/
In this method, hydrophilic scale based on peptide retention times during high-performance liquid chromatography (HPLC) on a reversed-phase column was constructed. A window of seven residues was used for analyzing epitope region. The corresponding value of the scale was introduced for each of the seven residues and the arithmetical mean of the seven residue value was assigned to the fourth, (i+3), residue in the segment.
Scale:
A C D E F G H I K L M N P Q R S T V W Y
2.1 1.4 10.0 7.8 -9.2 5.7 2.1 -8.0 5.7 -9.2 -4.2 7.0 2.1 6.0 4.2 6.5 5.2 -3.7 -10.0 -1.9
提出了一套新的亲水性高效液相色谱(HPLC)参数。这些参数来自20种模型合成肽Ac- Gly-X-X-(Leu)3-(Lys)2-amwihde的保留时间,其中X被蛋白质中发现的20个氨基酸取代。由于亲水性参数已在算法中广泛使用,以预测哪些氨基酸残基具有抗原性,因此我们以与其他九组参数相同的规模比较了我们的新型亲水性HPLC参数集生成的图谱。通常,发现在这项研究中获得的HPLC参数与抗原性最相关。另外,显示出用于预测抗原性的三个最佳参数的组合进一步改善了预测。然后将这些预测的表面位点,亲水,可及或可移动的区域与免疫学研究中已知的抗原位点和几种蛋白质的X射线晶体学数据确定的可及位点相关联。
一、前言
现在认为,当将蛋白质表面的肽片段用作免疫原或将不同物种用作免疫宿主时,可以认为蛋白质的整个表面具有抗原性(Green等,1982; Benjamin等, 1984)。 抗原性位点定义为与天然蛋白,天然蛋白片段,合成肽产生的抗体结合的天然蛋白残基。 按照定义,由于抗原位点是抗体识别的位点,因此这些位点很可能可访问或位于蛋白质表面,并且这些区域可能比内部区域更具移动性。 由于这些位点在表面上,因此它们可能是亲水的。 实际上,亲水性和可及性的算法已被用于预测抗原性。
我们已经通过实验确定了一个新的亲水性标度,该标度源自高效液相色谱(HPLC)中每个氨基酸侧链对模型合成肽Ac- Gly-X-X-(Leu)3-(Lys)2-amwihde 的保留时间的贡献,,其中X被蛋白质中的20个氨基酸取代。 这组新的亲水性参数用于改进的Hopp and Woods(1978)算法中,以预测蛋白质的哪些区域在表面上。 发现这些预测的表面位点与肌红蛋白,溶菌酶,细胞色素c和流感血凝素的已知抗原位点很好地相关。 肌红蛋白,溶菌酶,细胞色素c,牛胰蛋白酶的出色X射线数据,
二、实验
。。。。
三、结果与讨论
3.1 Retention Coefficients
….
3.2 亲水性量表 Hydrophilicity Scale
肽保留时间(来自图1)为蛋白质中发现的所有20个氨基酸残基提供了一组HPLC保留系数(表I)。 表II中所示的HPLC亲水性标度将表I中具有最大保留系数的氨基酸残基的疏水值为-10,而将显示出最小保留系数的氨基酸残基的亲水值为+ 10。 其余氨基酸残基按比例缩放。 在所有先前报道的亲水性等级中,没有考虑到N端游离氨基或C端羧基的贡献。 使用表I中的数据,我们确定这些值分别为9.7和14.3。 我们以类似的方式缩放了本文中使用的所有其他参数,以便可以直接对其进行比较(表I和表III)。

3.3 与其他参数的相关性
这些新的HPLC参数与其他九个参数的比较如表I和表III所示。计算得出,它们与HPLC参数具有以下相关性:可及性0.58;,本体疏水性为0.84; Hopp和Woods疏水性为0.82; global 0.82; Bull and Breese的疏水性为0.89; Faushere和Pliska x参数为0.93; Kyte和Doolittle hydropathy,0.72; 和水合电位为0.59。 尽管与x参数的相关性最高,但是在亲水性残基精氨酸和赖氨酸以及非极性残基脯氨酸,甘氨酸和半胱氨酸上观察到较大的差异。
3.4 表面轮廓 Surface Profile
通过将七个残基片段的每个残基的参数求和并将该总和分配给第四残基来确定蛋白质的表面轮廓,通过将片段从N-端移一个残基来重复此过程。 使用HPLC,可及性,本体疏水性,疏水性,总体,x或亲水性参数将这些值与残基数作图,可提供表面轮廓。此过程的唯一例外是B值的例外。 Karplus和Schultz(1985)在论文中描述了使用B值的算法。为了客观地解释所有这些特征,我们使用以下任意规则:
- 平均表面亲水性定义为使用特定参数集的蛋白质特征值的平均值。
- 任何具有特征值的残基大于平均表面亲水性值25%以上的区域被定义为表面位置,计算25%的值是该图的最大值与平均表面亲水性值之差的25%
- 在确定对于具有游离N末端氨基或C末端羧基的蛋白质,将9.7(氨基)和14.3(羧基)的实验值直接加到N或C末端氨基酸残基的值上表I列出了Nα-乙酰基和Cα-酰胺基团的系数。
对于本研究中测定的HPLC参数以外的所有参数,均未考虑游离的N或C端残基。
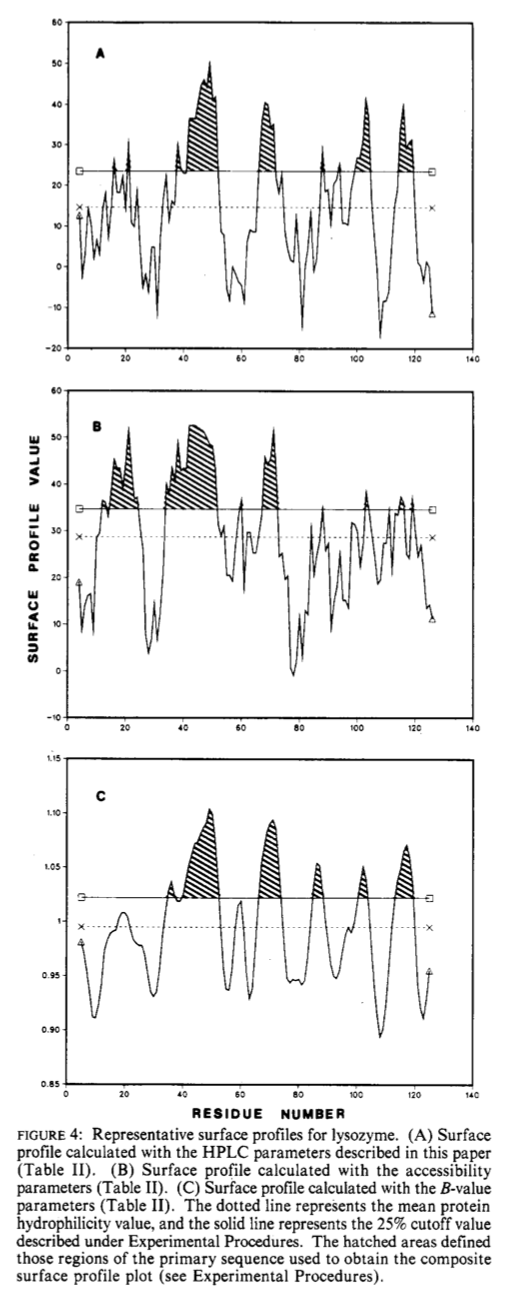
使用表II和表III中列出的所有参数,计算了四种蛋白质的肌红蛋白,溶菌酶,细胞色素c和流感血凝素的表面轮廓。这些轮廓的示例使用HPLC,Janin可及性以及Karplus和Schulz灵活性参数, 图4A-C显示了溶菌酶。 这三个配置文件的一般模式和所研究的所有参数的模式都是相似的。 但是,所有最大轮廓值的强度随每个参数集而变化。 为了解释这些特性并将其与抗原性相关联,检查了平均表面亲水性以上25%,50%和75%的残基(请参见实验程序)。
抗原性位点被定义为蛋白质上被针对该蛋白质或肽片段的抗体识别的那些位点。肌红蛋白,溶菌酶,细胞色素c(Atassi,1984; Benjamin等,1984)和流感(Wiley等,1981; Green等,1982; Muller等,1982)的抗原位点已经汇报过。通常,对于所研究的四种蛋白质,HPLC参数比表II和表III中列出的其他参数集预测了更多的这些已知抗原位点。很显然,没有一个单参数集能够预测所有抗原位点。这在溶菌酶的曲线图中得到了说明(见图4A-C)。在图4B中,通过Janin参数预测了包括残基15-22的强抗原位点。通过使用HPLC参数(图4A)可预测同一区域的抗原位点非常弱,而Karplus和Schulz参数(图4C)未显示出高于25%临界值的值。相反,HPLC亲水性参数以及Karplus和Schulz的HPLC亲水性参数都预测残基98-102和115-120的强抗原性位点,而使用Janin参数预测在相同区域中非常弱的抗原性位点。
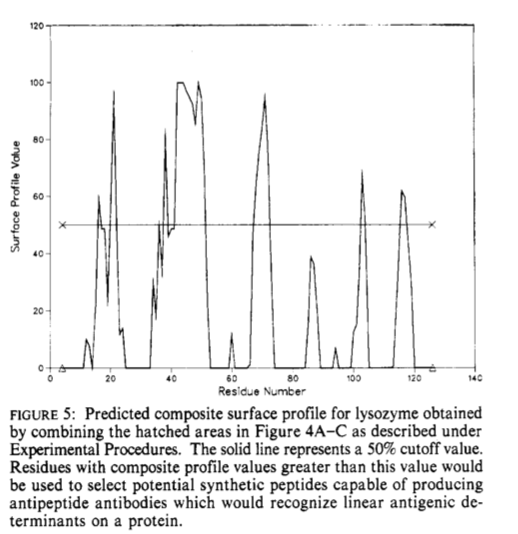
3.5 复合表面轮廓 Composite Surface Profile
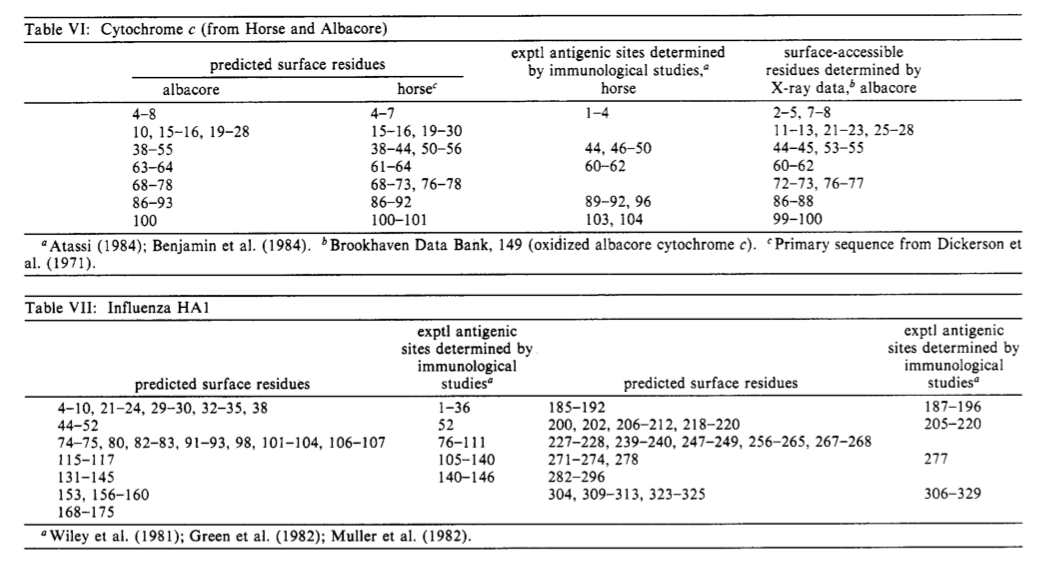
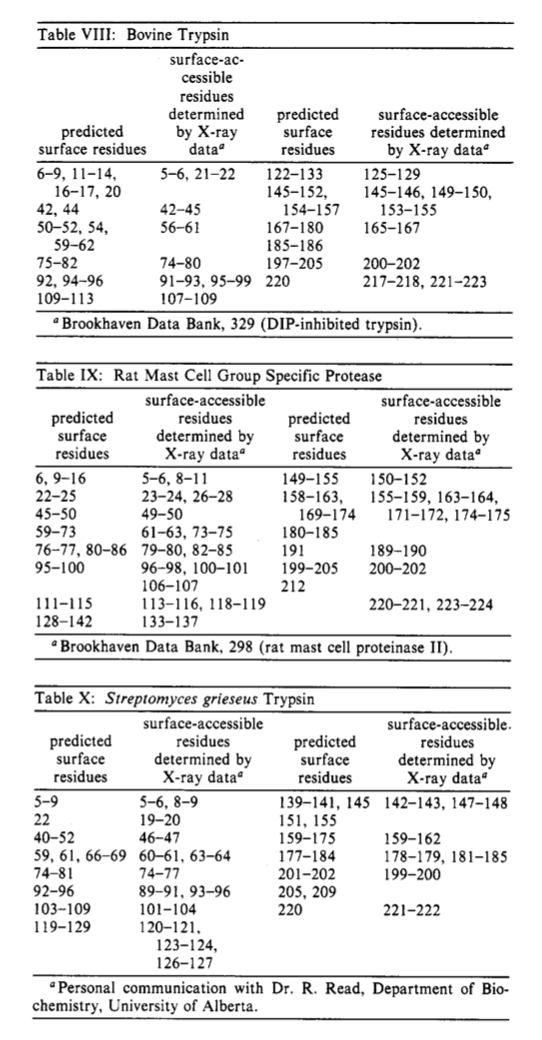
研究了各种参数的组合,以改善对抗原位点的预测。选择三个参数,分别在预测的抗原位点提供最高分。这些是HPLC亲水性(本研究),Janin可及性以及Karplus和Schultz柔韧性参数。使用HPLC(本文),可及性(Janin)和B值参数(Karplus和Schulz)绘制了三个表面轮廓图,以生成复合表面轮廓。将每个轮廓(图4A-C中的阴影区域)的表面位置(在“表面轮廓”下的规则2中定义)从0缩放为100,其中每个图中的最大表面位置值设置为等于100,且25%表面残位值等于0。然后将每个比例绘制的图叠加在另一个图上,并使用每个残基的最大值给出复合轮廓值。该程序可从艾e Alberta Peptide In- stitute (Biochemistry Department, University of Alberta)的IBM-PC或APPLE-MacIntosh计算机的软盘上获得。这样的复合图的一个例子在图5中显示了溶菌酶。然后绘制肌红蛋白,溶菌酶,细胞色素c(马和长鳍金枪鱼序列),流感A A1,牛胰蛋白酶,大鼠肥大细胞蛋白酶和灰链霉菌胰蛋白酶的复合材料表面轮廓。这些预测的表面位点在表IV-X中给出。检查这些表可发现,肌红蛋白和细胞色素c和流感的所有抗原性位点均被预测为表面位点。仅N和C末端抗原位点没有被预测溶菌酶。溶菌酶抗体复合物的首次X射线衍射研究(Amit等人,1985)已经证实了溶菌酶的几个预测表面位点。除了已知的抗原位点,仅预测有一个额外的表面位点(119-131)存在肌红蛋白。预测了细胞色素c的两个表面位点(15-30和68-78),而不是已知的抗原位点。预计还会有另外四个表面位点(153-1 59、168-175、227-264和282-299)。有趣的是,这些位点位于野生型流感中发现了几个突变的区域(Wiley等,1981)。
3.6 X射线数据中的表面可访问残基 Surface-Accessible Residues from X-ray Data
还发现这些预测的表面位点与X射线晶体学确定的表面可及残基有很好的相关性。 通过使用Lee和Richards(1971)的方法,使用1.4 A的探针半径计算所有表面可接近的残基。两个或更多个连续的,可及面积大于50 A2的连续残基定义为在蛋白质表面上。 以下蛋白质的坐标来自Brookhaven蛋白质数据库(1985年4月):肌球蛋白(268),溶菌酶(253),细胞色素c核(149),牛胰蛋白酶(329),大鼠肥大细胞蛋白酶(GSPRAT,298) ,灰链霉菌胰蛋白酶(SGT,人员通讯)。 这些蛋白质的可及残基示于表IV-VI和VIII-X。 应当注意,通过X射线晶体学数据显示,与免疫学数据不相关的肌红蛋白和细胞色素c的预测位点在蛋白质表面上。

四、总结
-
我们提出了一套从合成肽的HPLC衍生的新的亲水性参数。我们 还对其他9个参数进行了缩放,因此可以直接与这些新的HPLC参数进行比较。
-
由于没有一个单一的参数能够预测所有抗原位点,因此我们将HPLC,可及性和柔韧性参数结合起来以产生复合表面轮廓。该复合图谱显示与几种蛋白质的已知抗原和X射线确定的可及位点密切相关。以前,亲水性算法已被广泛用于从能够产生识别该蛋白质上线性抗原决定簇的抗肽抗体的蛋白质序列中选择潜在的合成肽。通过将这种肽与蛋白质载体偶联(Parker&Hodges,1985),并用这些肽结合物免疫动物宿主,可以制备对合成肽具有特异性的抗体。
-
我们建议首先选择表面轮廓大于50%的值的位置(请参见图5),然后选择那些具有较低截止值的位置。
参考资料
- Reference: Parker JM, Guo D, Hodges RS. New hydrophilicity scale derived from high-performance liquid chromatography peptide retention data: correlation of predicted surface residues with antigenicity and X-ray-derived accessible sites. Biochemistry. 1986 Sep 23; 25(19):5425-32.
