【6.2.3】设计热稳定蛋白--fireprot
fireprot从原理上讲,应该跟pross如出一辙,基于序列对结构和能量来突变。无非就是阈值上,工具整合上有一点点区别。
人们一直关注增加蛋白质的稳定性,以提高其在众多生物医学和生物技术应用中的可用性。最近开发了许多用于预测突变对蛋白质稳定性影响的计算机工具。然而,通常只使用现有工具预测对蛋白质稳定性影响很小的单点突变,然后必须进行费力的蛋白质表达,纯化和表征。在这里,我们介绍FireProt,一种用于自动设计多点热稳定突变蛋白的网络服务器,它在计算核心中结合了结构和进化信息。 FireProt利用十六种工具和三种蛋白质工程策略来制作可靠的蛋白质设计。该服务器配有交互式,易于使用的界面,允许用户直接分析和选择性地修改设计的热稳定突变体。 FireProt可以在http://loschmidt.chemi.muni.cz/fireprot免费获得。
一、前言
蛋白质广泛用于许多生物医学和生物技术应用中。 然而,天然存在的蛋白质通常不能承受恶劣的工业环境,因为它们大多进化为在温和条件下发挥作用。 蛋白质工程通过改善各种蛋白质特性(如稳定性,活性或对映选择性)来彻底改变天然蛋白质在不同工业应用中的利用,从而超越其天然限制。 蛋白质稳定性通常与其表达产率,半衰期,血清存活时间和在变性剂存在下的表现强烈相关。 因此,稳定性是蛋白质在生物技术过程中适用性的关键决定因素之一。
在理想情况下,饱和诱变(saturation mutagenesis)将用于评估工程蛋白每个位置的每个可能的突变(6)。然而,这样的搜索空间将是巨大的,并且实验评估可以延迟真正热稳定蛋白质的设计数月甚至数年。因此,需要对蛋白质稳定性进行有效和精确的预测计算。为了实现这一目标,最近开发了许多计算机工具。其中一些工具,如EASE-MM(7),I-Mutant(8)或mCSM(9),都是基于机器学习技术。其他人正在使用所谓的能量函数。这些程序可以进一步分为两组:
- 第一组利用物理有效能量函数来模拟原子之间的基本力,并由Rosetta(10)和Eris(11)等程序表示。
- 第二组基于统计学潜力,其能量来源于实验表征的蛋白质突变体数据集中报道的残基或原子接触的频率,例如, Pop-MuSiC(12)和FoldX(13)。
然而,由于突变的潜在拮抗作用,通常只能在计算机上预测单点突变,并且必须遵循费力且昂贵的蛋白质表达,纯化和表征。单点突变通常以度为单位提高目标蛋白的解链温度(3,14)。通过构建多点突变体可以实现更高程度的稳定化(15)。我们最近开发了FireProt(16),结合了基于能量和进化的方法,可靠地设计稳定的多点突变体。该协议包括几个前面的过滤器,通过省略潜在的有害突变来加速计算。 FireProt目前仅以独立格式提供,需要丰富的生物信息学经验才能执行所有必要的工作步骤。目前,我们只知道一个服务器用于设计稳定的多点突变体–PROSS(17),在其计算核心中利用Rosetta建模和系统发育序列信息。
在这里,我们提供了一个Web版本的FireProt,用于热稳定蛋白的自动设计。 FireProt集成了16种计算工具,并利用了序列和结构信息。 FireProt Web服务器为用户提供了热稳定蛋白,由三种不同的策略构建:
- 基于进化的方法,利用回归共识分析( back-to-consensus analysis )
- 基于能量的方法,评估突变时自由能的变化
- 基于进化和基于能量的方法的组合。
我们认为,采用这种综合方法是非常重要的,因为系统发育分析能够识别由熵稳定的突变,这不能通过力场计算来预测。该服务器允许用户将优选的突变包含在热稳定蛋白中,以产生相应的基因合成结构和序列。与之前发布的FireProt流程(16)相比,用户需要最少的努力和生物信息学知识来计算和分析结果。此外,所有输入参数和计算协议都经过优化,以最大限度地减少高时间需求程序。该服务器配有图形界面,允许用户直接分析感兴趣的蛋白质并设计多点突变体。
二、材料和方法
FireProt策略的基本工作概述如图1所示。为了设计高度可靠的热稳定多点突变体,用户定义的蛋白质使用多种预测工具和数据库进行注释(第1阶段)。 掌握了这些知识,应用基于能量和进化的方法来组合可能稳定的单点突变列表(第2阶段)。 最后,以组合的方式产生三个多点突变体,同时消除了突变的潜在拮抗作用(第3阶段)。
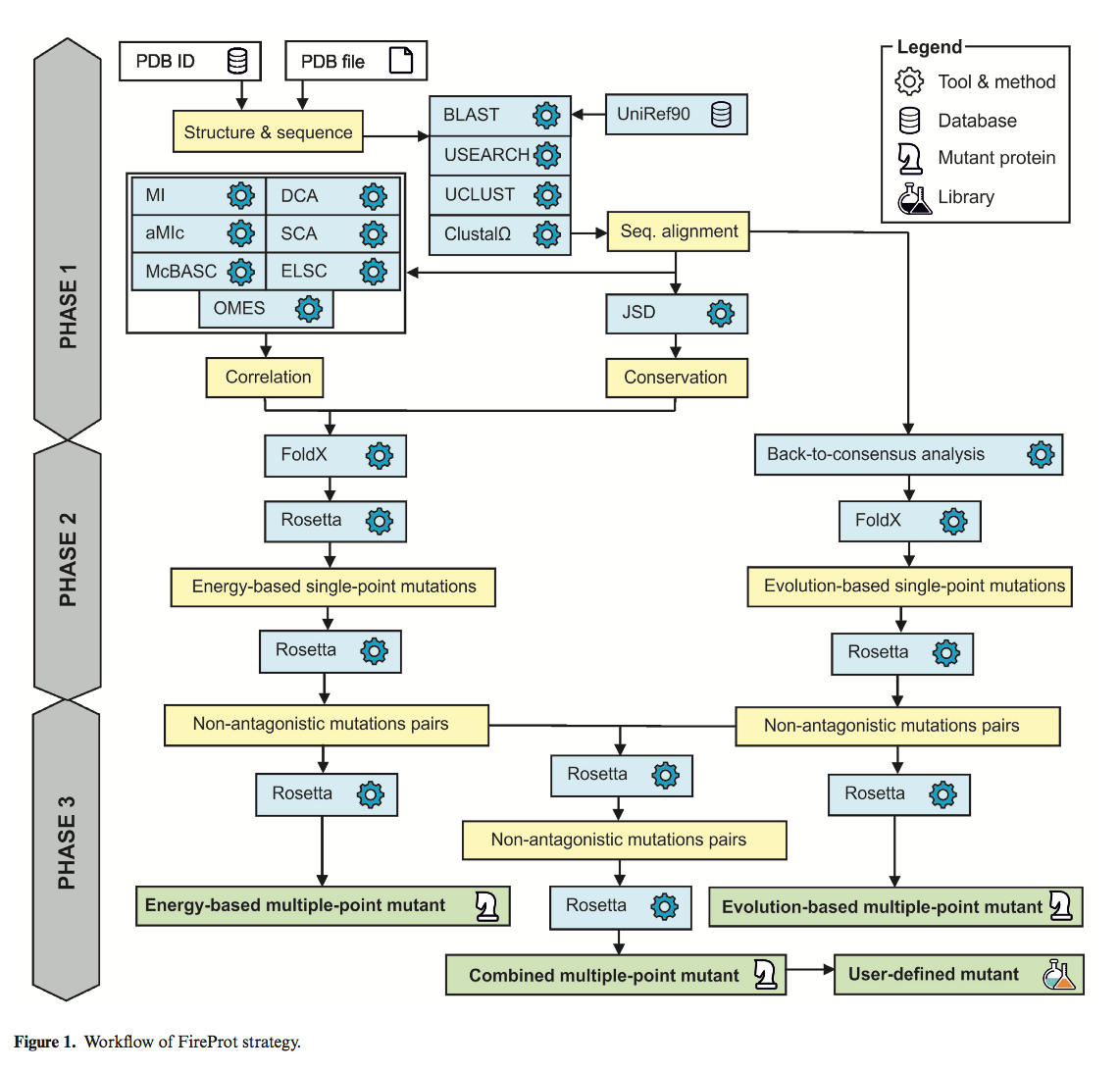
第1阶段:蛋白质的注释
- 最初,要求用户通过提供其PDB ID或通过上传用户定义的PDB文件来指定蛋白质结构。
- 然后由MakeMultimer工具( http://watcut.uwaterloo.ca/tools/makemultimer/ )自动生成目标蛋白的生物组装。
- 通过使用靶蛋白质序列作为输入查询对UniRef90数据库(19)进行BLAST搜索(18)获得序列同源物。
- 然后使用USE-ARCH(20)将鉴定的同源物与查询蛋白质对齐,而与查询的同一性低于或高于用户定义的阈值(默认值:30和90%)的序列从列表中排除。
- 使用UCLUST(20)对剩余序列进行聚类,具有90%的同一性阈值以去除紧密同源物。
- 群集代表基于BLAST查询覆盖率进行排序,默认情况下,它们中的前200个用于创建与Clustal Omega工具(21)的多序列比对。
- 多序列比对用于:(i)基于Jensen-Shannon熵估计蛋白质中每个残基位置的保守系数(22); (ii)使用OMES(23),MI(24),aMIc(25),DCA(26),SCA(27),ELSC(28),McBASC(29)和(iii)的共同决定确定相关位置。 iii)分析蛋白质中各个位置的氨基酸频率。
阶段2:单点突变的预测
根据最初的FireProt方案,可能通过两个独立的分支来识别潜在稳定的单点突变:一个依赖于估计突变时自由能的变化,另一个依赖于回归一致的方法(back-to-consensus approach)。
第一种基于能量的方法采用FoldX和Rosetta工具,这些工具在我们的测试数据集上表现最佳。前面的过滤器通过省略潜在的有害突变来加速计算。在识别单点突变本身之前,修改目标蛋白质结构并使其最小化。 FoldX协议用于在残基中丢失原子,因此使用Rosetta最小化模块最小化修补结构。保守和相关位置立即被排除在进一步分析之外。据观察,蛋白质的功能和结构限制通常会导致氨基酸残基的保守性。同样,相关残基通常有助于维持蛋白质功能,折叠或稳定性。因此,当前的FireProt策略认为在这些位置上进行的突变被认为是不安全的,即使有相关位置的更复杂处理的空间,这将在未来版本的FireProt服务器中进一步开发。
使用FoldX工具对剩余位置进行饱和突变。 将预测的△△G超过给定阈值(默认值:-1kcal / mol)的突变丢弃,并将剩余的发至Rosetta计算。 最后,Rosetta预测的突变强烈稳定(默认截止值:-1 kcal / mol)被标记为多点突变体设计的潜在候选者。 (这里的FoldX,看来就起一个粗筛的作用)
Rosetta分析的高时间要求是原始FireProt协议中最令人难以忍受的问题之一。 即使应用了100多个突变体,通常还需要进行精确但缓慢的Rosetta计算。 出于这个原因,我们用新组装的数据集评估了几个force fields和Rosetta协议,这些数据集包含来自ProTherm数据库(37)和HotMuSiC数据集(38)的1573个突变。 根据评估结果,选择时间要求和精度之间的最佳平衡。 使用Rosetta协议3,我们在计算速度方面实现了十倍以上的增长,同时保持了较高的预测精度。 有关数据集构建和协议评估的详细信息,请参阅补充说明1(补充表S1-S5)。
第二种方法基于从多序列比对获得的信息。 蛋白质序列每个位置中最常见的氨基酸通常对蛋白质稳定性产生不可忽略的影响(39-42)。 因此,FireProt实施多数和频率比方法来识别野生型氨基酸与最普遍的氨基酸不同的位置的突变。 默认情况下,单输出突变位于共有残基存在于所有分析序列的至少50%(多数方法,majority metho),或共有残基频率为40%且至少是比野生型氨基酸频繁5倍的位置(频率比法,frequency ratio method)。根据先前发布的HotSpot Wizard方法(43)选择这些阈值。 选择的突变由FoldX评估,稳定的突变被列为多点突变体工程的候选突变。( 哈哈,PROSS是根据PSSM来定义哪些残基被保留进行突变,跟fireprot有点区别)
阶段3:热稳定蛋白质的设计
总共有三种蛋白质设计由FireProt策略提供。 第一种设计仅包括基于能量的方法的突变,第二种设计包含基于进化的方法提出的突变,第三种设计包含两者的组合。 当然,由于个体突变之间可能存在拮抗作用,我们不能盲目地结合个体突变。
为了避免可能的冲突,FireProt策略试图通过利用Rosetta来最小化对抗效果。 在第一步中,对于基于能量和进化的方法,分别评估10Å范围内的所有单点突变对。 一旦获得所有残基对的自由能变化,FireProt就会根据其预测的稳定性开始将它们引入多点突变体,排除与已包含的突变相冲突的突变。 一旦没有突变或者分析对的稳定效果下降到低于定义阈值,算法就会停止。
完成前一步骤后,这次只考虑选择用于构建基于能量和进化的突变体的突变之间的对,重复该过程。 最后,使用Rosetta协议16对所有三种固体的结构进行建模。
三、Web服务器的描述
3.1 输入
Web服务器唯一需要的输入是感兴趣的蛋白质的三级结构,作为PDB ID或用户定义的PDB文件提供。然后,用户可以选择由MakeMultimer工具生成的预定生物单元,或者手动选择应进行计算的链。可以在基本模式或高级模式中配置计算。
在基本模式下,允许用户更改BLAST搜索和对齐构造的设置。高级模式扩展了与以下相关的可修改参数列表:
- 通过多数和频率比方法识别共识残基( consensus residues),
- FoldX和Rosetta预测工具使用的阈值
- 在相关位置的共识分析中使用的决策阈值。
高级模式允许专家用户根据研究的系统调整计算参数。但是,所提出的默认值已经过优化,可以为大多数系统提供可靠的结果,因此我们不建议在一般情况下对其进行更改。
3.2 结果
提交后,为每个作业分配一个唯一标识符以跟踪计算,“结果浏览器”会向用户通知Fire-Prot工作中各个步骤的状态(图2B)。 一旦完成工作,用户可以直接下载.zip存档中的结果,或者自己导航到“结果页面”以进行进一步的分析。 “结果页面”直观地组织成几个面板,如下所述。

蛋白质可视化 :野生型和突变体结构使用Jsmol小程序( http://wiki.jmol.org/index.php/JSmol )在Web浏览器中进行交互式可视化(图2D)。 用户可以在不同的蛋白质可视化样式之间切换,并突出显示蛋白质结构中的选定氨基酸。 包含在基于能量的突变体中的残基以橙色着色,基于进化的突变为蓝色,所有其他残基均为灰色。 用户选择的不属于任何重要部分的残留物用红色下划线标出。
突变概述。 “突变概述”面板组织成四个选项卡(图2A)。 前三个选项卡提供有关突变的信息,包括组合的,基于能量的和基于进化的突变体。 该复选框允许用户在Jsmol文件中可视化所选残差,可以在每一行中找到与给定计算方法相关的所有数据。 最后一个选项卡包含野生型结构中所有残基的列表。 当’wild-type’选项卡处于活动状态时,野生型结构在Jsmol applet中可视化而不是变异的结构,并允许用户通过最后一列中的’plus’图标将用户定义的突变引入多点突变体。
一般信息。 “FireProt协议设计”小组向用户提供有关目标蛋白质和FireProt策略构建的设计的一般信息,例如一些突变和估计的自由能变化(图2C)。
突变设计师。 “突变设计师”面板允许用户通过管理分为基于能量和进化的子集的突变来设计自己的多点突变体。 如果子集中的所有突变都具有指定的预测能量值,则假设简单加和,则立即估计吉布斯自由能的总变化。 用户还可以从设计的多点突变体生成氨基酸序列,该突变体将突变包括在基于能量和进化的子集中。 所有准备好的设计都可以在一个.zip存档中下载(图2E)。
四、实验验证
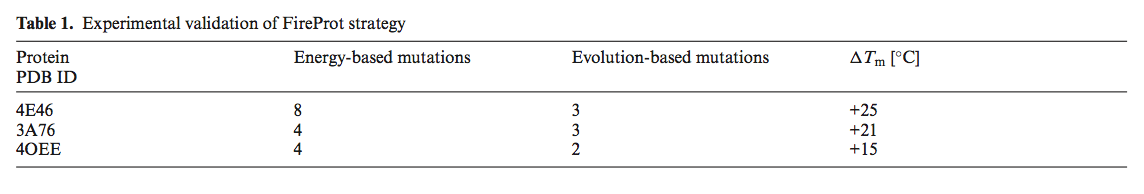
最初的FireProt策略用三种蛋白质(卤代烷脱卤素酶DhaA,PDB ID 4E46; - 六氯环己烷脱氢氯化酶LinA,PDB ID 3A76;和成纤维细胞生长因子2,PDB ID 4OEE)进行实验验证,并提供蛋白质的相应稳定性Tm = 25,21和15℃(表1)。 修改了原始协议,以便在合理的时间内实现全自动计算,同时保持较高的预测精度(补充表S6)。 使用该修饰方案预测8个多点突变体,使用FRESCO(44)的数据进行验证,并将鉴定的突变与另一个在线蛋白质稳定化工具PROSS(17)进行比较。 FireProt和PROSS显示出类似的预测能力,分别正确识别29和20个潜在的稳定位置(补充表S7)。
五、结论和展望
FireProt是一个Web服务器,为用户提供一站式解决方案,用于设计热稳定多点突变蛋白。 与独立的FireProt策略(16)相比,所有默认参数和计算协议都经过优化,可以提高计算速度,同时保持预测精度。 FireProt工作生产的设计经过实验验证,因此用户可以获得具有最低实验效率的高度可靠的热稳定蛋白。 该服务器由易于使用的图形界面补充,允许用户交互式分析作为能量或基于演化的方法的一部分选择的单个突变,以及在我们的强大策略之上设计自己的多点突变体的能力。
整个程序的自动化使得热稳定蛋白质的设计过程无需生物信息学的任何先前专业知识即可供用户使用,因为它无需选择,安装和评估工具,优化其参数并解释中间结果。 然而,FireProt战略的基于能量的方法取决于所提供的蛋白质结构的质量,因此在低分辨率结构或同源模型的情况下,预测准确性可能会受到影响。
在未来,我们计划实施新的策略,例如基于相关位置分析的设计,这将有助于构建最终组合突变体,消除高度灵活的区域和引入桥梁。 此外,我们计划为FireProt配备几个新的过滤器,例如 排除活性位点附近或参与寡聚化的氨基酸。
参考资料
- Musil, M., Stourac, J., Bendl, J., Brezovsky, J., Prokop, Z., Zendulka, J., … Damborsky, J. (2017). FireProt: web server for automated design of thermostable proteins. Nucleic Acids Research, 45(W1), W393–W399. https://doi.org/10.1093/nar/gkx285
