【4.3.1】来自大型开放实验室的RNA设计规则(EteRNABot、eternagame)
自组装 RNA 分子在整个生物学和生物工程中发挥着关键作用。为了加快 RNA 设计的进展,我们提出了 EteRNA,这是第一个通过高通量实验评分的互联网规模的公民科学“游戏”。一个由 37,000 名非专家组成的社区利用持续的远程实验室反馈来学习新的设计规则,这些规则大大提高了 RNA 结构设计的实验准确性。这些规则通过机器学习提炼成新的自动化算法 EteRNABot,在独立测试中也显着优于先前的算法。这些结果表明,在线社区可以进行大规模实验、假设生成和算法设计,从而在实证科学中取得实际进展。
摘要
自组装 RNA 分子为合理询问和控制生命系统提供了令人信服的底物。然而,不完善的计算机模型——即使是在二级结构水平——阻碍了合成时功能正常的新 RNA 的设计。在这里,我们针对此类经验问题提出了一种独特且可能通用的方法:大规模开放实验室。EteRNA 项目通过在线界面将 37,000 名爱好者连接到 RNA 设计难题。独特的是,EteRNA 参与者不仅可以操纵模拟分子,还可以控制用于高通量 RNA 合成和结构映射的远程实验管道。我们在此展示了 EteRNA 社区利用数十个循环的连续湿实验室反馈来学习解决自动化方法失败的体外 RNA 设计问题的策略。最重要的策略——包括几个以前未被认识到的负面设计规则——被机器学习提炼成一种算法,EteRNABot。在严格的 1 年测试阶段,EteRNA 社区和 EteRNABot 在十几个 RNA 二级结构设计测试中都显着优于先前的算法,包括为小分子传感器创建树枝状结构和支架。这些结果表明,在线社区可以进行大规模实验、假设生成和算法设计,从而在实证科学中取得实际进展。EteRNA 社区和 EteRNABot 在十几个 RNA 二级结构设计测试中都显着优于先前的算法,包括为小分子传感器创建树枝状结构和支架。这些结果表明,在线社区可以进行大规模实验、假设生成和算法设计,从而在实证科学中取得实际进展。EteRNA 社区和 EteRNABot 在十几个 RNA 二级结构设计测试中都显着优于先前的算法,包括为小分子传感器创建树枝状结构和支架。这些结果表明,在线社区可以进行大规模实验、假设生成和算法设计,从而在实证科学中取得实际进展。
一、前言
RNA折叠公民科学高通量实验众包 结构化 RNA 分子在从基因调控到病毒复制的生物过程中发挥着关键作用;这些 RNA 的表征、检测和再造是现代分子生物学和生物工程的主要目标(1 ⇓ ⇓ ⇓ ⇓ ⇓ – 7)。近年来,出现了优雅的 RNA 折叠模型,可以准确捕获环和简单螺旋的二级结构形成(8 ⇓ ⇓ ⇓ – 12)。然而,更复杂的图案,如多环,仍然具有建模挑战性(1),因此,算法设计的 RNA 在体外经常错误折叠。从业者必须经常依靠反复试验细化或针对特定问题的选择方法(1 ⇓ ⇓ ⇓ ⇓ ⇓ – 7)。
高通量合成和生化询问为开发更好的折叠模型提供了前景。尽管如此,一小群专业科学家必须解释这一洪流的经验数据,即使使用现代机器学习和可视化工具也是一项具有挑战性的任务。这种大数据科学的结果往往缺乏手工模型的简洁、优雅和预测能力。本文提出了一种替代方法,即大规模开放实验室,它将高通量实验生物化学的并行性与详细的人工指导实验设计和分析的优势相结合。
拥有 37,000 名成员的 EteRNA 项目现在已经生成了数百个以单核苷酸分辨率探测的设计,从而形成了一个包含近 100,000 个数据点的数据库。这个前所未有的设计数据集并没有超过人类的管理,而是与社区提出的详细手工假设同时创建,其中大部分以前在 RNA 建模文献中未曾探索过。通过机器学习筛选和自动化这些假设产生了一种自动化算法 EteRNABot,它简洁地描述了 RNA 设计的独特优化功能。一系列额外的设计目标测试了该算法,包括以前看不见的 RNA 二级结构以及小分子传感器的复杂支架,结合提供折叠精度的独立读数。这些测试证实,EteRNABot 设计的 RNA 和社区手工制作的 RNA 都优于现有的最先进算法。虽然以前的互联网规模的社区已经在计算机上解决了难题(13 ⇓ ⇓ – 16 ),这里的结果是独一无二的,它表明这样一个社区可以通过实际实验共同产生和检验假设,这是推进实证科学所必需的。
二、结果
EteRNA 将用于模拟生物分子的交互式界面与远程湿实验室实验管道相结合(材料和方法和图 1)。一个基于网络的界面要求参与者设计和排序在体外合成时将折叠成目标结构的序列(SI 附录,图 S1 和表 S1给出了所有设计目标),并制定了解释社区实验结果的设计规则。高通量合成和结构映射测量选择性 2'-羟基酰化与引物延伸 (SHAPE)(材料和方法和图1C) 每周评估八种社区选择设计的核苷酸配对。EteRNA 通过单核苷酸分辨率的数据可视化(图1D)以及 0-100(材料和方法)范围内的整体结构映射分数将这些实验结果返回给参与者,表明提供反应性的核苷酸的百分比与目标结构一致(实验误差±5)(SI附录,图S2)。由于参与者对实验成功设计的直觉特征,他们可以将启发式提交给设计规则集合(图 1 E)。平台设计的附加说明(SI 附录,图 S3)、参与者的培训、奖励结构、可视化、参与者分布(SI 附录,图 S4)和实验再现性(SI 附录,图 S2)在材料和方法和SI 附录中给出。
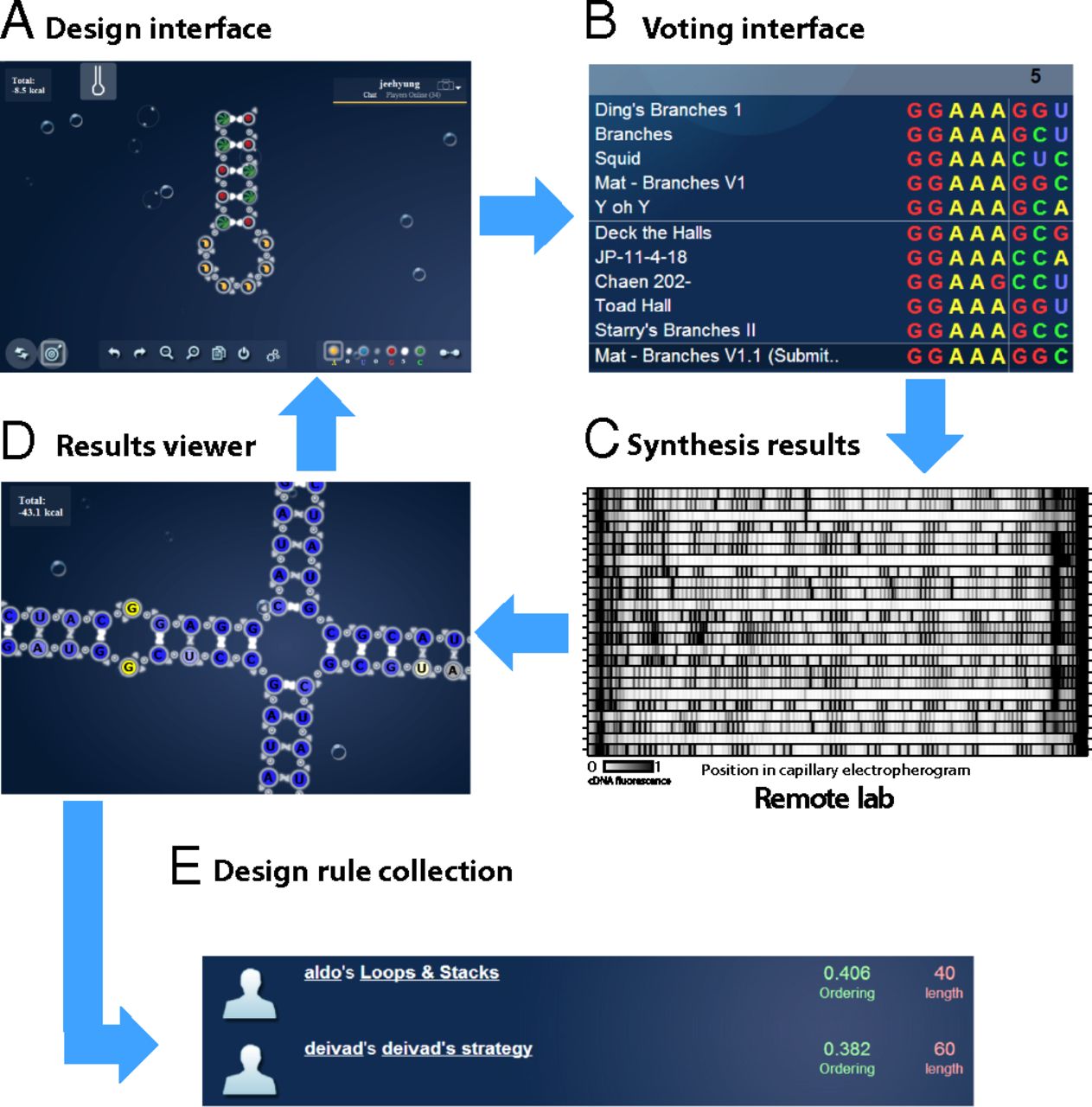
图 1。 EteRNA 工作流程。每周,参与者(A)在序列设计界面中设计可以折叠成目标 RNA 结构的序列,(B)通过投票界面审查和投票选出最佳设计。(C)在回合结束时,八个最高投票的序列被合成并通过单核苷酸分辨率化学反应性测量进行验证。( D ) 实验结果在线发布,可在结果查看器中查看。参与者然后创建新的假设并 ( A ) 开始下一个实验周期或 ( E ) 提交从结果(文本)中学到的设计规则,这些规则被编纂并根据迄今为止获得的分数(数字)自动排名。
最初的 6 个月训练期,称为阶段 I,看到 EteRNA 社区参与了六个 RNA 设计问题,其中包含越来越多的非螺旋元件(凸起和多螺旋连接,bulges and multihelix junctions)和更复杂的拓扑结构(图 2),模仿已知功能 RNA 的成分( 1 , 5 , 6 , 18 ); 合成了 189 个社区选择的序列以及来自 RNAInverse ( 11 ) 和 NUPACK ( 12 ) 算法的65 个序列以进行比较。最初,社区缺乏经验,他们的设计完全依赖于计算折叠模型(11)。这些设计表现不佳:(第一实验室竞争(三螺旋手指)期间图2甲),许多参与者的设计给结构映射得分大于70((有大于90的所有NUPACK设计相比)降低SI附录,表 S2 )。然而,随着社区获得经验性 RNA 设计周期的经验,性能得到提高,并且社区提交的所有目标在两到三轮内收敛到成功的设计(超过 90)(图 2)。 、

图 2。 第一阶段的谜题和结果按谜题发布日期的顺序排列。顶部显示每个拼图 ( A–F )的目标结构和标题。目标结构中的核苷酸着色表明理想的 SHAPE 反应性(金色代表高反应性,蓝色代表低反应性)。中间给出了为所有设计测量的单核苷酸分辨率反应性数据。黄色条纹表示如果形成目标二级结构(顶部),则应显示出高反应性的碱基。底部显示了与 EteRNA 参与者相比的 RNAInverse(黑色)和 NUPACK(灰色)算法设计的结构映射分数的摘要(彩色符号;在每轮设计中按分数排序)。每个设计都在两种溶液条件下进行 SHAPE 化学反应性映射,1 M NaCl(圆圈)和 10 mM MgCl 2(正方形),以及 50 mM Na-Hepes (pH 8.0) 在 24 °C。彩色边框线连接同一轮内的设计。
除了这种针对特定目标的学习之外,每个新目标的第一轮结构映射分数随着时间的推移而增加,这表明参与者正在开发可推广的设计规则(图 2 中的蓝色符号)。在所有六个目标中,这些第一轮分数不断增加。由第三目标(图2 Ç),参加者在其第一轮最高得分优于既RNAInverse和NUPACK。通过第五和第六个目标(图 2 E和F),第一轮参与者得分中位数超过了算法的最高分,顶级参与者设计的得分与给定实验误差 (>95) 的完美设计无法区分。相比之下,不断增加的结构复杂性(在茎和连接处测量)导致 RNAInverse 和 NUPACK 的性能下降(图 2)。在最后三个谜题中,EteRNA 参与者的第一轮设计明显优于 RNAInverse 和 NUPACK 的设计,两种算法的P值为 2.9 × 10 -4(图 2,结构映射数据和SI 附录,表 S3 B)。我们通过基于几个额外设计挑战(SI 附录,图 S5)、自动 SHAPE 指导的二级结构推断(SI 附录,图 S6)、一种不同的基于化学作图方法的额外测试,独立确认了 EteRNA 训练期间的这些结果。关于硫酸二甲酯烷基化 ( 19 )(SI 附录,图 S7),使用信息更丰富的突变和映射技术(20)进行2D 化学作图(这表明失败设计中的结构异质性;SI 附录,支持结果和图.S8),并由单独的实验者和替代技术(下一代测序)进行复制(SI 附录,图 S2)。SI 附录,支持结果给出了这些结果和结构模型的完整描述。
在培训挑战期间,社区提交的设计规则集合增加到 40 个贡献(图 3和SI 附录,表 S4),其中大部分编码了对成功 RNA 设计的独特见解。一方面,涉及的一些先前在RNA设计文献讨论的特征这些规则[例如,GC含量(SI附录,表S4,一个基本的测试),则系综缺陷(8,12)(SI附录,支持结果和表S4,Clean Dot Plot)和序列对称最小化(21)(SI 附录,表 S4,重复)]。一些特征类似于天然结构 RNA 的生物信息学分析中突出显示的模式,例如多环连接处 GC 闭合对的流行(SI 附录,表 S4,连接处的 GC 对)或茎外腺苷的普遍流行(SI 附录,表 S4,仅在循环中)(18)。另一方面,据我们所知,大多数 EteRNA 设计规则在 RNA 折叠和设计领域都是独一无二的,包括与茎相邻的未配对核苷酸的身份的规定(SI 附录,表 S4,钩区无蓝色核苷酸) , CG vs. GC 边缘碱基对在不同的上下文中(SI 附录,表 S4,多循环+ 颈部区域中 GC 对的方向),以及循环内 Gs 的位置(SI 附录,表 S4,Gs 代替任何末端循环右侧的最后一个 As)。

图 3。 EteRNA 参与者提出的 RNA 设计规则。(A)每个设计代理(EteRNA 参与者、NUPACK 和 RNAInverse)的最佳设计,用于阶段 I 的最后一个目标形状(图 2 F);核苷酸着色给出了实验化学反应性并且与图2中使用的着色相同。设计被注释为违反参与者提出的 40 条规则中的前 5 条规则,这些规则通过稀疏线性回归进行评估。( B ) EteRNABot 使用的五个规则语句。括号中的数值参数经过优化,以根据参与者提出的起始值(材料和方法)最好地解释训练集的结果。
这些规则中很少有以前被编码到能量模型或自动 RNA 设计方法中,更不用说经过实验证实了,而且目前还不清楚参与者提出的规则是否能解释他们优于先前设计方法的表现。因此,我们试图通过将规则整合到单个评分函数中来独立于 EteRNA 参与者评估规则。具有交叉验证的稀疏机器学习回归 ( 22 ) 选择了五个规则(图 3),我们通过将其合并到称为 EteRNABot 的独特自动化蒙特卡罗算法和严格的实验测试中进行测试。材料和方法以及SI 附录,支持结果提供关于此算法的其他讨论,一个不太简约的算法 EteRNABot-alt 重新加权所有 40 条规则,以及仅使用项目界面中预先存在的特征的变体算法。
在随后的测试阶段,称为第二阶段,九个独特的目标挑战了 EteRNA 参与者、EteRNABot 方法和先前的算法(图 4和SI 附录,图 S9)。前五个目标(图 4 A-E)是彼此不同的多结结构和拓扑结构中的 I 相结构。我们只评估了每个目标的一轮参与者设计,从而测试社区知识是否可以跨目标结构泛化。与 RNAInverse 和 NUPACK 相比,我们再次观察到参与者设计的优越性能(分别为P = 1.5 × 10 -4和 2.9 × 10 -4)(SI 附录,表 S3C )。此外,在五个案例中的三个(图 4 B、D和E)中,来自独特 EteRNABot 算法的自动化设计实现了参与者设计的 ±1.5 以内的最大分数和 ±5.5 以内的中位数。在剩下的两个例(图4阿和Ç),EteRNABot温和劣的参与者,其可以接近更多的实验数据和设计规则被收集的间隙。重要的是,EteRNABot 的表现优于 RNAInverse 和 NUPACK(分别为P = 3.0 × 10 -4和 1.2 × 10 -3)( SI 附录,表 S3 C),在所有情况下都具有更高的最高分数(图 4和SI 附录,表 S2 A)以及更好的对顶级设计进行排名的能力(SI 附录,图 S10)。
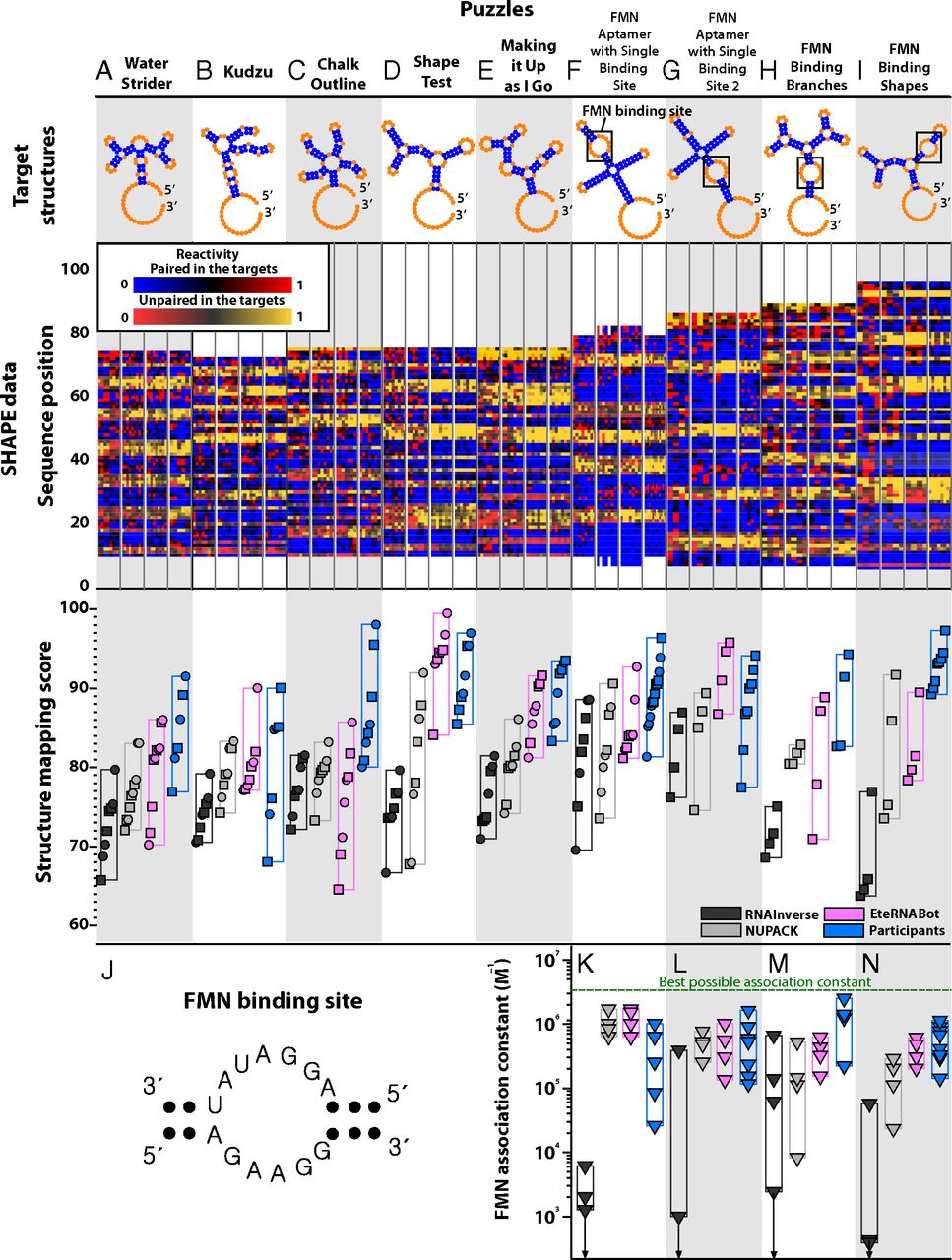
图 4。 第二阶段的谜题和结果,包括 EteRNABot 算法,按照谜题发布日期的顺序。(A-E)彼此不同的多结目标结构和拓扑中的第一阶段结构。( F–I ) 拼图给出 FMN 的结合位点作为固定序列,必须显示为复杂结构内的内部循环(在目标结构中框出;J显示序列)。配色方案与图 2 中使用的配色方案相同,其中 EteRNABot 显示为洋红色。( K-N) 为最后四个拼图合成的所有设计的 FMN 关联常数,通过硫酸二甲酯化学映射作为 FMN 浓度的函数测量。对显示 FMN 结合(绿色虚线)的简单结构的测量给出了可能的最佳关联常数,这只有通过正确的二级结构折叠才能实现。K-N 中的向下箭头标记了 RNAInverse 设计,它没有提供可观察的 FMN 结合,设置了关联常数的上限。
第二阶段的最后四个难题(图 4 F-I)提出了在基于 RNA 的开关工程中出现的挑战:包含传感器域(在这种情况下,结合小分子黄素的 13-nt 内部环单核苷酸 (FMN),其序列保持固定) ( 23,24 )。与之前的结果一致,EteRNA 参与者和 EteRNABot 算法在无 FMN 条件下的结构映射分数方面优于 NUPACK 和 RNAInverse(在所有比较中P < 0.06)(图 4 F-I和SI 附录,表 S3 D)。此外,这些设计的 FMN 结合关联常数提供了严格的折叠精度测试,完全独立于结构映射分数(图 4 K-N和SI 附录,支持结果和图 S11)。再次,EteRNA 参与者和 EteRNABot 在最佳和中值关联常数(在所有成对比较中P < 0.05)都优于 RNAInverse 和 NUPACK (图 4 N和SI 附录,表 S3 E)。这些小分子结合测量独立地证实了上述 SHAPE 结果:EteRNA 社区制定的规则允许比以前更准确的 RNA 二级结构自动化设计。由此产生的 EteRNABot 算法应该立即具有实际用途。
三、讨论
EteRNA 项目通过为互联网规模的公民科学家社区提供高通量湿实验室实验,发现了独特的 RNA 设计规则,总计近 100,000 个单核苷酸分辨率数据点。社区的设计规则已经通过设计测试得到了经验和严格的验证,这些测试涉及九个目标结构,不同于训练期间的六个结构,以及四个支架结构上的独立黄素单核苷酸结合滴定。在这些测试中优于先前的硅指标(SI 附录,图 S10 和 S12) 证实了实验在启发规则方面的重要性。将需要机械工作来为规则的预测能力提供原子级的解释。从这个意义上说,EteRNA 规则类似于能量模型,例如最近邻规则 ( 25 ),它们也是凭经验推导出来的,但还不能从第一原理 ( 26 , 27 ) 或其他设计启发式 ( 28 )推导出来。从机制的角度来看,迄今为止收集的许多 EteRNA 设计规则共有的一个有趣特征是使用否定设计规则。例如,对重复n 的惩罚-mers(重复)、不允许强弱四环的混合(四环相似性)以及相邻碱基对(扭曲碱基对)之间相似性的惩罚是潜在的策略,可以防止在任何合理的能量模型中错误折叠。这些特征可能无法在先前的 RNA 设计算法中捕获,这可能会通过过度优化特定能量模型的特性来揭示与错误折叠相比稳定目标结构的设计。EteRNA 项目中与能量函数无关的负设计规则的出现强调了实际实验证伪/验证在开发 RNA 设计方法中的重要性。
除了对 RNA 工程的影响之外,我们的方法代表了通过众包生成和实验测试假设的成功尝试。随着实验方法的数据吞吐量不断增长,这种方法提供了几个好处。目前,一小组专业科学家试图解决设计和分析高通量实验的复杂性,并列举出一个折叠假设空间来对这些数据进行计算分析。相反,这里的方法使大量参与者能够并行设计和执行远程实验,而机器学习算法则筛选社区的假设目录。这个大规模开放实验室模板可以推广到广泛的生物分子设计问题,26),假结的造型,RNA的工程开关用于蜂窝控制(5,6),和3D建模和设计,全部由高通量映射(评估1,7,16,20,29,30)。其他领域,例如分类学 ( 31 )、天文学 ( 13 ) 和神经映射 ( 32 ),正在互联网规模的科学发现游戏中做出开创性的努力。我们大规模开放实验室的结果表明,将及时的玩家提议的实验作为标准游戏的一部分进行整合,对于此类项目来说将是值得挑战的。
参考资料
- RNA design rules from a massive open laboratory 。 https://www.pnas.org/content/111/6/2122?utm_source=dlvr.it&utm_medium=linkedin
